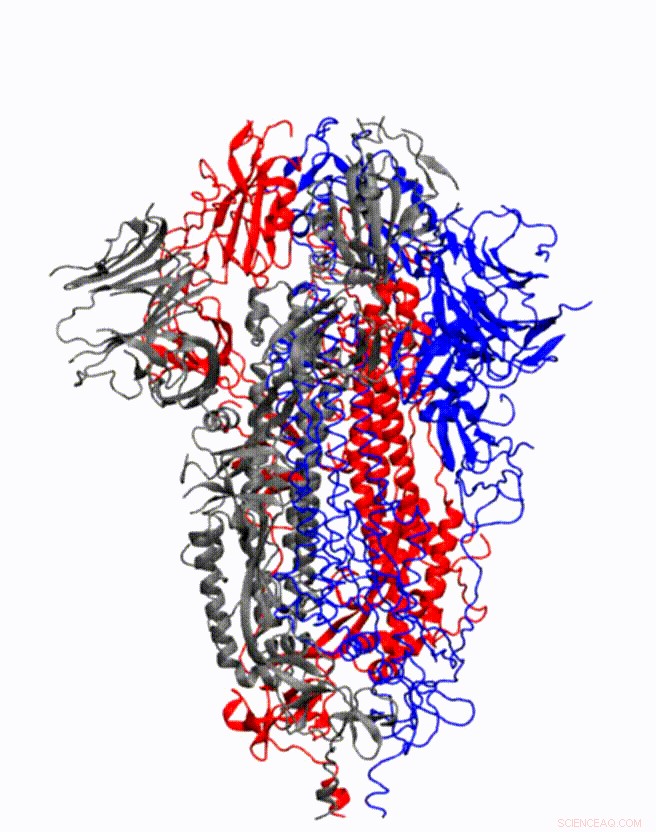
Animation montrant comment la protéine de pointe du coronavirus change de forme juste avant de se lier au récepteur cellulaire humain. Crédit :Illustration fournie par Mahmoud Moradi.
Le chimiste informaticien Mahmoud Moradi développera des Simulations 3D de la dynamique moléculaire des glycoprotéines de pointe du coronavirus pour mieux comprendre comment le virus se lie aux cellules humaines.
La cartographie de la façon dont ces protéines subissent des changements de conformation pour se lier aux récepteurs des cellules hôtes est essentielle au développement de vaccins et de thérapies contre les coronavirus. Les simulations sont particulièrement importantes car un cadre pour la conception de médicaments nécessitera une dynamique, visualisations tridimensionnelles des structures et du comportement cellulaires, plutôt qu'une image statique.
"Comme pour les autres virus, une étape cruciale dans le processus d'infection par le coronavirus est l'entrée virale, " dit Moradi, professeur assistant au J. William Fulbright College of Arts and Sciences. "Avec les coronavirus, nous savons que ces glycoprotéines de pointe médient l'entrée dans la cellule humaine. Les deux SARS-CoV-2, la cause du COVID-19, et SARS-CoV, la cause de l'épidémie de SRAS de 2002-2003, ont des protéines de pointe qui se fixent au même récepteur dans les cellules humaines."
Le travail de Moradi fait partie du Consortium de calcul haute performance COVID-19, une collaboration du gouvernement, l'industrie et les partenaires universitaires se sont concentrés sur les ressources informatiques pour la recherche COVID-19. Dirigé par le Bureau de la politique scientifique et technologique de la Maison Blanche, le département américain de l'Énergie et IBM, le consortium offre gratuitement du temps de calcul et des ressources sur certains des superordinateurs les plus puissants au monde.
Pour effectuer les simulations, Moradi a obtenu l'accès à Frontera, un supercalculateur parrainé par la National Science Foundation et hébergé à l'Université du Texas à Austin. Frontera est le plus grand superordinateur de tous les campus universitaires.
Le projet de Moradi bénéficie de plusieurs récents, modèles 3-D haute résolution des protéines de pointe du coronavirus. Ces modèles peuvent être utilisés comme structures initiales pour commencer des simulations qui permettront d'analyser les mécanismes détaillés des protéines et leur comportement lors de l'entrée virale. Amélioré, des simulations détaillées de cette dynamique moléculaire fourniront une image complète des changements structurels des protéines, ainsi que la façon dont ils se lient à l'enzyme de conversion de l'angiotensine 2, le récepteur spécifique des cellules humaines.
Les recherches de Moradi se situent à l'intersection de la biologie, la physique, chimie, mathématiques, statistiques et informatique. Ses simulations biomoléculaires et ses théories computationnelles expliquent comment les protéines, les molécules chevaux de bataille des cellules, fonctionner au niveau moléculaire. Ses travaux améliorent les modèles géométriques pour décrire comment les protéines changent de forme et comment ces changements affectent le comportement d'une protéine. En février, il a reçu 650 $, 000 National Science Foundation Faculty Early Career Development Award pour ce travail.