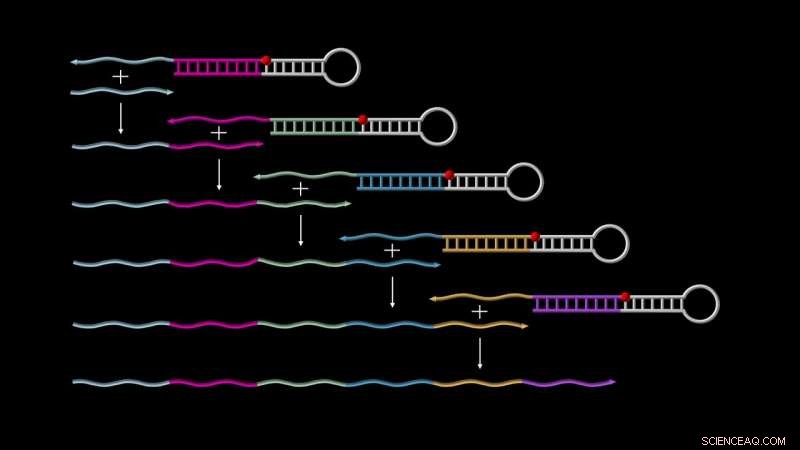
Les cascades de réaction d'échange d'amorces (PER) permettent la croissance autonome d'ADN simple brin. Au sommet, une « épingle à cheveux PER catalytique » lie une première « amorce » (représentée par un court brin gris), déclenche son allongement avec une séquence codée par l'épingle elle-même, et le relâche pour démarrer un autre cycle avec l'amorce déjà étendue, et ainsi de suite, jusqu'à ce qu'une longue transcription soit générée. Crédit :Institut Wyss de l'Université Harvard
Les biologistes synthétiques et les nanobiologistes réutilisent l'ADN, le matériel héréditaire présent dans presque toutes les cellules du corps, en tant que matériau d'auto-assemblage intelligent et stable pour construire des nano-usines, des nanostructures et des dispositifs moléculaires libérant des médicaments qui peuvent détecter leur environnement et réagir de différentes manières en, par exemple, détecter l'inflammation dans le corps ou les toxines dans l'environnement. Ces applications à l'échelle nanométrique impliquent souvent la synthèse de grandes séquences comprenant des milliers d'éléments constitutifs de l'ADN, connu sous le nom de A, T, bases nucléotidiques C et G, qui peut être encore plié et structuré en raison des capacités d'appariement de bases spécifiques entre As et Ts, et Cs et Gs, respectivement.
Cependant, jusque là, les chercheurs n'ont pas d'outils à leur disposition qui permettraient à de plus grandes séquences simple brin de croître de manière autonome puis de se rejoindre de bout en bout suivant un plan de conception moléculaire, une capacité qui pourrait générer des structures et des dispositifs avec des capacités diverses.
Publié aujourd'hui dans Chimie de la nature , les recherches de Peng Yin au Wyss Institute for Biologically Inspired Engineering de Harvard fournissent une solution largement applicable à ce problème. Yin et son équipe ont développé une méthode qui permet à des séquences d'ADN préconçues de croître et de se concaténer de manière autonome le long de voies d'assemblage spécifiques, fournissant ainsi la base d'une nouvelle génération de dispositifs moléculaires programmables. Mettant à l'épreuve leur nouveau concept de « réaction d'échange d'amorces » (PER) en cascade, ils ont conçu avec succès un premier ensemble d'appareils aux fonctions diverses, tels que l'ADN-origami auto-construit et les nanostructures d'ADN qui détectent, amplifier, enregistrer ou évaluer logiquement les signaux environnementaux.
Les méthodes antérieures produisaient des copies identiques d'une séquence fixe plus petite, mais ils sont incapables d'ajouter différentes séquences synthétisées les unes aux autres dans des modèles définis pour générer des assemblages plus importants de manière autonome sans intervention de l'utilisateur. « Les fonctionnalités autonomes et programmables offertes par les cascades PER pourraient engendrer une toute nouvelle génération de dispositifs et d'applications moléculaires programmables et combler les lacunes dans les efforts de conception, pour lesquels de nombreuses pièces mobiles existent déjà, " a déclaré Peng Yin, membre du corps professoral du Wyss Institute, Doctorat., qui a dirigé l'étude et est également professeur de biologie des systèmes à la Harvard Medical School (HMS). « Nous fournissons des données de validation de principe pour le PER dans un large éventail d'applications de biologie synthétique de pointe qui mettent clairement en évidence le vaste potentiel de la technologie. »
L'équipe du Wyss Institute a utilisé le nouveau concept pour concevoir une série de ces transcrits d'ADN PER pour des applications très diverses, y compris la synthèse autonome de grandes nanostructures d'ADN appelées ADN-origamis, et les approches de biologie synthétique, dans laquelle la synthèse d'un transcrit d'ADN repose sur un déclencheur, comme un petit micro-ARN associé au cancer. Leur approche PER peut même générer des transcrits d'ADN résultant d'une combinaison logiquement évaluée de différents déclencheurs, similaire à RNA Ribocomputing Devices que l'équipe de Yin a publié plus tôt cette année. De façon intéressante, Les transcrits d'ADN PER peuvent devenir eux-mêmes catalytiques, être capable de couper un ARN cible arbitraire, deviennent des sondes marquées par fluorescence qui amplifient la présence d'un stimulus moléculaire particulier, ou « enregistreurs moléculaires » qui indiquent fidèlement l'ordre dans lequel certains signaux moléculaires apparaissent dans leurs environnements.
Pour démarrer la cascade PER, deux composants de base sont nécessaires. L'un est appelé "médiateur en épingle à cheveux d'ADN catalytique", qui est une molécule d'ADN simple brin qui s'apparie partiellement avec elle-même pour former une structure en épingle à cheveux avec un court brin simple en surplomb. Ce porte-à-faux est destiné à capter le deuxième composant des cascades PER, le "primaire, " qui contient une région complémentaire du porte-à-faux. Par une série de réactions d'allongement et de déplacement, l'amorce est étendue avec une séquence fournie par le médiateur en épingle à cheveux catalytique puis expulsée. Cela libère le médiateur en épingle à cheveux catalytique pour cascader le prochain cycle du processus, soit en capturant une nouvelle amorce de départ ou l'amorce déjà allongée - et ainsi de suite.
Ces voies de synthèse complexes se déroulent de manière autonome, comparable à un robot moléculaire effectuant une tâche donnée, et à une seule température, ce qui rend la technologie très robuste. "L'approche nous donne une immense liberté de création :nous pouvons non seulement synthétiser le même morceau d'ADN encore et encore en tant que nouveaux ajouts d'une séquence en croissance, mais nous pouvons également varier les types de séquences d'ADN à ajouter simplement en changeant la composition des ADN en épingle à cheveux catalytiques et des amorces dans le mélange pendant que l'assemblage est en cours. Cela nous permet de faire bifurquer la synthèse dans différentes directions et de modeler de manière complexe la composition du transcrit d'ADN final, " a déclaré le premier auteur de l'étude, Jocelyn Kishi, qui, en tant que chercheur diplômé de la National Science Foundation (NSF) au HMS, travaille dans l'équipe de l'Institut Wyss de Yin. « Nous travaillons maintenant à la mise en œuvre de cascades PER pour une variété d'applications, y compris les enregistreurs moléculaires, diagnostics sophistiqués, et l'imagerie tissulaire. Nous espérons également qu'un jour, ces systèmes pourront être utilisés dans des cellules vivantes en tant que dispositifs pouvant enregistrer des événements ou reprogrammer le comportement des cellules de manière spécifique, " dit Kishi.
"Cette nouvelle avancée qui montre comment les molécules d'ADN peuvent être programmées pour s'auto-assembler en structures 3D spécifiques et exécuter des fonctions et des tâches prédéfinies représente une avancée majeure dans le domaine de la robotique moléculaire, et donne un aperçu de l'avenir des dispositifs pour les applications médicales et non médicales, " a déclaré le directeur fondateur du Wyss Institute, Donald Ingber, MARYLAND., Doctorat., qui est également le professeur Judah Folkman de biologie vasculaire au HMS et le programme de biologie vasculaire du Boston Children's Hospital, ainsi que professeur de bio-ingénierie à la Harvard John A. Paulson School of Engineering and Applied Sciences (SEAS).