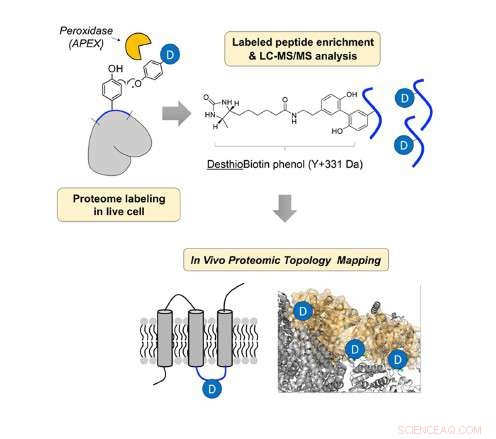
Identification structurale in vivo de la protéine mitochondriale, à l'aide de peroxydase et de desthiobiotine-phénol. Crédit :UNIST
Les protéines membranaires représentent environ un quart de tous les produits génétiques et sont les cibles de plus de 50 pour cent de tous les médicaments pharmaceutiques modernes. Le protéome de la membrane mitochondriale interne (IMM) joue un rôle central dans le maintien de la physiologie mitochondriale et du métabolisme cellulaire. Malgré leur importance, il n'y a eu aucune méthode pour révéler la topologie des protéines membranaires mitochondriales dans les cellules vivantes, jusqu'à maintenant.
Une étude récente, affilié à UNIST a développé une nouvelle technique pour comprendre les architectures correctes des protéines IMM, à l'aide d'outils chimiques spéciaux. En résolvant l'étape la plus difficile du développement de nouveaux médicaments, leur travail contribuera à accélérer le développement de nouvelles thérapies et traitements.
Cette recherche a été dirigée par l'équipe du professeur Hyun-Woo Rhee de chimie à l'UNIST en collaboration avec le professeur Jong-Seo Kim au Center du Center for RNA Research, au sein de l'Institut des sciences fondamentales (IBS) de l'Université nationale de Séoul et du professeur Jeong-Kon Seo des installations centrales de recherche de l'UNIST (UCRF). Les résultats de l'étude ont été publiés dans l'édition du 15 mars du Journal de l'American Chemical Society ( JACS ).
L'IMM est l'un des sites les plus actifs pour le métabolisme cellulaire et il est profondément lié à diverses maladies métaboliques humaines, notamment le cancer et les maladies neurodégénératives. Par conséquent, il est crucial de comprendre l'architecture correcte du protéome IMM dans les cellules vivantes pour un développement réussi et efficace de thérapies ciblées sur les mitochondries.
Dans l'étude, Le professeur Lee et son équipe de recherche ont révélé la direction topologique in vivo de 135 protéines IMM, en utilisant une sonde radicalaire desthiobiotine-phénoxyle générée in situ avec une peroxydase génétiquement ciblée (APEX).
"La détermination de la structure des protéines membranaires est l'une des tâches les plus difficiles de l'analyse de la structure des protéines, ", déclare le professeur Lee. "Notre identification des informations structurelles sur le protéome de la membrane interne mitochondriale peut fournir des informations précieuses pour l'architecture et le connectome du protéome IMM dans les cellules vivantes."
L'équipe de recherche a conçu une nouvelle sonde chimique, desthiobiotine-phénol et l'a appliqué aux protéines IMM dans les cellules vivantes. Puis, ils ont identifié la structure des protéines membranaires par spectrométrie de masse (MS).
La peroxydase peut réagir avec le peroxyde d'hydrogène pour former le radical phénoxyle. Puis, le radical phénoxyle peut réagir avec le résidu tyrosine sur la protéine proximale en formant une liaison covalente. Dans l'étude, l'équipe de recherche a obtenu les informations topologiques en analysant le site tyrosine marqué de la protéine membranaire.
La majorité des analyses de séquences protéiques utilise aujourd'hui la spectrométrie de masse (MS), qui digère l'échantillon de protéine en peptides en utilisant une enzyme appropriée. Analyses précédentes, qui a utilisé l'ascorbate peroxydase génétiquement ciblée (APEX), n'a pas pu résoudre l'identification structurelle car ces analyses étaient basées sur la détection de peptides non marqués. Cependant, seul le peptide marqué peut fournir des informations structurelles utiles, selon l'équipe de recherche.
Contrairement aux biomolécules marquées au biotine-phénol, les protéines et autres cibles marquées au desthiobiotine-phénol peuvent être éluées sans agressivité, conditions dénaturantes. De plus, à mesure que le nombre d'échantillons de structure de protéines membranaires disponibles obtenus via MS augmente, l'efficacité de l'identification structurelle des protéines membranaires augmente également.
En raison de la courte durée de vie des radicaux phénoxyles générés in situ par les APEX ciblés subjectochondriaux et de l'imperméabilité de l'IMM aux petites molécules, les résidus de tyrosine exposés au solvant des côtés de la matrice et de l'espace intermembranaire (IMS) des protéines IMM ont été exclusivement marqués avec la sonde radicalaire dans les cellules vivantes par Matrix-APEX et IMS-APEX, respectivement et identifiés par spectrométrie de masse.
A travers cette analyse, l'équipe de recherche a confirmé 58 topologies de protéines IMM et déterminé la direction topologique de 77 protéines IMM dont la topologie à l'IMM n'a pas été entièrement caractérisée.