Crédit :Université de Pennsylvanie
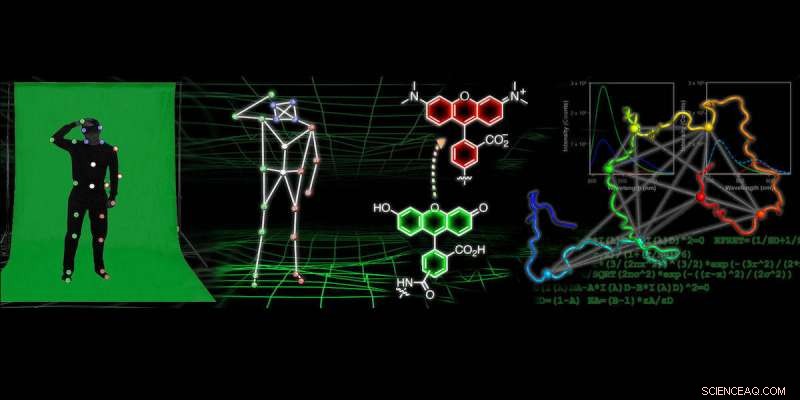
Dans de nombreux films d'animation modernes, l'astuce pour obtenir des mouvements réalistes pour des personnages et des objets individuels réside dans la technologie de capture de mouvement. Ce processus implique souvent une personne portant une combinaison de pistage recouverte de petits, boules colorées tandis qu'une caméra capture la position de ces boules colorées, qui est ensuite utilisé pour représenter comment la personne se déplace.
Des chercheurs de l'Université de Pennsylvanie développent une technologie similaire pour obtenir des "films" à résolution atomique qui suivent comment les protéines se replient et changent de forme. Pour générer ces films, les scientifiques marquent la protéine avec des sondes à de nombreuses positions et observent le mouvement de ces marqueurs. Les données de fluorescence sur les positions relatives des sondes peuvent ensuite être utilisées pour construire des modèles informatiques de la structure de la protéine en détail atomique. Cette recherche pourrait conduire à des améliorations des médicaments utilisés pour traiter les maladies neurodégénératives, ainsi que de nouvelles méthodes d'imagerie qui pourraient conduire à leur détection plus précoce.
La recherche était un effort multidisciplinaire dirigé par E. James Petersson. Le document a été publié en Journal biophysique .
"L'une des grandes questions fondamentales en biochimie est de savoir comment les protéines se replient dans une certaine forme, " dit Petersson, "Et cela est dicté par la séquence d'acides aminés dans la protéine. L'information dans toutes les interactions des chaînes latérales d'acides aminés conduit en quelque sorte à se replier dans une forme appropriée."
Dans les scénarios sains, Petersson a dit, cette forme appropriée permet à la protéine d'avoir différentes fonctions, comme transporter de l'oxygène dans le sang ou devenir des "machines moléculaires" qui conduisent finalement à des mouvements musculaires, tels que ceux nécessaires à la marche et à la course.
Mais dans certains états pathologiques, notamment dans les maladies neurodégénératives telles que la maladie d'Alzheimer et la maladie de Parkinson, les protéines se replient mal dans une forme malsaine, ce qui peut entraîner l'agrégation de plusieurs copies des protéines en « enchevêtrements semblables à des spaghettis ou de longues fibrilles ». Ces fibrilles, Petersson a dit, sont toxiques pour les neurones, qui sous-tend les maladies d'Alzheimer et de Parkinson.
« Comme tout cela implique le repliement des protéines dans une certaine forme, alors ce que nous aimerions faire, c'est suivre les changements de forme des protéines, " Il a dit. " Il existe un certain nombre de techniques différentes qui peuvent être utilisées pour ce faire, mais nous aimons la fluorescence parce que vous pouvez acquérir des données de fluorescence suffisamment rapidement pour pouvoir observer le repliement des protéines en temps réel. En fin de compte, nous aimerions essayer de regarder les protéines se replier dans les cellules. »
Pour obtenir des informations sur la forme des protéines à l'aide de sondes fluorescentes, les chercheurs utilisent une technique appelée transfert d'énergie par résonance de fluorescence, ce qui les oblige à mesurer de nombreuses distances entre différents points de la protéine, puis à utiliser cette information pour comprendre sa forme, similaire à la technologie de capture de mouvement.
Dans ce document, les chercheurs ont effectué environ 30 mesures de différentes distances au sein de la protéine alpha-synucléine sous différents états où elle change de forme. Ils ont ensuite utilisé cette collection de mesures de distance en combinaison avec une modélisation informatique complexe pour obtenir des structures de résolution atomique de la forme de la protéine.
Ferrie a reçu une bourse de la Parkinson's Disease Foundation pour passer l'été dans le laboratoire de David Baker à l'Université de Washington, où Rosette, l'un des programmes les plus couramment utilisés pour la modélisation des protéines, était développé.
" Rosetta est conçu pour modéliser des protéines stables et bien repliées, " Petersson a dit, "pas de protéines désordonnées qui peuvent changer de forme, Jack a donc dû faire beaucoup de réécriture du code lui-même pour pouvoir modéliser ces protéines indisciplinées. »
Ferrie et Yoon ont effectué une série de mesures expérimentales qui pourraient être utilisées pour diriger le repliement des protéines afin qu'il soit cohérent avec les mesures expérimentales, ce qui lui a permis de modéliser la forme des protéines.
Comme il s'agissait d'une nouvelle approche, les chercheurs voulaient démontrer que les structures issues des modèles informatiques étaient cohérentes avec la réalité. Pour ce faire, ils ont mené trois types d'expériences pour faire correspondre les données réelles avec les modèles issus de ces expériences de fluorescence.
Ils ont collaboré avec le groupe de Rhoades pour valider les structures modélisées à l'aide de mesures de fluorescence de molécules uniques effectuées par Pan. Travaillant dans le groupe de Fakhraii, Lin, utilisé un autre type de technique appelée microscopie à force atomique pour imager la protéine et valider les modèles. Chris Dobson, professeur de chimie à l'université de Cambridge, partagé des données de résonance magnétique nucléaire sur l'alpha-synucléine qui ont fourni une confirmation supplémentaire de l'exactitude des modèles informatiques.
L'article représente l'une des plus grandes bibliothèques de protéines marquées avec des fluorophores synthétiques à ce jour. Selon Petersson, les chercheurs ont dû effectuer de nombreuses mesures de distance sur différentes régions de la protéine pour disposer de suffisamment de données pour générer des modèles informatiques. Ferrie et Haney ont dû proposer une approche simplifiée pour attacher différents ensembles de sondes qui fonctionneraient sur différentes plages de distance.
Les chercheurs travaillent maintenant à appliquer cette technique pour modéliser la structure des protéines dans les formes agrégées qui sont toxiques pour les neurones et pour modéliser sa réponse aux médicaments qui la feraient changer de forme, empêchant cette agrégation.
"La capacité d'observer une protéine lorsqu'elle change de forme, " dit Petersson, « Et obtenir des structures à partir de cela est un objectif scientifique fondamental très important sur lequel nous travaillons depuis 10 ans. Il y a eu des percées très impressionnantes dans l'obtention de structures de protéines dans les maladies neurodégénératives, mais la technique de fluorescence a le potentiel de le faire dans des cellules vivantes, ce qu'aucune autre technique n'a la capacité de faire."
Selon Petersson, protéines adoptant des formes multiples, empiler différentes copies les unes sur les autres et les agréger, comme ils le font dans les maladies neurodégénératives, are an important basic biochemistry problem that other structural biology techniques can't really tackle. A better understanding of how that works and what those shapes are has the potential to make an impact on diseases such as Parkinson's and to give researchers an opportunity to figure out how drug or diagnostic models interact with the protein.
"We're working on being able to generate model structures that actually show what is the effect of these drugs, " Petersson said. "We take the protein with the fluorescent labels, add the drug, allow the protein to change shape, make fluorescence measurements and then take those back to the computational modeling so we can actually see the structural effect of these drugs. Hopefully this will lead to more of a rational understanding so that better second and third generation drugs can be made."
The researchers are also collaborating with Robert Mach, the Britton Chance Professor of Radiology in Penn's Perelman School of Medicine. Mach's group is interested in developing positron emission tomography imaging probes that can be used to bind to the aggregated forms of proteins and image them in patients.
"There are some promising drugs for treating neurodegenerative diseases such as Alzheimer's and Parkinson's, that could block this formation of aggregates, " Petersson said, "but the problem is that, by the time people show cognitive or motor-tremor symptoms, it's too late to use these drugs because there's already too much neurodegeneration. If you're getting aggregates in your brain, even if you're not showing any behavioral changes or learning deficits, these probes could noninvasively image the aggregates. By achieving a rational understanding of what the protein structure is, we hope we can help with that work moving forward."