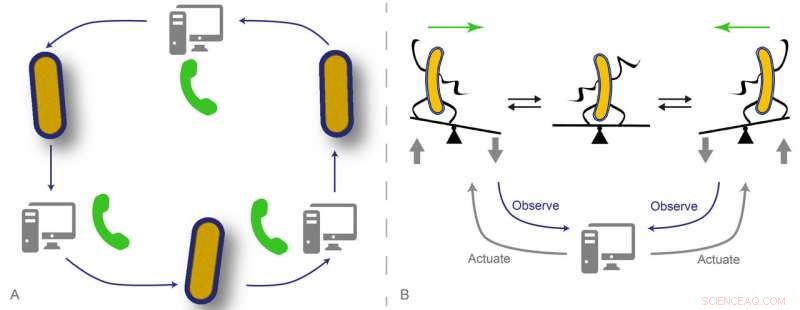
A. Un nouveau téléphone portable :la communication directe de cellule à cellule rendue possible via des interfaces informatiques.B. Stabilisation dynamique :le contrôle en temps réel permet de maintenir les cellules dans des configurations instables. Crédit :Gregory Batt, Jacob Ruess, Chetan Aditya (Inria / Institut Pasteur)
Des chercheurs de l'Institut Pasteur et d'Inria, avec des chercheurs du CNRS et de l'Université Paris Diderot, et de l'Institut des sciences et de la technologie (IST) en Autriche, ont publié deux articles dans Communication Nature sur le contrôle informatique des processus cellulaires. Des plateformes expérimentales hybrides combinant microscopes et logiciels permettent aux chercheurs d'interfacer en temps réel des cellules vivantes avec des algorithmes de contrôle. Les deux articles illustrent que ces solutions permettent de créer de nouveaux comportements facilement reprogrammables de populations cellulaires. Ce contrôle externe du tissu vivant deviendrait alors un formidable outil de recherche pour acquérir une compréhension fine du rôle biologique de certaines protéines et optimiser les processus de bio-production.
Le but de la biologie synthétique, qui allie biologie et ingénierie, est de (re)programmer des cellules afin d'améliorer leurs performances dans une tâche spécifique, ou pour qu'ils puissent effectuer efficacement une nouvelle tâche. L'un des enjeux de cette discipline est donc de contourner les limitations des systèmes biologiques existants. Par exemple, il est difficile d'obtenir la même expression génique dans différentes cellules, même s'ils sont cultivés dans le même milieu. Grâce à ces technologies de pointe, les chercheurs sont capables d'assurer un contrôle homogène d'un processus cellulaire sur une très longue période.
Des chercheurs de l'Institut Pasteur et d'Inria, le CNRS et l'Université Paris Diderot, et l'IST Autriche ont développé deux plates-formes reliant un microscope à un ordinateur. Les cellules sont placées dans un dispositif microfluidique dans lequel l'environnement chimique peut être varié ou les cellules peuvent être exposées à des stimulations lumineuses. Un programme informatique décide des modifications à apporter à l'environnement chimique ou lumineux en fonction du comportement observé des cellules et de l'objectif de l'expérience. L'ordinateur gère également l'acquisition des images par le microscope et leur analyse, quantifier les réponses cellulaires en temps réel.
Dans le premier article, les chercheurs de l'Institut Pasteur et de deux groupes de l'IST Autriche ont utilisé l'optogénétique pour activer l'expression d'un gène en exposant des cellules à la lumière. Une protéine fluorescente est utilisée pour mesurer la quantité de protéine produite. Un contrôleur, en utilisant un modèle du système, peut alors prendre des décisions en temps réel sur les perturbations dynamiques à appliquer en fonction du comportement futur attendu des cellules. Grâce aux programmes informatiques créés par les chercheurs, ils peuvent contrôler chaque cellule individuellement de diverses manières, ou créer une communication virtuelle entre plusieurs cellules, qui font circuler les messages dans un ordre facilement reconfigurable. " Nous avons réussi à construire une plateforme nous permettant de concevoir des circuits partiellement biologiques et partiellement virtuels. Les parties virtuelles de ces circuits peuvent être arbitrairement modifiées pour créer et explorer rapidement des comportements cellulaires, même au-delà de ce qui est biologiquement possible, " dit Jakob Ruess, co-premier auteur du premier article.
Dans le deuxième article, Grégory Batt, chef de l'unité InBio et co-auteur avec Pascal Hersen du CNRS/Université Paris Diderot explique comment ils ont réussi à placer un système cellulaire dans une configuration instable :« Nous avons conçu un programme informatique qui vise à forcer les cellules à prendre des décisions binaires de manière aléatoire. Pour faire ça, les cellules sont conduites vers une région d'instabilité - comme les grimpeurs sur une ligne de crête de montagne - et on les laisse ensuite évoluer librement vers l'une des deux configurations stables possibles. De façon inattendue, nous avons observé qu'une stimulation donnée, s'il est correctement choisi, était capable d'emmener des groupes de cellules différentes dans la région d'instabilité et de les y maintenir. Ces résultats pourraient aider à mieux comprendre comment les populations cellulaires prennent collectivement des décisions solides sans coordination individuelle. »
Les avancées scientifiques décrites dans ces articles ont été rendues possibles par une alliance entre deux disciplines aujourd'hui complémentaires :la biologie et les sciences du numérique. Collaboration étroite entre l'Institut Pasteur et Inria, sous la forme du groupe de recherche InBio join, dont l'objectif est de développer un cadre méthodologique pour parvenir à une compréhension quantitative du fonctionnement des processus cellulaires, est la parfaite illustration de l'intérêt d'une recherche interdisciplinaire alliant approches expérimentales et développements méthodologiques.