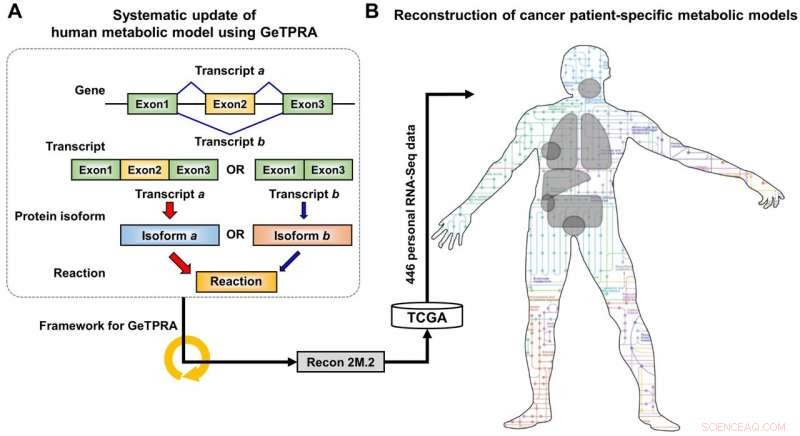
Schéma de développement de Recon 2M.2 et son utilisation dans la reconstruction de modèles métaboliques personnels à l'échelle du génome (GEM). (A) Lors du développement de Recon 2M.2, un concept d'épissage alternatif de gènes humains a été envisagé, et incorporé dans la modélisation via le cadre informatique pour les associations gène-transcription-protéine-réaction (GeTPRA). (B) Lors du développement de Recon 2M.2, Les GEM spécifiques aux patients atteints de cancer pourraient être reconstruits à l'aide de données biologiques personnelles (données RNA-Seq). Dans cette étude, Les données RNA-Seq spécifiques au patient ont été obtenues auprès de The Cancer Genome Atlas (TCGA; https://cancergenome.nih.gov/). Crédit :KAIST
Une équipe de recherche coréenne de KAIST a développé un cadre informatique qui permet la reconstruction d'un modèle informatique complet du métabolisme humain, qui permet une prédiction précise des caractéristiques métaboliques personnelles (ou phénotypes).
Comprendre les phénotypes métaboliques personnels nous permet de concevoir des stratégies thérapeutiques efficaces pour diverses maladies chroniques et infectieuses. Un modèle informatique humain appelé modèle métabolique à l'échelle du génome (GEM) contient des informations sur des milliers de gènes métaboliques et leurs réactions et métabolites correspondants, et a joué un rôle important dans la prédiction des phénotypes métaboliques. Bien que plusieurs versions de GEM humains aient été publiées, ils avaient de la place pour un développement ultérieur, notamment quant à l'incorporation d'informations biologiques provenant d'un mécanisme de génétique humaine appelé « l'épissage alternatif ». L'épissage alternatif est un mécanisme génétique qui permet à un gène de donner lieu à de multiples réactions, et est fortement associée à la pathologie.
Pour s'attaquer à ce problème, Jae Yong Ryu (étudiant au doctorat), Dr Hyun Uk Kim (boursier de recherche), et le professeur distingué Sang Yup Lee, tous issus du Département de génie chimique et biomoléculaire du KAIST, développé un cadre de calcul qui génère systématiquement des réactions métaboliques, et les ajoute au GEM humain. Il a été démontré que le GEM humain résultant prédisait avec précision les phénotypes métaboliques dans des conditions environnementales variées. Les résultats de la recherche ont été publiés en ligne dans Actes de l'Académie nationale des sciences ( PNAS ) le 24 octobre, 2017, sous le titre "Cadre et ressource pour plus de 11, 000 associations gène-transcription-protéine-réaction dans le métabolisme humain."
L'équipe de recherche a d'abord mis à jour le contenu biologique d'une version précédente du GEM humain. Le contenu biologique mis à jour comprend les gènes métaboliques et leurs métabolites et réactions correspondants. En particulier, des réactions métaboliques catalysées par des isoformes de protéines déjà connues ont en outre été incorporées dans le GEM humain; les isoformes de protéines sont de multiples variantes de protéines générées à partir de gènes individuels par le processus d'épissage alternatif. Chaque isoforme de protéine est souvent responsable du fonctionnement d'une réaction métabolique. Bien que plusieurs isoformes de protéines générées à partir d'un gène puissent jouer différentes fonctions en ayant différents ensembles de domaines protéiques et/ou de localisations subcellulaires, ces informations n'étaient pas correctement prises en compte dans les versions précédentes des GEM humains.
Lors de la mise à jour initiale du GEM humain, nommé Recon 2M.1, l'équipe de recherche a ensuite mis en œuvre un cadre informatique qui génère systématiquement des informations sur les associations gène-transcription-protéine-réaction (GeTPRA) afin d'identifier les isoformes de protéines qui n'étaient pas identifiées auparavant. Ce cadre a été développé dans cette étude. Suite à la mise en œuvre du framework pour GeTPRA, plus de 11, 000 GeTPRA ont été prédits automatiquement, et bien validé. Des réactions métaboliques supplémentaires ont ensuite été ajoutées à Recon 2M.1 sur la base du GeTPRA prédit pour les isoformes de protéines précédemment non caractérisées ; Recon 2M.1 a été renommé Recon 2M.2 à partir de cette mise à niveau. Finalement, Recon 2M.2 a été intégré à 446 ensembles de données biologiques personnelles (données RNA-Seq) afin de créer des modèles de cancer spécifiques au patient. Ces modèles de cancer spécifiques au patient ont été utilisés pour prédire les activités du métabolisme du cancer et les cibles anticancéreuses.
Le développement d'une nouvelle version des GEM humains ainsi que le cadre informatique de GeTPRA devraient stimuler les études en génétique humaine fondamentale et en médecine. Fichiers modèles des GEM humains Recon 2M.1 et 2M.2, une liste complète du GeTPRA et le code source du cadre de calcul pour prédire le GeTPRA sont tous disponibles dans le cadre de la publication de cette étude.
Le distingué professeur Lee a dit, "Le GeTPRA prédit à partir du cadre informatique devrait servir de ligne directrice pour les futures expériences sur la génétique humaine et la biochimie, tandis que le Recon 2M.2 qui en résulte peut être utilisé pour prédire des cibles médicamenteuses pour diverses maladies humaines."