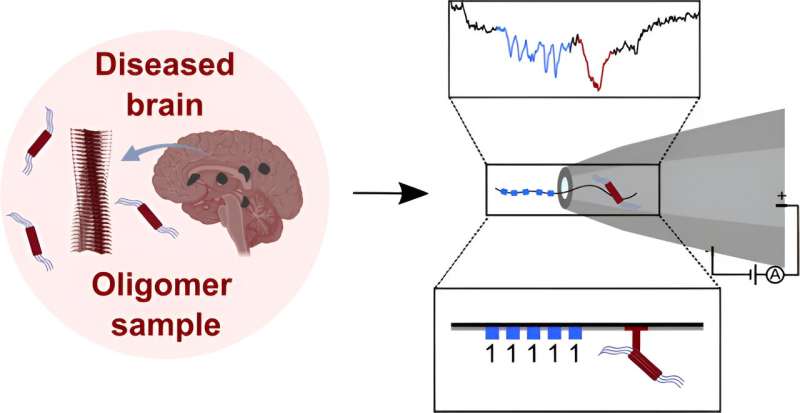
Une équipe de chimistes, de microbiologistes et de physiciens de l'Université de Cambridge au Royaume-Uni a développé un moyen d'utiliser des nanopores à l'état solide et des codes-barres d'ADN multiplexés pour identifier des protéines mal repliées telles que celles impliquées dans les troubles neurodégénératifs dans les échantillons de sang. Dans leur étude, rapportée dans le Journal of the American Chemical Society , le groupe a utilisé des techniques de codes-barres d'ADN multiplexés pour surmonter les problèmes liés aux techniques de filtrage des nanopores pour isoler les oligomères nocifs.
Des recherches antérieures ont montré que la présence d'oligomères nocifs dans le cerveau peut entraîner un mauvais repliement des protéines associées à des maladies neurodégénératives telles que la maladie de Parkinson et la maladie d'Alzheimer. Les chercheurs en médecine ont cherché un moyen de les détecter dans le sang afin de diagnostiquer une maladie neurodégénérative et de suivre sa progression une fois qu'elle a été confirmée.
Malheureusement, les trouver dans des mélanges complexes tels que le sang s’est avéré être une tâche ardue. Une approche s’est révélée prometteuse – utilisant des capteurs nanopores – mais à ce jour, ils ne peuvent pas suivre les oligomères cibles lorsqu’ils traversent des capteurs nanopores à semi-conducteurs personnalisables. Dans ce nouvel effort, l'équipe de recherche a surmonté ce problème en utilisant des nanostructures d'ADN personnalisables.
Dans leur travail, l'équipe de recherche a lié des protéines à leurs nanostructures d'ADN personnalisées afin de créer une sorte de « code-barres » qui pourrait être utilisé pour identifier des molécules à l'aide de capteurs nanopores à l'état solide. Pour utiliser leur code-barres, ils ont placé un morceau d'ADN marqué avec un produit chimique qui se lie uniquement aux oligomères cibles, ce qui a généré un pic supplémentaire, ce qui a permis d'isoler la cible et de la caractériser complètement.
L'équipe a ensuite testé sa technique en utilisant des oligomères d'α-synucléine, le type de protéines impliquées dans le repliement de la maladie de Parkinson, et a pu les isoler comme souhaité dans des conditions de laboratoire. Ils ont également mesuré le taux de formation d’oligomères. Ils ont ensuite testé la technique dans un environnement plus proche des conditions biologiques et ont constaté que les performances n’en souffraient pas. Ils suggèrent que leur technique offre la promesse d'un futur processus de dépistage pour les patients à risque de développer des troubles neurodégénératifs.
Plus d'informations : Sarah E. Sandler et al, Caractérisation numérique multiplexée d'oligomères protéiques mal repliés via des nanopores à l'état solide, Journal of the American Chemical Society (2023). DOI : 10.1021/jacs.3c09335
Informations sur le journal : Journal de l'American Chemical Society
© 2023 Réseau Science X