En attachant des molécules entre elles, des scientifiques de l'Université de Montréal pensent avoir découvert comment les systèmes moléculaires à l'origine de la vie ont évolué pour créer des fonctions d'autorégulation complexes.
Publié dans Angewandte Chemie , leurs découvertes promettent de fournir aux chimistes et aux nanotechnologues une stratégie simple pour créer la prochaine génération de nanosystèmes dynamiques.
La vie sur Terre est soutenue par des millions de minuscules nanostructures ou nanomachines différentes qui ont évolué au fil de millions d'années, a expliqué Alexis Vallée-Bélisle, professeur à l'UdeM et chercheur principal de l'étude.
Ces structures, souvent inférieures à 10 000 fois le diamètre d’un cheveu humain, sont généralement composées de protéines ou d’acides nucléiques. Alors que certains sont fabriqués à partir d'un seul composant ou d'une seule pièce (souvent des polymères linéaires qui se replient dans une structure spécifique), la plupart d'entre eux sont fabriqués à partir de plusieurs composants qui s'assemblent spontanément en de grands assemblages dynamiques.
"Ces assemblages moléculaires sont très dynamiques et s'activent ou se désactivent précisément en réponse à divers stimuli tels qu'une variation de température, d'oxygène ou de nutriments", a déclaré Vallée-Bélisle.
"De la même manière que les voitures qui nécessitent un allumage séquentiel, un desserrage des freins, un changement de vitesse et un apport de gaz pour avancer, les systèmes moléculaires nécessitent l'activation ou la désactivation séquentielle de diverses nanomachines pour effectuer des tâches spécifiques allant du déplacement à la respiration en passant par la réflexion."
Les chercheurs ont soulevé une question fondamentale :comment des assemblages moléculaires dynamiques ont-ils été créés, programmés et affinés pour soutenir la vie ?
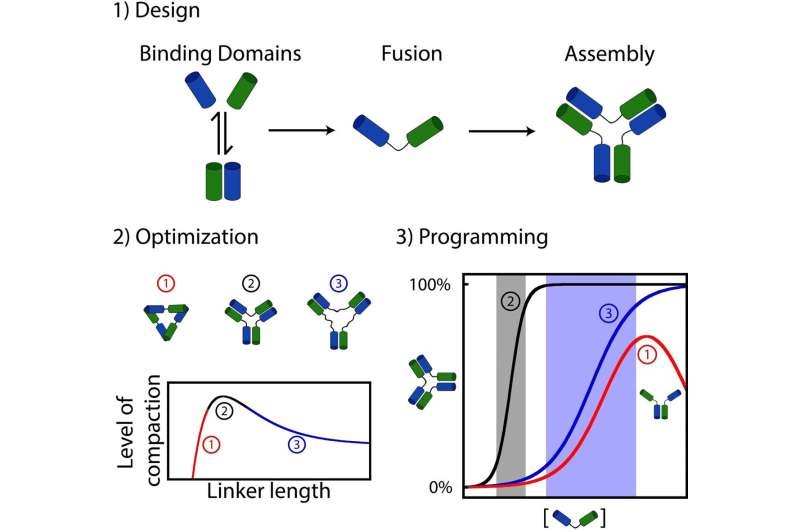
Ce qu'ils ont découvert, c'est que de nombreux assemblages biologiques étaient probablement formés en attachant de manière aléatoire des molécules en interaction (par exemple, des protéines ou des acides nucléiques tels que l'ADN ou l'ARN) avec des agents de liaison agissant comme un « connecteur » entre chaque partie.
"Comme ces assemblages biomoléculaires jouent un rôle crucial en permettant aux organismes vivants de répondre à leur environnement, nous avons émis l'hypothèse que la nature de la connectivité entre les composants attachés pourrait également contribuer à l'évolution de leurs réponses dynamiques", a déclaré Vallée-Bélisle, titulaire de la Chaire de recherche du Canada en bioingénierie et bio-nanotechnologie.
Pour explorer cette question, Dominic Lauzon, étudiant au doctorat au moment de l'étude, a décidé de synthétiser et d'attacher ensemble des dizaines de molécules d'ADN interagissant pour explorer l'impact de la connectivité sur la dynamique d'assemblage.
"La chimie programmable et facile à utiliser des acides nucléiques tels que l'ADN en fait une molécule pratique pour étudier les questions fondamentales liées à l'évolution des biomolécules", a déclaré Lauzon, le premier auteur de l'étude. "De plus, on pense également que les acides nucléiques sont la molécule à l'origine de la vie sur Terre."
Lauzon et Vallée-Bélisle ont découvert qu'une simple variation de la longueur du « lieur » entre les molécules en interaction entraîne des variations significatives dans leur dynamique d'assemblage. Par exemple, certains assemblages présentaient une grande sensibilité aux variations des stimuli, tandis que d'autres manquaient d'une telle sensibilité, ou nécessitaient même des changements de stimuli beaucoup plus importants pour promouvoir l'assemblage.
Plus surprenant, certains agents de liaison ont même créé de nouvelles fonctions de régulation complexes telles que des propriétés d'auto-inhibition, où l'ajout d'un stimulus favoriserait à la fois son assemblage et son désassemblage. Tous ces différents comportements réactifs sont également souvent observés dans les nanomachines « vivantes » naturelles.
À l'aide d'expériences et d'équations mathématiques, les chercheurs ont également pu expliquer pourquoi une variation aussi simple de la longueur du lieur était si efficace pour modifier la dynamique de l'assemblage moléculaire.
"Les lieurs créant les assemblages les plus stables étaient ceux qui créaient également les mécanismes d'activation les plus sensibles, tandis que les lieurs créant les assemblages les moins stables créaient les mécanismes d'activation les moins sensibles, au point même d'introduire l'auto-inhibition", a expliqué Lauzon. P>
La capacité de détecter avec précision les signaux moléculaires est cruciale pour les assemblages biologiques mais également dans le développement de la nanotechnologie qui dépend de la détection et de l'intégration d'informations moléculaires.
Les chercheurs pensent donc que leur découverte pourrait également fournir le cadre fondamental pour créer des nanomachines ou des nanosystèmes plus programmables avec des activités régulées de manière optimale, par exemple en attachant simplement des molécules en interaction avec différents lieurs. De tels assemblages moléculaires trouvent déjà des applications dans la biodétection ou l'administration de médicaments.
En plus de fournir une stratégie de conception simple pour créer la prochaine génération de nanosystèmes autorégulés, les découvertes des scientifiques mettent également en lumière la manière dont les assemblages biomoléculaires naturels pourraient avoir acquis leur dynamique optimale.
"Une stratégie d'évolution moléculaire bien connue des organismes vivants est la fusion génétique, où l'ADN codant pour deux domaines protéiques en interaction est fusionné de manière aléatoire", a déclaré Vallée-Bélisle.
"Nos résultats fournissent également la compréhension fondamentale requise pour comprendre comment une simple variation de la longueur du lieur entre les protéines fusionnées peut avoir efficacement créé des assemblages biologiques affichant une variété de dynamiques, certaines mieux adaptées que d'autres pour offrir un avantage aux organismes vivants." /P>
Plus d'informations : Dominic Lauzon et al, Principes de conception et de thermodynamique pour programmer la coopérativité des assemblages moléculaires, Angewandte Chemie International Edition (2023). DOI :10.1002/anie.202313944
Informations sur le journal : Angewandte Chemie International Edition , Angewandte Chemie
Fourni par l'Université de Montréal