Dans une première démonstration de « vidéographie électronique », des chercheurs ont capturé une image microscopique animée de la danse délicate entre les protéines et les lipides présents dans les membranes cellulaires. La technique peut être utilisée pour étudier la dynamique d'autres biomolécules, en s'affranchissant des contraintes qui limitaient la microscopie aux images fixes de molécules fixes, affirment les chercheurs Urbana-Champaign de l'Université de l'Illinois et les collaborateurs du Georgia Institute of Technology.
"Nous allons au-delà de la prise d'instantanés uniques, qui donnent une structure mais pas une dynamique, pour enregistrer continuellement les molécules dans l'eau, leur état d'origine", a déclaré Qian Chen, responsable de l'étude et professeur de science et d'ingénierie des matériaux dans l'Illinois. "Nous pouvons vraiment voir comment les protéines changent de configuration et, dans ce cas, comment l'ensemble de la structure auto-assemblée protéine-lipide fluctue au fil du temps."
Les chercheurs ont publié leur technique et leurs résultats dans la revue Science Advances. .
Les techniques de microscopie électronique imagent à l’échelle moléculaire ou atomique, produisant des images détaillées à l’échelle nanométrique. Cependant, ils s'appuient souvent sur des échantillons qui ont été gelés ou fixés en place, ce qui oblige les scientifiques à tenter de déduire comment les molécules se déplacent et interagissent, comme si elles essayaient de cartographier la chorégraphie d'une séquence de danse à partir d'une seule image d'un film.
"C'est la première fois que nous examinons une protéine à une échelle individuelle sans la congeler ni l'étiqueter", a déclaré Aditi Das, professeur à Georgia Tech et auteur correspondant de l'étude. "Habituellement, nous devons cristalliser ou congeler une protéine, ce qui pose des problèmes pour capturer des images haute résolution de protéines flexibles. Alternativement, certaines techniques utilisent une étiquette moléculaire que nous suivons, plutôt que d'observer la protéine elle-même. Dans cette étude, nous constatons la protéine telle qu'elle est, se comportant comme elle le fait dans un environnement liquide et observant comment les lipides et les protéines interagissent les uns avec les autres."
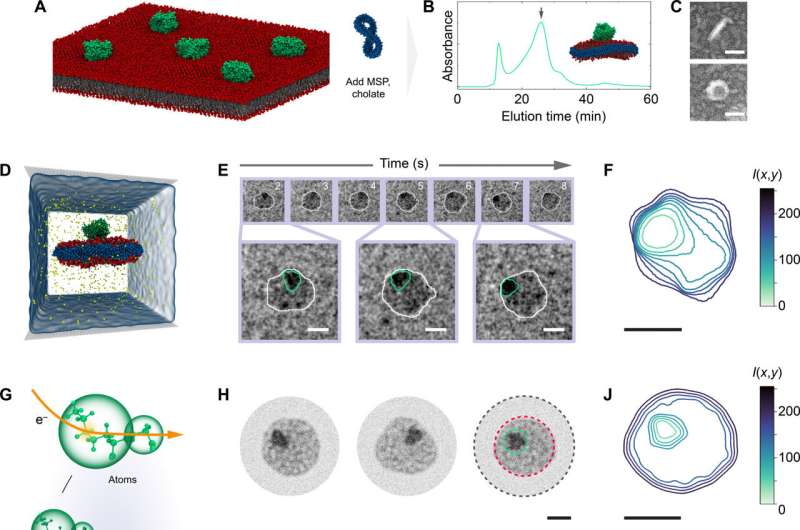
Les chercheurs ont réalisé la vidéographie en combinant une nouvelle méthode de microscopie électronique à transmission à base d’eau avec une modélisation informatique détaillée au niveau de l’atome. La technique à base d’eau consiste à encapsuler des gouttelettes à l’échelle nanométrique dans du graphène afin qu’elles puissent résister au vide dans lequel fonctionne le microscope. La comparaison des données vidéo obtenues avec des modèles moléculaires, qui montrent comment les choses devraient bouger selon les lois de la physique, aide les chercheurs non seulement à interpréter mais également à valider leurs données expérimentales.
"Actuellement, c'est vraiment la seule façon expérimentale de filmer ce type de mouvement au fil du temps", a déclaré John W. Smith, le premier auteur de l'article, qui a terminé ses travaux alors qu'il était étudiant diplômé à l'Illinois. "La vie est liquide et elle est en mouvement. Nous essayons d'obtenir les moindres détails de cette connexion de manière expérimentale."
Pour la nouvelle étude – la première démonstration publiée de la technique de vidéographie électronique – les chercheurs ont examiné des disques nanométriques de membranes lipidiques et comment ils interagissaient avec des protéines normalement trouvées à la surface ou intégrées dans les membranes cellulaires.
"Les protéines membranaires se trouvent à l'interface entre les cellules et entre l'intérieur et l'extérieur de la cellule, contrôlant ce qui entre et sort", a déclaré Smith. "Ils sont majoritairement des cibles pour la médecine; ils sont impliqués dans toutes sortes de processus comme la façon dont nos muscles se contractent, le fonctionnement de notre cerveau, la reconnaissance immunitaire; et ils maintiennent les cellules et les tissus ensemble. Et toute la complexité du fonctionnement d'une protéine membranaire vient de non seulement sa propre structure, mais aussi la façon dont il subit les lipides qui l'entourent."
La vidéographie électronique a permis aux chercheurs de voir non seulement comment l’ensemble lipide-protéine se déplaçait, mais également la dynamique de chaque composant. Les chercheurs ont découvert qu'il y avait des régions distinctes au sein du nanodisque, et à la fois plus de fluctuations et plus de stabilité que prévu.
Bien qu'on suppose souvent que l'influence du mouvement d'une protéine membranaire est limitée aux molécules lipidiques qui l'entourent directement, les chercheurs ont observé des fluctuations plus spectaculaires sur une plage plus large, a déclaré Smith. Les fluctuations prirent la forme d’un doigt, comme de la bave éclaboussée sur un mur. Pourtant, même après un mouvement aussi spectaculaire, le nanodisque reviendrait à sa configuration normale.
"Le fait que nous ayons vu ces domaines, et que nous les ayons vu se rétablir de ces processus, suggère que les interactions entre la protéine et la membrane ont en réalité une portée plus grande qu'on ne le pense généralement", a déclaré Smith.
Les chercheurs prévoient d'utiliser leur technique de vidéographie électronique pour étudier d'autres types de protéines membranaires et d'autres classes de molécules et de nanomatériaux.
"Nous pourrions étudier les canaux ioniques qui s'ouvrent et se ferment pour réguler le flux et les interactions cellule à cellule en utilisant cette plateforme", a déclaré Chen.
Qian Chen est également affilié au département de chimie, au Beckman Institute for Advanced Science and Technology, au Carle Illinois College of Medicine et au Materials Research Laboratory de l'Illinois.
Plus d'informations : John W. Smith et al, Vidéographie électronique d'un tango lipide-protéine, Science Advances (2024). DOI :10.1126/sciadv.adk0217
Informations sur le journal : Progrès scientifiques
Fourni par l'Université de l'Illinois à Urbana-Champaign