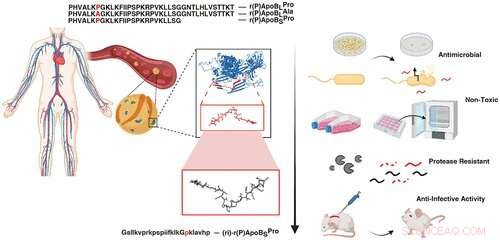
Résumé graphique. Crédit :DOI :10.1021/acsnano.1c04496
L'augmentation des infections par des bactéries résistantes aux médicaments est l'un des problèmes de santé mondiaux les plus graves au monde, dont on estime qu'il causera 10 millions de décès par an d'ici 2050. Certains des pathogènes bactériens les plus virulents et résistants aux antibiotiques sont la principale cause de maladies potentiellement mortelles. , infections nosocomiales, particulièrement dangereuses pour les patients immunodéprimés et gravement malades. La synthèse traditionnelle et continue d'antibiotiques ne pourra tout simplement pas suivre l'évolution des bactéries.
Pour éviter le processus continu de synthèse de nouveaux antibiotiques pour cibler les bactéries au fur et à mesure de leur évolution, les ingénieurs de Penn se sont penchés sur une nouvelle ressource naturelle pour les molécules antibiotiques.
Une étude récente sur la recherche de peptides cryptés aux propriétés antimicrobiennes dans le protéome humain a localisé des antibiotiques naturels dans notre propre corps. En utilisant un algorithme pour identifier des séquences spécifiques dans notre code protéique, une équipe de chercheurs de Penn avec des collaborateurs, dirigée par César de la Fuente, professeur adjoint présidentiel en psychiatrie, bioingénierie, microbiologie et génie chimique et biomoléculaire, et Marcelo Torres, un post-doc dans le laboratoire de de la Fuente, ont pu localiser de nouveaux peptides, ou chaînes d'acides aminés, qui, une fois clivés, indiquaient leur potentiel à repousser les bactéries nocives.
Maintenant, dans une nouvelle étude publiée dans ACS Nano , l'équipe avec Angela Cesaro, l'auteur principal et post-doc dans le laboratoire de de la Fuente, ont identifié trois peptides antimicrobiens distincts dérivés d'une protéine dans le plasma humain et démontrent leurs capacités dans des modèles murins. Angela Cesaro a effectué une grande partie des activités pendant son doctorat. sous la direction de l'auteur correspondant, le professeur Angela Arciello, de l'Université de Naples Federico II. L'étude collaborative inclut également l'Université d'Utrecht aux Pays-Bas.
"Nous avons identifié le système cardiovasculaire comme un point chaud pour les antimicrobiens potentiels en utilisant une approche algorithmique", explique de la Fuente. "Ensuite, nous avons examiné de plus près une protéine spécifique dans le plasma."
L'apolipoprotéine B est une protéine du plasma sanguin qui transporte les lipides, tels que le cholestérol, dans tout le corps. Cependant, lorsque cette protéine est décomposée, ses blocs de construction peptidiques présentent des fonctions complètement différentes.
À l'aide de leur algorithme, l'équipe a isolé trois peptides de l'apolipoprotéine B et a testé leur capacité à repousser différents types de bactéries, y compris celles qui causent des infections à staphylocoques et des pneumonies.
Chacun des trois peptides a pu pénétrer la membrane cytoplasmique de la bactérie, tuer la cellule et empêcher la croissance des biofilms. De plus, lorsqu'ils sont utilisés conjointement les uns avec les autres ou avec des antibiotiques pharmaceutiques, leur effet antibiotique a augmenté de manière significative, nécessitant une dose plus faible pour combattre l'infection.
L'équipe a également évalué si ces peptides favorisent la résistance aux antibiotiques chez ces bactéries.
"Il existe de nombreuses façons dont nos cellules immunitaires et nos peptides antimicrobiens attaquent et combattent les infections bactériennes", explique de la Fuente. "Ce qui est unique dans les peptides que nous examinons, c'est leur capacité à attaquer la membrane bactérienne, une structure qui nécessite la construction et le maintien de plusieurs gènes. Les antibiotiques typiques ne ciblent qu'un seul gène ou aspect des cellules bactériennes, ce qui permet aux bactéries de développer relativement facilement une résistance. , donc les antimicrobiens tels que les peptides que nous décrivons ici qui attaquent plusieurs cibles à la fois réussissent mieux à entraver la résistance bactérienne."
"Dans l'expérience d'évolution de la résistance que nous avons réalisée dans notre laboratoire, il était surprenant de voir à quelle vitesse de nouvelles bactéries résistantes aux antibiotiques courants sont sélectionnées et, au contraire, comment les peptides cryptés découverts dans le plasma ne conduisent pas à ce type de sélection. ", explique Cesaro. "Ce comportement pourrait provenir d'un mécanisme de défense de l'hôte développé chez l'homme et conservé de manière évolutive au fil du temps. Ce travail ouvre de nouvelles voies pour la découverte d'antimicrobiens dans des protéines non liées au système immunitaire et c'est très excitant car à l'heure actuelle, de nouveaux antibiotiques sont indispensables. ."
En fait, ce sont les propriétés physicochimiques de la membrane bactérienne elle-même qui permettent aux peptides d'être si efficaces dans ce combat.
"Les peptides agissent rapidement sur les membranes des bactéries envahissantes par différents mécanismes", explique Torres. "Dans ce cas, les membranes bactériennes agissent comme des aimants, attirant les peptides antimicrobiens, et parce que ces propriétés membranaires sont complexes et ne peuvent pas être facilement modifiées pour éviter l'attraction des peptides, les bactéries sont par conséquent vaincues par les peptides antimicrobiens et détruites, avec de multiples obstacles dans le façon de développer une quelconque résistance dans la prochaine génération."
L'élimination du potentiel de résistance signifie que ces peptides pourraient être utilisés comme antibiotique pour un large éventail d'infections bactériennes et rester efficaces plus longtemps que les antibiotiques traditionnels.
Enfin, pour augmenter la stabilité pour tester la fonction antimicrobienne in vivo, un peptide a été conçu, synthétisé et utilisé dans un modèle murin. L'expérience a montré une infection bactérienne cutanée traitée avec le peptide synthétique basé sur les antibiotiques naturels identifiés à partir de l'apolipoprotéine B, qui a pu éradiquer l'infection en quatre jours avec une seule dose.
"Le sang était un endroit clair pour rechercher des peptides cryptés tels que déterminés par l'algorithme, et ces résultats fournissent un lien entre les protéines plasmatiques humaines et notre immunité innée", explique de la Fuente. "Nous continuons à rechercher ces peptides au-delà du sang, dans tous les autres sites du corps pour fournir également ce lien avec les systèmes nerveux, digestif et immunitaire." Des chercheurs montrent que les peptides "cryptés" pourraient être une source d'antibiotiques naturels