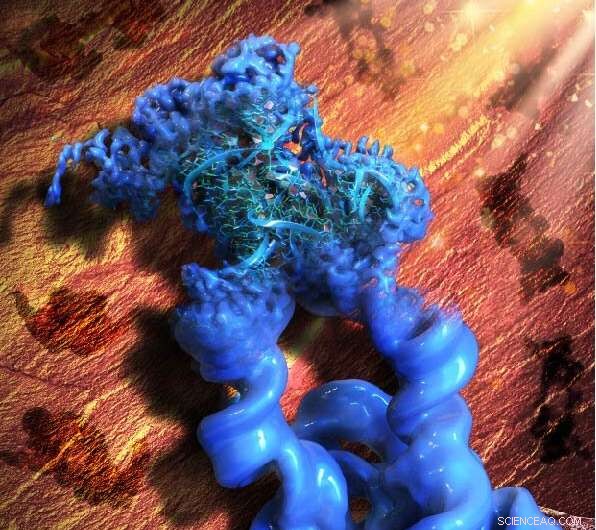
Cette illustration est inspirée de la peinture rupestre paléolithique de la grotte de Lascaux, signifiant l'acronyme de notre méthode, ROCK. Au sens figuré, les motifs de l'art rupestre à l'arrière-plan (marron) sont les projections 2D de la construction dimérique modifiée de l'intron du groupe I de Tetrahymena, tandis que l'objet principal à l'avant (bleu) est la carte cryo-EM 3D reconstruite du dimère, avec un monomère au point et raffiné à la haute résolution qui a permis aux collaborateurs de construire un modèle atomique de l'ARN. Crédit :Wyss Institute de l'Université de Harvard
Nous vivons dans un monde créé et dirigé par l'ARN, le frère tout aussi important de la molécule génétique ADN. En fait, les biologistes de l'évolution émettent l'hypothèse que l'ARN existait et s'auto-répliquait avant même l'apparition de l'ADN et des protéines codées par celui-ci. Avance rapide vers l'homme moderne :la science a révélé que moins de 3 % du génome humain est transcrit en molécules d'ARN messager (ARNm) qui, à leur tour, sont traduites en protéines. En revanche, 82% de celui-ci est transcrit en molécules d'ARN avec d'autres fonctions, dont beaucoup restent encore énigmatiques.
Pour comprendre ce que fait une molécule d'ARN individuelle, sa structure 3D doit être déchiffrée au niveau de ses atomes constitutifs et de ses liaisons moléculaires. Les chercheurs ont régulièrement étudié les molécules d'ADN et de protéines en les transformant en cristaux régulièrement emballés qui peuvent être examinés avec un faisceau de rayons X (cristallographie aux rayons X) ou des ondes radio (résonance magnétique nucléaire). Cependant, ces techniques ne peuvent pas être appliquées aux molécules d'ARN avec presque la même efficacité car leur composition moléculaire et leur flexibilité structurelle les empêchent de former facilement des cristaux.
Maintenant, une collaboration de recherche dirigée par Peng Yin, Ph.D., membre de la Wyss Core Faculty. au Wyss Institute for Biologically Inspired Engineering de l'Université de Harvard, et Maofu Liao, Ph.D. à la Harvard Medical School (HMS), a fait état d'une approche fondamentalement nouvelle de l'étude structurelle des molécules d'ARN. ROCK, comme on l'appelle, utilise une technique nanotechnologique d'ARN qui lui permet d'assembler plusieurs molécules d'ARN identiques en une structure hautement organisée, ce qui réduit considérablement la flexibilité des molécules d'ARN individuelles et multiplie leur poids moléculaire. Appliquée à des ARN modèles bien connus avec différentes tailles et fonctions comme références, l'équipe a montré que leur méthode permet l'analyse structurelle des sous-unités d'ARN contenues avec une technique connue sous le nom de cryo-microscopie électronique (cryo-EM). Leur avance est rapportée dans Méthodes Nature .
« ROCK brise les limites actuelles des investigations structurelles de l'ARN et permet de déverrouiller les structures 3D des molécules d'ARN qui sont difficiles ou impossibles d'accès avec les méthodes existantes, et à une résolution quasi atomique », a déclaré Yin, qui, avec Liao, a dirigé l'étude. . "Nous nous attendons à ce que cette avancée revigore de nombreux domaines de la recherche fondamentale et du développement de médicaments, y compris le domaine en plein essor des thérapies à base d'ARN." Yin est également un chef de file de l'initiative de robotique moléculaire de l'Institut Wyss et professeur au département de biologie des systèmes au HMS.
Gagner le contrôle de l'ARN
L'équipe de Yin à l'Institut Wyss a été la pionnière de diverses approches qui permettent aux molécules d'ADN et d'ARN de s'auto-assembler en grandes structures basées sur différents principes et exigences, y compris les briques d'ADN et l'origami d'ADN. Ils ont émis l'hypothèse que de telles stratégies pourraient également être utilisées pour assembler des molécules d'ARN naturelles dans des complexes circulaires hautement ordonnés dans lesquels leur liberté de flexion et de mouvement est fortement limitée en les liant spécifiquement les unes aux autres. De nombreux ARN se replient de manière complexe mais prévisible, avec de petits segments appariés les uns aux autres. Le résultat est souvent un "noyau" stabilisé et des "boucles de tige" bombées vers la périphérie.
"Dans notre approche, nous installons des 'boucles de baiser' qui relient différentes tiges-boucles périphériques appartenant à deux copies d'un ARN identique de manière à permettre la formation d'un anneau stabilisé global, contenant plusieurs copies de l'ARN d'intérêt", a déclaré Di Liu, Ph.D., l'un des deux premiers auteurs et boursier postdoctoral du groupe de Yin. "Nous avons émis l'hypothèse que ces anneaux d'ordre supérieur pourraient être analysés avec une haute résolution par cryo-EM, qui avait été appliqué aux molécules d'ARN avec un premier succès."
Imager l'ARN stabilisé
En cryo-EM, de nombreuses particules individuelles sont congelées instantanément à des températures cryogéniques pour empêcher tout autre mouvement, puis visualisées avec un microscope électronique et à l'aide d'algorithmes de calcul qui comparent les différents aspects des projections de surface 2D d'une particule et reconstruisent son architecture 3D. . Peng et Liu ont fait équipe avec Liao et son ancien étudiant diplômé François Thélot, Ph.D., l'autre co-premier auteur de l'étude. Liao et son groupe ont apporté d'importantes contributions à l'évolution rapide du domaine de la cryo-EM et à l'analyse expérimentale et informatique de particules uniques formées par des protéines spécifiques.
"Cryo-EM présente de grands avantages par rapport aux méthodes traditionnelles pour voir les détails à haute résolution des molécules biologiques, y compris les protéines, les ADN et les ARN, mais la petite taille et la tendance mobile de la plupart des ARN empêchent la détermination réussie des structures d'ARN. Notre nouvelle méthode d'assemblage de multimères d'ARN résout ces deux problèmes en même temps, en augmentant la taille de l'ARN et en réduisant son mouvement », a déclaré Liao, qui est également professeur agrégé de biologie cellulaire au HMS. "Notre approche a ouvert la porte à la détermination rapide de la structure de nombreux ARN par cryo-EM." L'intégration de la nanotechnologie de l'ARN et des approches cryo-EM a conduit l'équipe à nommer sa méthode "Cryo-EM activé par l'oligomérisation de l'ARN via l'installation de boucles de baiser" (ROCK).
Pour fournir une preuve de principe pour ROCK, l'équipe s'est concentrée sur un grand ARN d'intron de Tetrahymena, un organisme unicellulaire, et un petit ARN d'intron d'Azoarcus, une bactérie fixatrice d'azote, ainsi que le soi-disant FMN riboswitch . Les ARN d'intron sont des séquences d'ARN non codantes dispersées dans les séquences d'ARN fraîchement transcrits et doivent être «épissées» pour que l'ARN mature soit généré. Le riboswitch FMN se trouve dans les ARN bactériens impliqués dans la biosynthèse des métabolites de flavine dérivés de la vitamine B2. En se liant à l'un d'eux, la flavine mononucléotide (FMN), il change sa conformation 3D et supprime la synthèse de son ARN mère.
"L'assemblage de l'intron du groupe I de Tetrahymena dans une structure en forme d'anneau a rendu les échantillons plus homogènes et a permis l'utilisation d'outils informatiques tirant parti de la symétrie de la structure assemblée. Bien que notre ensemble de données soit de taille relativement modeste, les avantages innés de ROCK nous ont permis pour résoudre la structure à une résolution sans précédent », a déclaré Thélot. "Le noyau de l'ARN est résolu à 2,85 Å [un Ångström est un dix milliards (US) de mètre et la métrique préférée utilisée par les biologistes structurels], révélant des caractéristiques détaillées des bases nucléotidiques et du squelette de sucre. Je ne pense pas que nous aurait pu y arriver sans ROCK, ou du moins pas sans beaucoup plus de ressources."
Cryo-EM est également capable de capturer des molécules dans différents états si, par exemple, elles modifient leur conformation 3D dans le cadre de leur fonction. En appliquant ROCK à l'ARN de l'intron d'Azoarcus et au ribocommutateur FMN, l'équipe a réussi à identifier les différentes conformations par lesquelles passe l'intron d'Azoarcus au cours de son processus d'auto-épissage, et à révéler la rigidité conformationnelle relative du site de liaison au ligand du ribocommutateur FMN. .
"Cette étude de Peng Yin et de ses collaborateurs montre avec élégance comment la nanotechnologie de l'ARN peut fonctionner comme un accélérateur pour faire avancer d'autres disciplines. Être capable de visualiser et de comprendre les structures de nombreuses molécules d'ARN naturelles pourrait avoir un impact considérable sur notre compréhension de nombreux facteurs biologiques et pathologiques. processus à travers différents types de cellules, tissus et organismes, et même permettre de nouvelles approches de développement de médicaments », a déclaré le directeur fondateur de Wyss, Donald Ingber, M.D., Ph.D. De nouvelles connaissances sur les structures et les mécanismes des protéines clés impliquées dans la photosynthèse microbienne