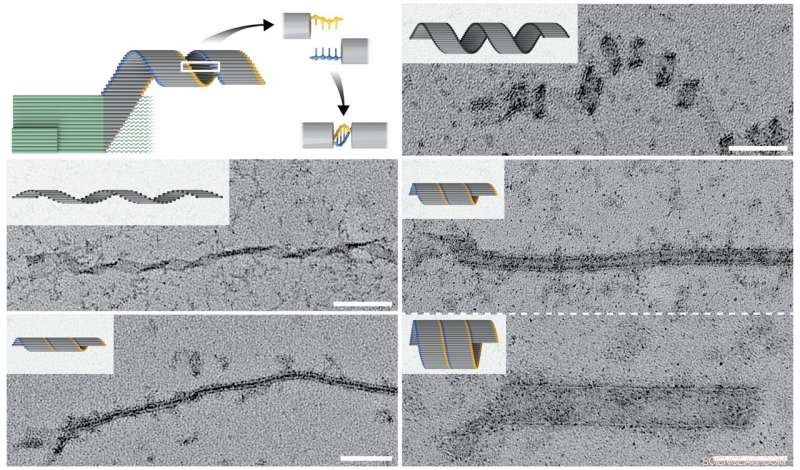
La polymérisation croisée strictement dépendante des graines (vertes) permet la formation de tubes et de rubans enroulés de formes diverses (gris), dans lequel des rubans allongés de différents diamètres sont fermés selon des motifs différents par de courts surplombs d'ADN simple brin complémentaires (jaune et bleu). Cette série d'images MET montre une variété de nanoconstructions allongées avec la barre d'échelle mesurant 100 nanomètres. Crédit :Institut Wyss de l'Université Harvard
Une équipe de nanobiotechnologues du Wyss Institute for Biologically Inspired Engineering de Harvard et du Dana-Farber Cancer Institute (DFCI) dirigée par William Shih, membre fondateur du corps professoral de Wyss, Doctorat., a conçu une stratégie d'auto-assemblage d'ADN programmable qui résout le défi clé du contrôle robuste de la nucléation et ouvre la voie à des applications telles que la détection de biomarqueurs de diagnostic ultrasensible et la fabrication évolutive de structures de taille micrométrique avec des caractéristiques de taille nanométrique.
En utilisant la méthode, appelée "polymérisation croisée", les chercheurs peuvent initier le tissage de nanorubans à partir de simples brins d'ADN allongés (appelés « lamelles ») par un événement de nucléation strictement dépendant des graines. L'étude est publiée dans Communication Nature .
Les nanostructures d'ADN ont un grand potentiel pour résoudre divers diagnostics, thérapeutique, et les défis de fabrication en raison de leur biocompatibilité et programmabilité élevées. Pour faire fonctionner des appareils de diagnostic efficaces, par exemple, une nanostructure d'ADN pourrait avoir besoin de répondre spécifiquement à la présence d'une molécule cible en déclenchant une lecture amplifiée compatible avec des instruments à faible coût accessibles dans les établissements de soins ou de laboratoire clinique.
La plupart des nanostructures d'ADN sont assemblées en utilisant l'une des deux stratégies principales qui ont chacune leurs forces et leurs limites. Les "origami d'ADN" sont formés à partir d'un long brin d'échafaudage monocaténaire qui est stabilisé dans une configuration bidimensionnelle ou tridimensionnelle par de nombreux brins d'agrafes plus courts. Leur assemblage est strictement dépendant du brin d'échafaudage, conduisant à un pliage tout ou rien robuste. Bien qu'ils puissent être formés avec une grande pureté dans une large gamme de conditions, leur taille maximale est limitée. Les "briques d'ADN" quant à elles peuvent assembler des structures beaucoup plus grandes à partir d'une multitude de brins modulaires courts. Cependant, leur assemblage nécessite des conditions environnementales rigoureusement contrôlées, peut être faussement initiée en l'absence d'une graine, et produit une proportion importante de structures incomplètes qui doivent être purifiées.
"L'introduction de l'origami d'ADN a été l'avancée la plus percutante dans le domaine de la nanotechnologie de l'ADN au cours des deux dernières décennies. L'approche de polymérisation croisée que nous avons développée dans cette étude s'appuie sur ce fondement et sur d'autres pour étendre échelles de longueur plus grandes, " dit Shih, qui co-dirige l'Initiative de robotique moléculaire de Wyss, et est également professeur à la Harvard Medical School et à la DFCI. "Nous envisageons que la polymérisation croisée permettra largement la formation tout ou rien de microstructures bidimensionnelles et tridimensionnelles avec des caractéristiques nanométriques adressables, auto-assemblage algorithmique, et l'amplification du signal sans bruit de fond dans les applications de diagnostic qui nécessitent une sensibilité extrême."
Planter une graine
Ayant expérimenté les limites des nanostructures d'origami d'ADN et de brique d'ADN, l'équipe a commencé par demander s'il était possible de combiner la dépendance absolue des semences de l'assemblage d'origami d'ADN avec la taille illimitée des constructions de briques d'ADN dans un troisième type de nanostructure d'ADN qui se développe rapidement et de manière cohérente jusqu'à une grande taille.
"Nous avons fait valoir que l'assemblage tout ou rien de structures d'ADN à l'échelle du micron pouvait être réalisé en concevant un système doté d'une barrière d'énergie libre élevée à l'assemblage spontané. La barrière ne peut être contournée qu'avec une graine qui se lie et organise un ensemble de lattes de « nucléation » pour la capture conjointe des lattes de « croissance ». " a déclaré le co-premier auteur Dionis Minev, Doctorat., qui est boursier postdoctoral dans l'équipe de Shih.
"Ce type de très coopératif, la nucléation strictement dépendante des graines suit certains des mêmes principes régissant l'initiation et la croissance de l'actine cytosquelettique ou des filaments de microtubules dans les cellules. à son tour est nécessaire pour la liaison du suivant. « La polymérisation entrecroisée fait passer cette stratégie à un niveau supérieur en permettant à des voisins non les plus proches d'être requis pour le recrutement des monomères entrants. Le niveau extrême de coordination qui en résulte est la sauce secrète, " dit Minev.
Du concept à la ou aux structures réelles
Mettre leur concept en pratique, l'équipe a conçu et validé un système dans lequel une structure de graine minuscule offre une concentration de départ élevée de sites de liaison préformés sous la forme de brins d'ADN uniques en saillie. Ceux-ci peuvent être détectés par des lattes d'ADN avec six (ou dans un système croisé alternatif huit) sites de liaison disponibles, chacun se liant à l'un des six (ou huit) brins d'ADNsb saillants voisins selon un motif entrecroisé, et les lattes d'ADN suivantes sont ensuite ajoutées en continu à la structure d'allongement.
"Notre conception est remarquable car nous avons atteint une croissance rapide d'énormes structures d'ADN, mais avec un contrôle de la nucléation qui est d'ordres de grandeur supérieur à d'autres approches. C'est comme avoir son gâteau et le manger aussi, parce que nous avons facilement créé des assemblages à grande échelle et ne l'avons fait que où et quand nous le voulions, " a déclaré le co-premier auteur Chris Wintersinger, un doctorat étudiant dans le groupe de Shih qui a collaboré sur le projet avec Minev. "Le contrôle que nous avons obtenu avec l'entrecroisement dépasse largement celui observé pour les méthodes d'ADN existantes où la nucléation ne peut être dirigée que dans une fenêtre étroite de conditions où la croissance est extrêmement lente."
En utilisant la polymérisation croisée, L'équipe de Shih a généré des rubans d'ADN qui se sont auto-assemblés à la suite d'un seul événement d'ensemencement spécifique dans des structures mesurant jusqu'à des dizaines de micromètres de long, avec une masse presque cent fois plus grande qu'un origami d'ADN typique. De plus, en tirant parti de la grande programmabilité des conformations et des interactions des lamelles, les chercheurs ont créé des rubans avec des tours et des torsions distincts, résultant en des structures enroulées et en forme de tube.
Dans les études futures, cela pourrait être exploité pour créer des structures fonctionnalisées qui peuvent bénéficier de compartiments spatialement séparés. "Une application immédiate de notre méthode de nanoconstruction entrecroisée est une stratégie d'amplification dans les tests de diagnostic suite à la formation de nanograines à partir de biomarqueurs spécifiques et rares, " a déclaré la co-auteur Anastasia Ershova, qui est également docteur. étudiant encadré par Shih.
« Le développement de cette nouvelle méthode de nanofabrication est un exemple frappant de la façon dont l'Initiative de robotique moléculaire du Wyss Institute continue de s'inspirer des systèmes biologiques, dans ce cas, filaments du cytosquelette en croissance, et continue d'élargir les possibilités dans ce domaine passionnant. Cette avancée rapproche le potentiel de la nanotechnologie de l'ADN pour résoudre des défis diagnostiques urgents pour lesquels il n'existe actuellement aucune solution, " a déclaré le directeur fondateur de Wyss, Donald Ingber, MARYLAND., Doctorat., qui est également le professeur Judah Folkman de biologie vasculaire à la Harvard Medical School et au Boston Children's Hospital, et professeur de bio-ingénierie à la Harvard John A. Paulson School of Engineering and Applied Sciences.