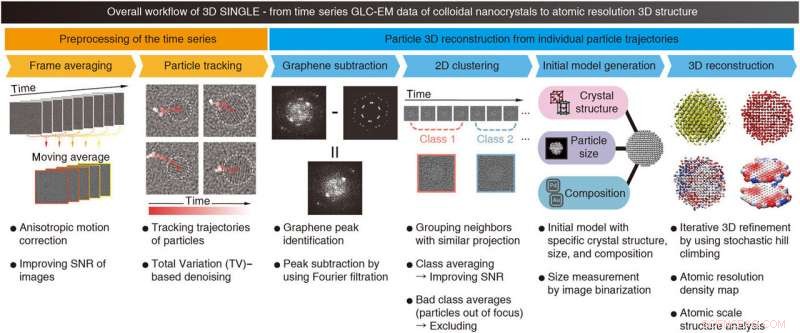
Flux de travail global de SINGLE. Le flux de travail global et les descriptions de chaque étape de SINGLE sont affichés. SINGLE se compose de deux étapes principales :Le prétraitement de la série temporelle (orange), comprenant (i) le calcul de la moyenne des trames de la fenêtre temporelle avec correction de mouvement anisotrope et (ii) le suivi de la trajectoire des particules avec l'utilisation d'un débruitage basé sur la variation totale (TV), et reconstruction 3D de particules à partir de trajectoires de particules individuelles (bleu), comprenant (i) l'identification et la soustraction de fond de graphène, (ii) clustering 2D limité dans le temps avec exclusion des images floues, (iii) génération initiale du modèle, et (iv) reconstruction 3D et analyse de structure à l'échelle atomique. Crédit :Avancées scientifiques, doi:10.1126/sciadv.abe6679
Les scientifiques des matériaux utilisent généralement la microscopie électronique à transmission (MET) en phase solution pour révéler les propriétés physicochimiques uniques des structures tridimensionnelles (3-D) des nanocristaux. Dans un nouveau rapport sur Avancées scientifiques , Cyril F. Reboul et une équipe de recherche de l'Université Monash, Australie, Université Nationale de Seoul, Corée du Sud, et le Lawrence Berkeley National Laboratory aux États-Unis, a développé une méthode de reconstruction 3D brownienne à particule unique. Pour y parvenir, ils ont imagé des ensembles de nanocristaux colloïdaux à l'aide de la microscopie électronique à transmission à cellule liquide de graphène. L'équipe a obtenu des images de projection de nanocristaux ayant subi une rotation différente à l'aide d'un détecteur d'électrons direct pour obtenir un ensemble de reconstructions 3D. Dans ce travail, ils ont introduit des méthodes de calcul pour reconstruire avec succès des nanocristaux 3D à une résolution atomique et y sont parvenus en suivant des particules individuelles au fil du temps, tout en soustrayant le bruit de fond perturbateur. La méthode pourrait également identifier/rejeter des images de faible qualité pour faciliter des stratégies personnalisées pour l'alignement 2-D/3-D qui différaient de celles de la cryomicroscopie biologique. L'équipe a rendu les développements disponibles via un progiciel open source connu sous le nom de SINGLE. Le logiciel gratuit est disponible sur GitHub.
Utilisation de SINGLE pour la cristallographie
Les chercheurs ont soutenu les progrès de la cristallographie au cours des 50 dernières années pour transformer la compréhension existante de la chimie et de la biologie. Néanmoins, certaines cibles, y compris les nanocristaux solubilisés, restent intraitables par les méthodes cristallographiques traditionnelles. Par exemple, les nanocristaux colloïdaux contiennent des dizaines à des centaines d'atomes et maintiennent une variété d'applications dans des domaines multidisciplinaires, notamment l'électronique, catalyse et capteurs biologiques. Les polyvalences découlent de la grande sensibilité des propriétés des nanocristaux à la taille, composition chimique et d'autres variables au cours de la synthèse. Typiquement, les scientifiques utilisent une seule particule, Reconstruction 3-D en biologie structurale pour déterminer la structure des protéines. La technique est relativement nouvelle pour la reconstruction 3-D in-situ de nanocristaux individuels solubilisés. Dans ce travail, Reboul et al. développé UNIQUE; une méthode qui reposait sur la reconstruction 3D indépendante de nanocristaux individuels solubilisés, y compris le mouvement brownien. La technique est une première avancée dans l'étude pour résoudre les structures atomiques 3D des nanocristaux directement à partir de la phase de solution.
Suivi des trajectoires individuelles des nanocristaux. Suivi des résultats des particules 1 (A et B) et 2 (C et D) tout au long du film (bleu à rouge). Des moyennes temporelles représentatives d'images de particules brutes non alignées (50 images) sont présentées (B et D). Barres d'échelle, 1 nanomètre. Crédit :Avancées scientifiques, doi:10.1126/sciadv.abe6679 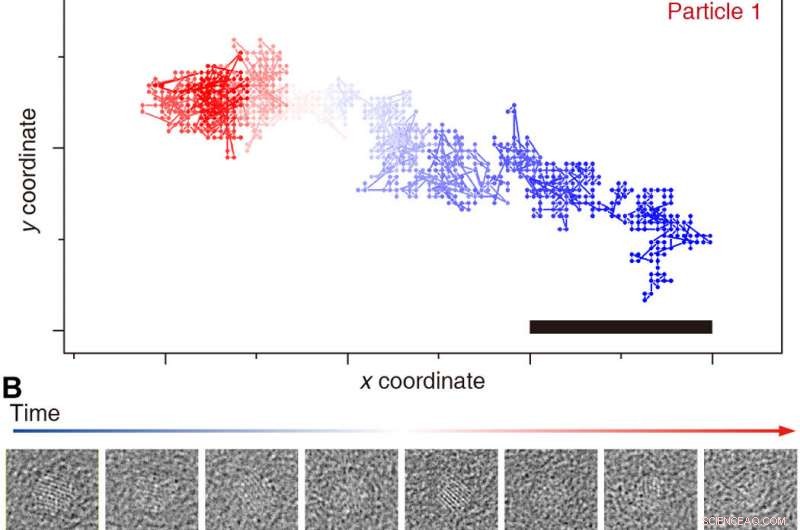
Les scientifiques ont introduit de nouvelles méthodes de prétraitement pour améliorer le rapport signal sur bruit (SNR) afin de suivre les trajectoires des particules tout en supprimant le signal de fond induit par le graphène. Des méthodes de calcul avancées pourraient réussir à reconstruire en 3D à partir des données de microscopie électronique à transmission de cellules de graphène liquide (GLC) in situ. Par rapport aux techniques existantes, le travail a présenté l'applicabilité d'une méthode de calcul sans précédent pour obtenir des reconstructions 3D à résolution atomique pour les nanocristaux dispersés en solution. Ils ont divisé le flux de travail SINGLE en deux étapes principales (1) le prétraitement et (2) la reconstruction 3-D des particules. Les scientifiques visaient à fournir les performances et l'efficacité les plus élevées possibles sur n'importe quel matériel CPU, y compris les superordinateurs aux postes de travail ou même aux ordinateurs portables.
En premier, l'équipe a fait la moyenne de la fenêtre temporelle sur plusieurs images avec un mouvement anisotrope pour améliorer le rapport signal/bruit, conduisant à des particules visibles et à un signal de graphène amélioré. L'équipe a ensuite identifié les positions des particules manuellement dans la moyenne de la première fenêtre temporelle. Après, l'équipe a développé un modèle de départ basé sur la structure cristallographique attendue, diamètre des particules et des éléments constitutifs et produit des reconstructions 3D avec des coordonnées atomiques adaptées pour l'analyse de la structure à l'échelle atomique.
Clustering 2D limité dans le temps. (A) Fraction du changement angulaire tout au long de la série chronologique. La ligne pointillée rouge est la ligne de tendance. Les directions de projection changent rapidement dans les régions entre les images 1500 à 1600 (orange), trames 3800 à 3900 (vert), et cadres 5600 à 5700 (bleu). Les encarts sont des tracés représentant la différence angulaire dans la direction de projection dans ces régions. (B) Représentation schématique de la moyenne de classe 2D limitée dans le temps. (C) Graphique montrant les classes allouées pour les trames individuelles dans la région 1 à 400. L'encart est un tracé montrant les classes allouées sur toutes les images. (D) Moyennes de classe obtenues avec un regroupement et un alignement 2D limités dans le temps. Crédit :Avancées scientifiques, doi:10.1126/sciadv.abe6679 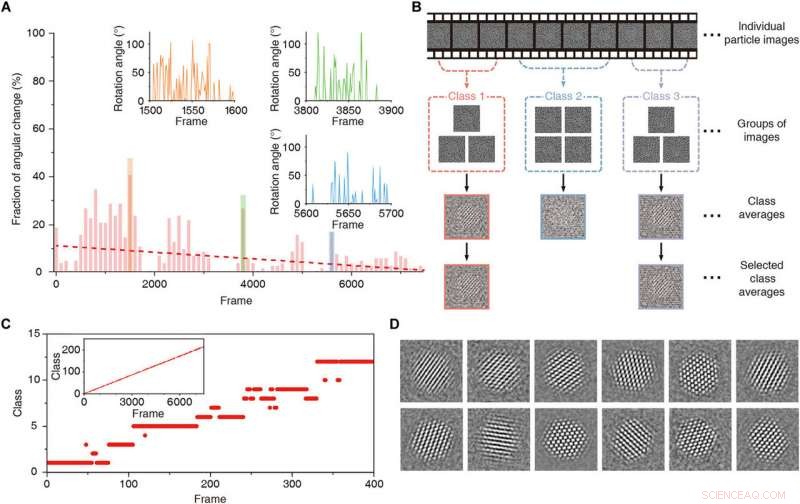
Reboul et al. introduit une nouvelle méthode de suivi utilisant des transformées de Fourier rapides et la corrélation de phase pour identifier un maximum de corrélation avec une précision inférieure au pixel. L'équipe a débruité la fenêtre temporelle d'extraction en utilisant un débruitage basé sur la variation totale (TV) et un débruitage combiné et une moyenne temporelle pour fournir une méthode robuste pour suivre le mouvement des nanocristaux individuels dans l'échantillon. La méthode leur a permis de discerner la forme globale des nanocristaux et/ou leurs caractéristiques cristallines, ce qui atteste de la robustesse de l'algorithme de suivi. En utilisant la méthode, ils ont également récupéré des trajectoires auparavant difficiles pour obtenir des reconstructions 3D et ont utilisé une trajectoire de particules soustraite en arrière-plan dans toutes les étapes de traitement d'image pour la soustraction de graphène du GLC (cellule liquide de graphène). L'équipe a en outre caractérisé la nature des rotations des nanocristaux dans l'espace très confiné de la cellule liquide de graphène. La méthode n'était pas triviale en raison de la nature probabiliste de l'algorithme de reconstruction 3-D. L'équipe a donc intégré une approche déterministe pour améliorer la précision du cluster, tout en améliorant le rapport signal sur bruit par rapport aux images individuelles.
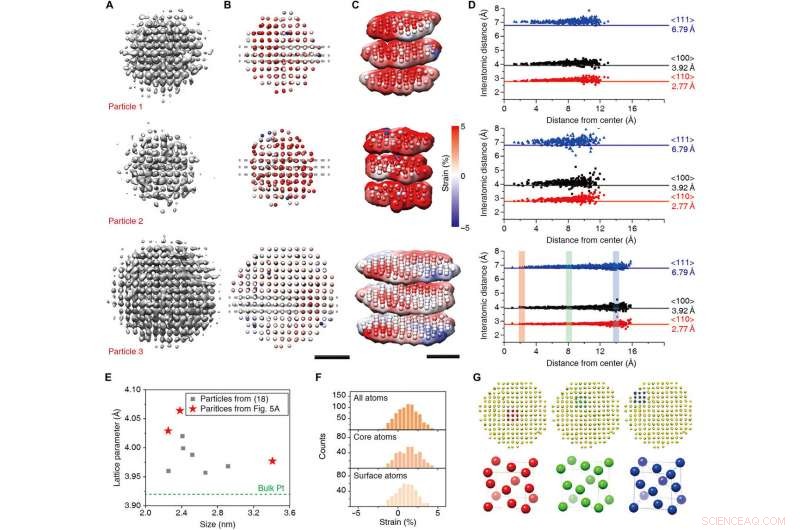
Résultats de reconstruction 3D et analyse de la structure au niveau atomique. (A à C) Cartes de densité 3D (A), cartes de déformation radiale à partir des coordonnées atomiques ajustées (B), et leur représentation en tranches (C). (D) Distances interatomiques dans les directions de <110> (rouge), <100> (le noir), et <111> (bleu) pour trois nanocristaux. (E) Paramètres de réseau ajustés des nanocristaux précédemment rapportés (18) (carrés gris) et nouveaux résultats (étoiles rouges). (F) Histogramme de déformation radiale de tous les atomes (en haut), atomes de noyau (milieu), atomes de surface (en bas) de la particule 3. (G) Structure cellulaire unitaire du noyau (rouge), milieu (vert), et surface (bleu) de la particule 3. Barres d'échelle, 1 nanomètre. Crédit :Avancées scientifiques, doi:10.1126/sciadv.abe6679
Génération de modèles
Les chercheurs ont ensuite développé un modèle de départ en utilisant la connaissance que les particules ont un arrangement de position atomique approximativement cubique. Ils ont simulé les densités atomiques en utilisant des facteurs de diffusion atomique 5-gaussiens. Les projections 2D de la densité 3D simulée représentaient le caractère des projections au cœur du nanocristal, pour surmonter les problèmes liés à la symétrie translationnelle et à un signal de fond brouilleur. La méthode de raffinement 3-D utilisée pour la cryomicroscopie biologique n'a pas pu être directement appliquée aux données de séries chronologiques de nanocristaux; donc, Reboul et al. introduit des modifications critiques. Ils ont utilisé un schéma de raffinement en deux étapes pour établir la forme correcte du nanocristal pour permettre aux atomes et à leurs formes de piloter l'alignement 3-D. Les chercheurs ont choisi trois nanocristaux de différentes tailles qui n'avaient pas été reconstruits auparavant pour l'analyse comparative, puis en utilisant des cartes atomiques produites avec la méthode, Reboul et al. obtenu des détails structurels microscopiques au niveau atomique. Le travail a également facilité les cartes atomiques détaillant l'analyse des contraintes et l'analyse de la structure cellulaire unitaire.
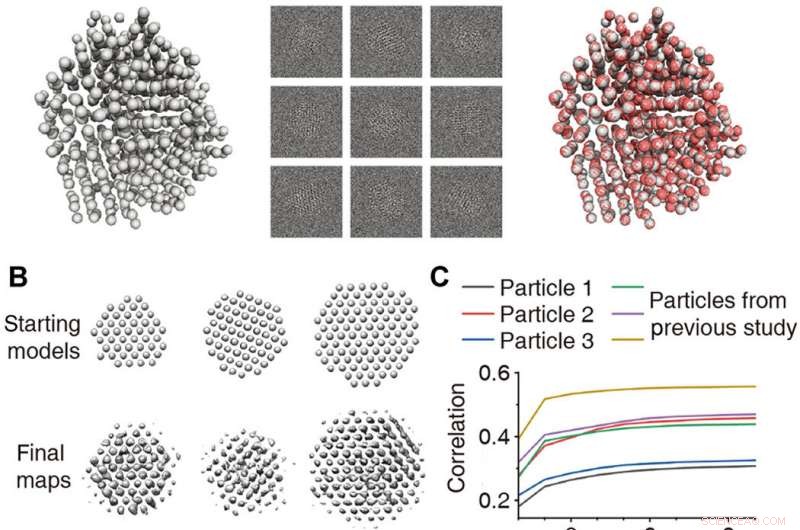
Validation des reconstructions 3D. (A) Reconstruction 3D d'une particule désordonnée simulée avec une structure atomique connue. A gauche :Modèle d'un nanocristal désordonné obtenu par simulation de dynamique moléculaire. Milieu :5000 images simulées multitranches avec bruit ajouté pour donner un SNR =0,1. À droite :carte atomique (rouge) résultant d'une reconstruction 3D superposée au modèle de vérité terrain (gris). (B) Modèles 3D de départ et cartes de densité 3D finales obtenues à partir de données expérimentales. (C) Corrélation entre les reprojections de la carte de densité 3D raffinée et les vues de particules expérimentales tracées en fonction de l'itération pour la première étape du raffinement 3D. Les particules présentées dans cet article (noir, rouge, et bleu) et présenté dans une étude précédente (18) (vert, violet, et couleur ocre) sont tracés. (D) Comparaison des moyennes des classes (indiquées comme projection) avec des reprojections pour la validation des trois structures. (E) Représentation atomique dépendante du temps des directions de projection pour les trois structures :blanc (début) à rose (milieu) à rouge (fin). Rouge, jaune, et les flèches bleues indiquent x, oui, et axes z, respectivement. Crédit :Avancées scientifiques, doi:10.1126/sciadv.abe6679
Validation des reconstructions 3D
Les chercheurs ont en outre généré un modèle de nanocristal désordonné à l'aide de simulations de dynamique moléculaire pour comprendre l'applicabilité de SINGLE aux nanocristaux hautement désordonnés. À l'aide de simulations multi-tranches, ils ont appliqué un mouvement de translation et des variations de défocalisation aléatoires pour représenter le mouvement réaliste des particules. Ils ont ensuite obtenu une carte de densité 3D du nanocristal désordonné à partir de 500 images simulées avec un rapport signal sur bruit de 0,1 et un modèle de départ avec un ordre cristallin parfait pour s'accorder parfaitement avec les particules d'origine. L'équipe a obtenu la distribution des directions de projection des nanocristaux en rotation pour valider la qualité de la reconstruction 3D et nécessitera d'autres études pour comprendre comment les structures atomiques réelles des nanocristaux affectent la dynamique de rotation.
De cette façon, Cyril F. Reboul et ses collègues ont démontré des méthodes de calcul dans SINGLE pour obtenir des cartes de densité de nanocristaux à résolution atomique. En utilisant une configuration avancée de cellules liquides telles que des cellules liquides de graphène avec des nanochambres ordonnées, l'équipe a permis de contrôler l'épaisseur du liquide pour étendre l'applicabilité de SINGLE pour une acquisition de données efficace. La suite SINGLE a fourni une première plate-forme analytique efficace pour comprendre l'origine structurelle des propriétés physiques et chimiques uniques des nanocristaux dans leur phase de solution native.
© 2021 Réseau Science X