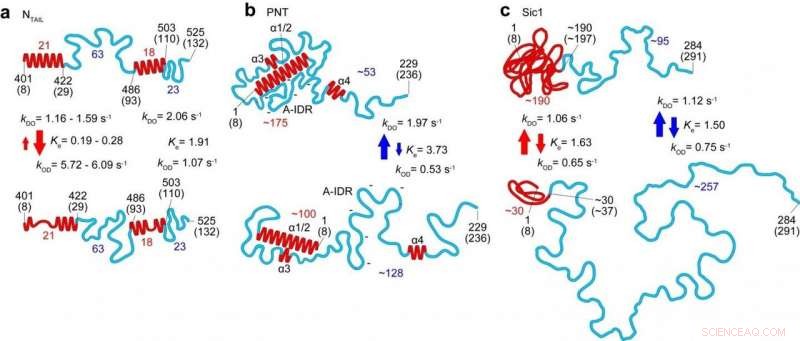
Caractéristiques structurelles et dynamiques de trois PDI (N
Notre compréhension des protéines biologiques n'est pas toujours en corrélation avec leur fréquence ou leur importance. La moitié de toutes les protéines, molécules qui jouent un rôle essentiel dans les processus cellulaires, sont intrinsèquement désordonnés, ce qui signifie que de nombreuses techniques standard pour sonder les biomolécules ne fonctionnent pas sur elles. Aujourd'hui, des chercheurs de l'Université de Kanazawa au Japon ont montré que leur technologie de microscopie à force atomique à grande vitesse peut fournir des informations non seulement sur les structures de ces protéines, mais aussi sur leur dynamique.
Comprendre comment une protéine est assemblée fournit des indices précieux sur ses fonctions. Le développement de la cristallographie des protéines dans les années 1930 et 1950 a mis en évidence pour la première fois plusieurs structures protéiques, mais il est progressivement devenu évident qu'une grande partie des protéines manque d'une structure unique, ce qui les rend intraitables pour la cristallographie aux rayons X. Comme ils sont trop minces pour la microscopie électronique, les seules alternatives viables pour bon nombre de ces protéines intrinsèquement désordonnées (IDP) sont l'imagerie par résonance magnétique nucléaire et la diffusion des rayons X aux petits angles. Les données recueillies à partir de ces techniques sont moyennées sur des ensembles et ne donnent donc aucune indication claire des conformations individuelles des protéines ou de la fréquence à laquelle elles se produisent. D'autre part, la microscopie à force atomique est capable d'imagerie biologique à résolution nanométrique à grande vitesse, il peut donc capturer la dynamique ainsi que les structures des protéines.
Dans ce dernier travail des chercheurs de l'Université de Kanazawa aux côtés de collaborateurs au Japon, La France et l'Italie ont appliqué la technique à l'étude de plusieurs PDI et identifié des paramètres définissant la forme, taille et longueur de chaîne des régions protéiques, ainsi qu'une loi de puissance reliant la taille de la protéine à la longueur de la protéine, et une description quantitative de l'effet de la surface du mica sur les dimensions des protéines. La dynamique des conformations protéiques capturées grâce aux capacités à grande vitesse de la technique a révélé des globules qui apparaissent et disparaissent, et des transformations entre des conformations totalement non structurées et faiblement repliées dans des segments jusqu'à 160 acides aminés de long.
Les études de la nucléoprotéine du virus de la rougeole en particulier ont permis d'identifier non seulement la forme et les dimensions mais aussi les caractéristiques des transitions ordre-désordre dans la région responsable de la reconnaissance moléculaire, qui permet aux virus d'identifier les facteurs de l'hôte afin qu'ils puissent se reproduire. Ils pourraient également déterminer des structures à plus grande échelle de la phosphoprotéine du virus qui ne sont pas accessibles à la résonance magnétique nucléaire (qui ne peut donner qu'une indication des distances entre les acides aminés séparés de moins de 2 nm). Les chercheurs suggèrent que la formation de certaines formes compactes observées pourrait expliquer la résistance à la protéolyse, c'est-à-dire la dégradation des protéines.
Dans leur rapport sur les travaux, les chercheurs soulignent qu'en plus d'être un outil puissant à part entière, "Lorsque toutes les caractéristiques moléculaires révélées par HS-AFM sont combinées avec la structure locale repliée donnée par RMN, les informations combinées permettent une délimitation quantitative des caractères structurels et dynamiques des personnes déplacées, d'une manière plus réaliste par rapport aux images représentées individuellement, comme démontré pour le PNT [la phosphoprotéine du virus de la rougeole]. »
Microscopie à force atomique à grande vitesse
La microscopie à force atomique a été développée dans les années 1980 et a apporté la résolution à l'échelle atomique obtenue par la microscopie à effet tunnel (qui a remporté le prix Nobel de physique 1986) aux échantillons non conducteurs. Il fonctionne à l'aide d'un petit porte-à-faux avec une pointe nanométrique à l'extrémité, qui sent la surface un peu comme une aiguille de disque vinyle ou la tape. Que ce soit en ajustant la hauteur de la pointe ou la fréquence de résonance du tapotement, the interactions between tip and surface provide a signal that can be used to generate an image.
While AFM images brought huge benefits to biological research, these studies were able to move up a gear again when Toshio Ando and his team at Kanazawa University reported an atomic force microscope that operated at high speed. Atomic scale resolution images became movies bringing not just structures but also dynamics within grasp. Previous work on ordered proteins, which are reasonably well understood, as well as the IDP facilitates chromatin transcription (FACT) protein, has established that the technique can be used to image these biomolecules without effects from contact between tip and sample distorting the data.
Intrinsically disordered proteins
The arrival of xray crystallography gave researchers a clear view of vast numbers of biomolecule structures for the first time. But with the hundreds of thousands of biomolecule structures analyzed using protein crystallography since the technique first came into use in the 1930s and 1950s, a mounting body of evidence began to build that not all proteins have a single set structure. The observations ran counter to the prevailing paradigm of protein function determined by a fixed structure.
Over the past ten to twenty years the ubiquity of these intrinsically disorderd proteins and their importance in cell functions from signaling to the regulation of transcription and subsequent translation has become widely recognized. In the current work the researchers study IDPs including polyglutamine tract binding protein-1 (PQBP-1, involved in different processes, such as pre-mRNA splicing, transcription regulation, innate immunity and neuron development), autophagy proteins (which are invovolved in removing dysfunctional cell components) containing intrinsically disordered regions (IDRs) and the measles virus nucleoprotein.