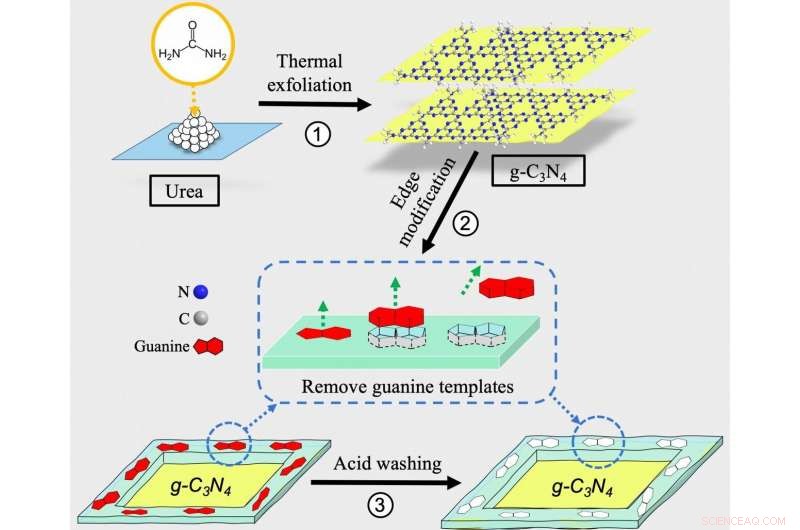
Un schéma montre la méthode en trois étapes pour produire des nanofeuilles de nitrure de carbone graphitique à empreinte moléculaire. Le processus développé par les chercheurs de l'Université Rice pourrait aider à capturer et à tuer les gènes résistants aux antibiotiques flottants trouvés dans les effluents secondaires produits par les stations d'épuration. Crédit :Danning Zhang/Université Rice
Il ne suffit pas d'éliminer les bactéries résistantes aux antibiotiques des eaux usées pour éliminer les risques qu'elles représentent pour la société. Les morceaux qu'ils laissent derrière eux doivent également être détruits.
Des chercheurs de la Brown School of Engineering de l'Université Rice ont une nouvelle stratégie pour « piéger et zapper » les gènes résistants aux antibiotiques, les morceaux de bactéries qui, même si leurs hôtes sont morts, peuvent se frayer un chemin dans et augmenter la résistance d'autres bactéries.
L'équipe dirigée par l'ingénieur environnemental de Rice Pedro Alvarez utilise des nanofeuilles de nitrure de carbone graphitique à empreinte moléculaire pour absorber et dégrader ces restes génétiques dans les eaux usées des systèmes d'égouts avant qu'ils n'aient la possibilité d'envahir et d'infecter d'autres bactéries.
Les chercheurs ont ciblé des gènes de résistance aux antibiotiques (ARG) codés dans un plasmide codant pour la métallo-bêta-lactamase 1 (NDM1) de New Delhi, connu pour résister à plusieurs médicaments. Lorsqu'il est mélangé en solution avec les ARG et exposé à la lumière ultraviolette, les nanofeuillets traités se sont avérés 37 fois plus efficaces pour détruire les gènes que le nitrure de carbone graphitique seul.
Les travaux effectués sous les auspices du Rice-based Nanosystems Engineering Research Center for Nanotechnology-Enabled Water Treatment (NEWT) sont détaillés dans la revue American Chemical Society Sciences et technologies de l'environnement .
« Cette étude répond à une préoccupation croissante, l'émergence de bactéries multirésistantes appelées superbactéries, " dit Alvarez, directeur du Centre ASPIC. "Ils devraient causer 10 millions de décès par an d'ici 2050.
"En tant qu'ingénieur en environnement, Je crains que certaines infrastructures hydrauliques puissent abriter des superbactéries, " dit-il. " Par exemple, une station d'épuration à Tianjin que nous avons étudiée est un vivier, déchargeant cinq souches NDM1-positives pour chacune d'entre elles. Le bassin d'aération est comme un hôtel de luxe où toutes les bactéries se développent.
"Malheureusement, certaines superbactéries résistent à la chloration, et les bactéries résistantes qui meurent libèrent des ARG extracellulaires qui se stabilisent par l'argile dans les milieux récepteurs et transforment les bactéries indigènes, devenir des réservoirs résistomes. Cela souligne le besoin d'innovation technologique, pour empêcher la décharge d'ARG extracellulaires.
"Dans ce document, nous discutons d'une stratégie trap-and-zap pour détruire les ARG extracellulaires. Notre stratégie consiste à utiliser des revêtements à empreinte moléculaire qui améliorent la sélectivité et minimisent les interférences des composés organiques de fond."

A gauche, une image au microscope électronique à balayage montre la structure mésoporeuse de nanofeuillets de nitrure de carbone graphitique à empreinte moléculaire. A droite, une image au microscope électronique à transmission montre le bord de la feuille et sa structure cristalline. Des chercheurs de l'Université Rice ont imprimé les nanofeuillets pour capturer et tuer les gènes flottants résistants aux antibiotiques trouvés dans les effluents secondaires produits par les stations d'épuration. Crédit :Alvarez Research Group/Rice University
L'empreinte moléculaire, c'est comme fabriquer une serrure qui attire une clé, un peu comme les enzymes naturelles avec des sites de liaison qui ne s'adaptent qu'aux molécules de la bonne forme. Pour ce projet, les molécules de nitrure de carbone graphitique sont le verrou, ou photocatalyseur, personnalisé pour absorber puis détruire NDM1.
Pour faire le catalyseur, les chercheurs ont d'abord enduit les bords des nanofeuilles d'un polymère, acide méthacrylique, et de la guanine incorporée. "La guanine est la base d'ADN la plus facilement oxydée, " dit Alvarez. " La guanine est ensuite lavée avec de l'acide chlorhydrique, qui laisse son empreinte. Cela sert de site d'adsorption sélective pour l'ADN environnemental (eDNA)."
Danning Zhang, étudiant diplômé de Rice, co-auteur principal de l'article, ledit nitrure de carbone a été choisi pour les nanofeuillets de base car il n'est pas métallique et est donc plus sûr à utiliser, et pour sa disponibilité facile.
Alvarez a noté que tous les catalyseurs sont efficaces pour éliminer les ARG de l'eau distillée, mais pas aussi efficace dans les effluents secondaires, un produit des stations d'épuration après élimination des solides et des composés organiques.
« Dans les effluents secondaires, vous avez des piégeurs d'espèces réactives de l'oxygène et d'autres composés inhibiteurs, " Alvarez a déclaré. "Cette stratégie piège et zap améliore considérablement l'élimination du gène eDNA, surpassant clairement les photocatalyseurs commerciaux."
Les chercheurs ont écrit que les procédés de désinfection conventionnels utilisés dans les usines de traitement des eaux usées, y compris la chloration et le rayonnement ultraviolet, sont modérément efficaces pour éliminer les bactéries résistantes aux antibiotiques, mais relativement inefficaces pour éliminer les ARG.
Ils espèrent que leur stratégie pourra être adaptée à l'échelle industrielle.
Zhang a déclaré que le laboratoire n'avait pas encore effectué de tests approfondis sur d'autres ARG. "Comme la guanine est un constituant commun de l'ADN, et donc les ARG, cette approche devrait également dégrader efficacement d'autres eARG, " il a dit.
Il est possible d'améliorer le processus actuel, malgré son extraordinaire succès initial. « Nous n'avons pas encore tenté d'optimiser le matériau photocatalytique ou le procédé de traitement, ", a déclaré Zhang. "Notre objectif est d'offrir une preuve de concept que l'empreinte moléculaire peut améliorer la sélectivité et l'efficacité des processus photocatalytiques pour cibler les eARG."
Qingbin Yuan de l'Université technique de Nanjing, Chine, est co-auteur principal de l'article. Les co-auteurs sont les étudiants diplômés de Rice Ruonan Sun et Hassan Javed, et Gang Wu, professeur adjoint d'hématologie au Centre des sciences de la santé de l'Université du Texas à la faculté de médecine McGovern de Houston. Pingfeng Yu, chercheur postdoctoral à Rice, est co-auteur. Alvarez est professeur George R. Brown de génie civil et environnemental et professeur de chimie et de génie chimique et biomoléculaire.