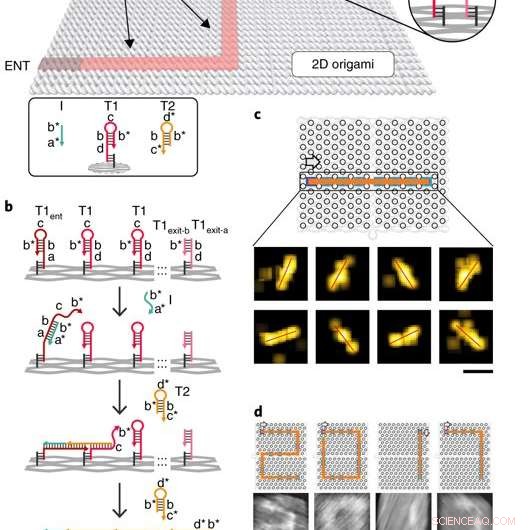
Implémentation du navigateur d'ADN à molécule unique. a) Illustration schématique du système PSEC. Un graphe connecté acyclique (c'est-à-dire un arbre) est construit sur un substrat d'origami d'ADN rectangulaire de 100 x 70 nm^2. T1 et T2 sont deux types de carburants entraînant la PSEC sur l'arbre. L'initiateur I a été utilisé pour déclencher l'initiation de la PSEC à partir du sommet d'entrée ENT. b) mécanisme de propagation du système PSEC. L'initiateur I reconnaît et ouvre l'épingle à cheveux T1ent au sommet ENT. L'épingle à cheveux ouverte capture et ouvre ensuite une épingle à cheveux T2 de l'environnement pour démarrer la cascade. c) Une ligne droite pavée par PSEC et visualisée avec DNA-PAINT. d) Quatre chiffres pavés de « 2017 » imagés par microscopie à force atomique (AFM). Les épingles à cheveux T1 aux points d'angle ont été modifiées pour éviter les travées indésirables. Les flèches indiquent le sens de propagation. Crédit: Matériaux naturels , doi:10.1038/s41563-018-0205-3.
Le domaine de la nanorobotique intelligente repose sur la grande promesse des dispositifs moléculaires dotés de capacités de traitement de l'information. Dans une nouvelle étude qui soutient la tendance des supports d'information basés sur l'ADN, les scientifiques ont conçu un système de navigateur d'ADN qui peut exécuter une seule molécule, parallèle, des opérations de recherche en profondeur sur une plate-forme d'origami en deux dimensions.
Les opérations de recherche de chemin avec des navigateurs d'ADN utilisent un processus en cascade d'échange de brins localisé initié sur un site de déclenchement unique sur la plate-forme d'origami. La progression automatique le long des chemins est rendue possible par des épingles à cheveux d'ADN contenant une séquence de traversée universelle. Intentionnellement, chaque navigateur à molécule unique peut explorer de manière autonome n'importe lequel des chemins possibles à travers un arbre enraciné à 10 sommets construit dans l'étude. Les labyrinthes équivalaient à un arbre avec une entrée à la racine et une sortie par l'une des feuilles. L'étude menée par Jie Chao et ses collègues a permis d'explorer tous les chemins empruntés par les navigateurs ADN pour extraire un chemin de solution spécifique qui connectait une paire donnée de sommets de début et de fin dans le labyrinthe. Par conséquent, le chemin de la solution a été posé clairement sur la plate-forme d'origami et illustré à l'aide d'une imagerie à molécule unique. L'approche est maintenant publiée dans Matériaux naturels , détaillant la réalisation de matériaux moléculaires avec des fonctions de calcul biomoléculaires intégrées pour fonctionner au niveau de la molécule unique avec le potentiel de concevoir des nanorobots intelligents pour des applications futures dans l'industrie et la médecine.
Des outils moléculaires sophistiqués ont été utilisés dans le passé pour créer des machines moléculaires qui convertissent les produits chimiques, l'énergie photonique ou électrique en mouvements rotatifs ou linéaires à l'échelle nanométrique. Par exemple, Le mouvement brownien à l'échelle nanométrique peut être converti de manière contrôlée en mouvements dirigés au sein de nanomachines à base d'ADN à l'aide de réactions d'hybridation d'ADN. De telles machines basées sur l'ADN fonctionnent de manière autonome en suivant un « programme moléculaire » intégré préconçu comme une réaction en cascade déclenchée manuellement via un stimulus externe pour chaque étape de l'opération.
L'objectif du domaine s'est progressivement déplacé pour actualiser des circuits logiques basés sur l'ADN en utilisant des aptamères et des ADNzymes pour concevoir des portes logiques moléculaires. Par exemple, en 2006, Stojanovic et ses collègues ont intégré plus de 100 portes logiques ADN pour concevoir une automatisation appelée MAYA-II pour jouer à un jeu de Tic-Tac-Toe. Les études précédentes ont démontré un système informatique sans enzyme basé sur des réactions en chaîne d'hybridation (HCR) pour créer des portes logiques et des circuits logiques pour des performances plus robustes et efficaces que les systèmes d'origine.
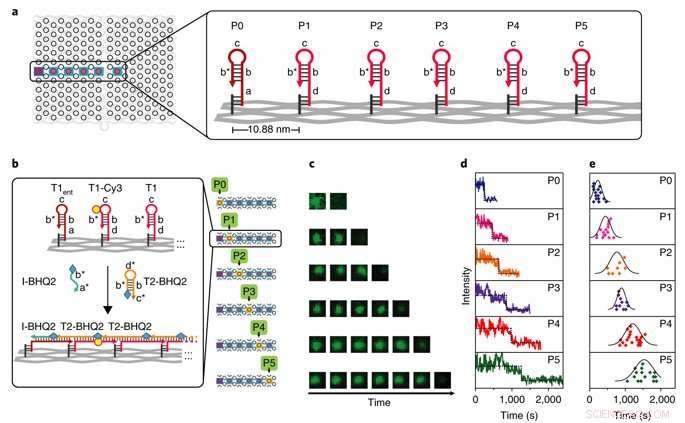
Caractérisation d'une molécule unique de la cinétique de la PSEC. a) Une ligne droite au milieu de l'origami d'ADN a été utilisée comme banc d'essai. Le sommet P0 est le point de départ et les sommets P1-P5 sont des points intermédiaires. b) Détails de la conception de six tests parallèles pour mesurer la cinétique avec TIRF résolue en temps. T2 marqué avec BHQ2 a été utilisé pour éteindre T1 marqué avec Cy3, assemblage d'un profil cinétique à chaque étape. L'exemple illustré montre le mécanisme de trempe. c) Des exemples d'images TIRF montrent l'évolution de la fluorescence avec le temps sur les six tests parallèles de P0 à P5. d) Traces typiques de fluorescence à molécule unique utilisées pour surveiller les événements d'extinction se produisant aux sommets P0 à P5. e) Diagrammes de dispersion montrant une analyse statistique de la distribution des longueurs par trajet. Crédit: Matériaux naturels , doi:10.1038/s41563-018-0205-3.
Dans la présente étude de Chao et al, le même principe de base du schéma de réaction HCR a été utilisé dans un contexte informatique différent pour développer un système de navigateur d'ADN à molécule unique. La plate-forme a exploré tous les chemins possibles à travers un graphe arborescent conçu sur une structure en origami comme un labyrinthe simplement connecté sans chemins cycliques. De telles structures d'origami d'ADN sont par nature des nanostructures porteuses d'informations avec une géométrie à l'échelle nanométrique bien définie. Le labyrinthe pourrait être exploré par une cascade d'échange de brins proximale (PSEC) basée sur des réactions en chaîne d'hybridation. Les chercheurs ont démontré qu'un système avec un grand nombre de navigateurs d'ADN à molécule unique pouvait effectuer collectivement une recherche parallèle en profondeur d'abord (PDFS) sur l'arbre pour effectuer efficacement une résolution de labyrinthe dans un origami 2D. Initialement, les chercheurs ont mené des études pour tester la conception PSEC.
Le système de cascade d'échange de brins proximal (PSEC) (principe de fonctionnement du navigateur d'ADN) a été facilité sur un substrat d'origami rectangulaire composé de trois composants, qui comprenait la mise en œuvre physique d'un graphe arborescent, brins pleins et un brin initiateur. Les zones vacantes sans extensions d'agrafes correspondaient aux murs du labyrinthe, empêchant la propagation de la cascade d'échange de brins. The entrance and exit were defined and denoted as ENT and EXIT respectively. In the second component, two types of DNA hairpins, T1 and T2, were used as fuels to drive the PSEC on the tree graph. The two hairpins coexisted metastably in solution to hybridize and fuel the PSEC process with free energy .
Intentionnellement, information only propagated through the network in the presence of an initiator (Initiator I). Upon addition of initiator I, PSEC was conducted and observed using atomic force microscopy (AFM). To visualize an established formation, the scientists enabled DNA-navigator-based formation of the number 2017 as a proof-of-principle. Another technique known as DNA-PAINT was employed as a single-molecule, super-resolution imaging technique to reveal molecular features at the nanoscale to further substantiate the PSEC-based path paving process. The on-origami PSEC was highly specific, without intra- or inter-origami crosstalk.
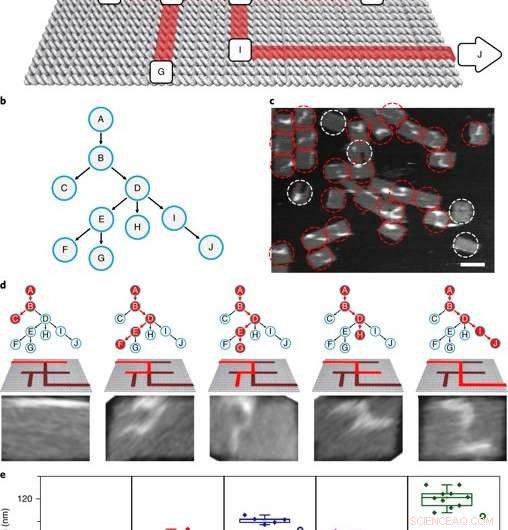
The PSEC-driven graph traversal on a maze. a) A maze design with 10 vertices. Arrows indicate the entrance vertex A and exit vertex J. b) The maze is equivalent to a rooted tree with 10 vertices. The entrance vertex A corresponds to the root of the tree. c) An AFM image showed the result of a transversal experiment generating all possible paths. In this DNA computing implementation of a PDFS algorithm, a vast number of PSEC events simultaneously occurred to realize the graph traversal on the maze. PSEC ending at the exit or deadends were highlighted in red circles. Invalid structures were highlighted with white circles. d) Typical paths found in the mixture seen from left to right. Only P
Kinetics of the process were investigated at the single-molecule level in the study using time-resolved total internal reflection fluorescence microscopy (TIRF) in a setup with a prescribed starting point (P0) and five intermediate steps (P1-P5). Fluorescence in the setup was quenched using fluorescence resonance energy transfer (FRET), and the cascade was observed in real time by recording fluorescence signals continuously. The average speed of propagation was recorded to be 2.46 nm per minute, propagation across the straight line (54.4 nm) took approximately 22 minutes.
The scientists then constructed the main model maze with 10 vertices that included an entrance vertex A and an exit vertex J, three junctions (B, RÉ, E) an intermediate vertex (I) and four dead ends (C, F, G, H). Each path of the maze that was equivalent to a tree with 10 vertices was investigated using DNA investigators starting at root A. The PSEC reactions produced a mixture of various paths on the maze, confirmed with AFM. Each individual PSEC could progress on one of the five possible paths. Statistical analysis of the length distribution showed that the measured paths coincided well with the values predicted.
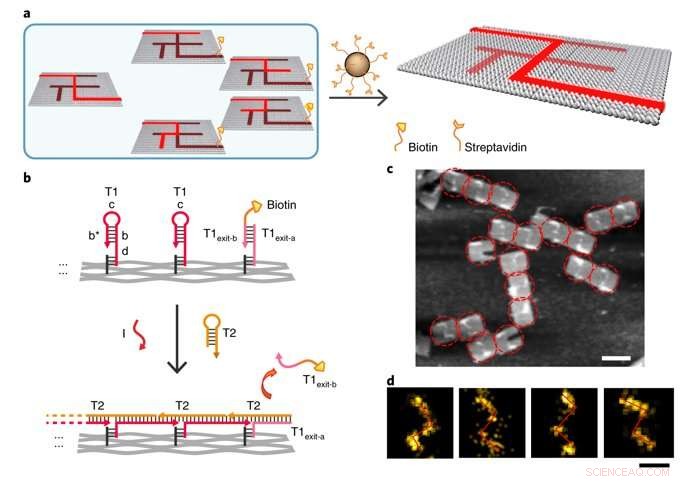
Single-molecule DNA navigators for maze-solving. a) Schematic illustration of magnetic bead-based selection. Exit vertex J is labelled with biotin to enable differentiation between the correct and wrong paths. Only if the PSEC reaches the correct exit J, would the biotin-modified T1exit-B strand be released. All wrong paths could be captured and removed by the Streptavidin-modified magmatic beads therefore. b) Details of the release of biotin modification at the exit J by the PSEC. c) AFM characterization of the correct solution after selection. The remaining structures all showed the correct solution path PABDIJ. d) Single-molecule and class-averaged DNA-PAINT characterization of the correct solution after selection. Crédit: Matériaux naturels , doi:10.1038/s41563-018-0205-3.
To prevent the navigators from propagating through a wrong path with dead ends, the scientists designed a streptavidin-biotin tag-based method to selectively eliminate inaccurate path navigation. Only the correct path (P
The main advantage of the described biomolecular computer schemes in comparison to conventional electronic computing is they can be interfaced directly with biologically relevant processes. Par conséquent, the scientists envision translational biomedical sensing and decision-making platforms with DNA origami and single-molecule diagnostics using decision trees. Future applications will also include simple sensors or those coupled to a molecular actuator to trigger downstream molecular cascades.
© 2018 Réseau Science X