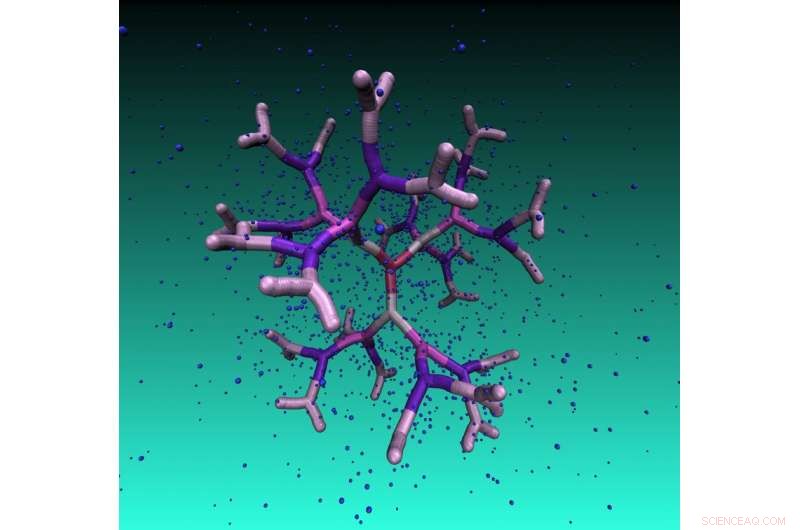
Un dendrimère à base d'ADN de cinquième génération dans une solution avec ses contre-ions. Des vides réguliers à l'intérieur de cette molécule peuvent être utilisés comme cages de transport pour les nanoparticules. Crédit :Université de technologie de Vienne
Comment créer des nanocages, c'est à dire., des objets robustes et stables avec des vides réguliers et des propriétés ajustables ? De courts segments de molécules d'ADN sont des candidats parfaits pour la conception contrôlable de nouvelles structures complexes. Physiciens de l'Université de Vienne, l'Université technique de Vienne, le Jülich Research Center en Allemagne et l'Université Cornell aux États-Unis, ont étudié des méthodologies pour synthétiser des dendrimères à base d'ADN en laboratoire et pour prédire leur comportement à l'aide de simulations informatiques détaillées. Leurs résultats sont publiés dans Nanoéchelle .
Les nanocages sont des constructions moléculaires très intéressantes, du point de vue à la fois de la science fondamentale et des applications possibles. Les cavités de ces objets de taille nanométrique peuvent être utilisées comme supports de molécules plus petites, qui est d'une importance critique en médecine pour l'administration de médicaments ou de gènes dans les organismes vivants. Cette idée a réuni des chercheurs de divers domaines interdisciplinaires qui ont étudié les dendrimères en tant que candidats prometteurs pour la création de tels nano-supports. Leur architecture arborescente et leur croissance par étapes avec des unités auto-similaires répétées donnent des dendrimères contenant des cavités, objets creux avec un design contrôlable. Néanmoins, des décennies de recherche ont montré qu'un grand nombre de types de dendrimères différents subissent un repliement arrière des branches externes avec des générations croissantes de dendrimères, donnant lieu à une densité plus élevée de constituants à l'intérieur de la molécule. L'effet de repliement est renforcé lors de l'ajout de sel dans la solution, où les dendrimères flexibles subissent un rétrécissement important, deviennent des objets compacts sans espaces creux à l'intérieur.
L'équipe de collaborateurs était composée de Nataša Adžić et Christos Likos (Université de Vienne), Clemens Jochum et Gerhard Kahl (TU Vienne), Emmanuel Stiakakis (Jülich) ainsi que Thomas Derrien et Dan Luo (Cornell). Les chercheurs ont trouvé un moyen de créer des dendrimères suffisamment rigides pour empêcher le repliement arrière des bras externes, même dans le cas de générations à forte ramification, préservant des vides réguliers dans leur intérieur. De plus, leurs nouvelles macromolécules se caractérisent par une résistance remarquable au sel ajouté :ils ont montré que la morphologie et les caractéristiques conformationnelles de ces systèmes restent inchangées même lors de l'ajout de sel, même à haute concentration. Les nanocages qu'ils ont créées, en laboratoire et étudiés informatiquement sont des dendrimères à base d'ADN, ou soi-disant, ADN de type dendrimère (DL-ADN). Le bloc de construction dont ils sont composés est une unité d'ADN double brin en forme de Y, une structure à trois bras constituée d'ADN double brin (ds-ADN), formé par hybridation de trois chaînes d'ADN simple brin (ADN-ss), dont chacun a des séquences partiellement complémentaires aux deux autres. Chaque bras est composé de 13 paires de bases et d'une extrémité collante simple brin avec quatre nucléobases qui agissent comme une colle. Alors qu'un seul ADN-Y correspond à la première génération de dendrimère, la fixation d'autres éléments d'ADN-Y donne l'ADN-DL des générations supérieures. Le dendrimère résultant est un assemblage macromoléculaire chargé et creux avec une architecture arborescente. En raison de la rigidité de l'ADNdb, les branches du DL-ADN sont rigides de sorte que toute la molécule est rigide. Puisque l'ADN est chargé, la répulsion électrostatique augmente la rigidité de la molécule.
DL-DNA molecules have been assembled in the laboratory by the Jülich and Cornell partners with remarkable control and sub-nanometer precision through programmable sticky-end cohesions. Their step-wise growth is highly controllable, unidirectional and non-reversible. This property is of high importance, as it has been shown that DNA-based dendrimers have been envisioned to play a promising role in developing nanoscale-barcodes, DNA-based vaccine technologies, as well as a structural probes involving multiplexed molecular sensing processes. Sizes, shapes as well as additional conformational details invisible to the experimentalists, such as the size of voids and the degree of branches back-folding, have been analyzed by computer simulations in Vienna. To describe the complex structure of DNA units, the group used a simple monomer-resolved model with interactions carefully chosen to mimic the equilibrium properties of DNA in physiological solution. The excellent agreement obtained between experiments and simulations for the dendrimer characteristics validates the theoretical models employed and paves the way for further investigation of the nanocages' properties and their applications as functional and smart nanocarriers and as building blocks for engineering biocompatible artificial materials.