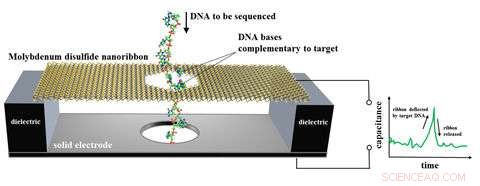
Conception proposée par le NIST pour un séquenceur d'ADN basé sur un capteur de mouvement électronique. Un ruban nanométrique de bisulfure de molybdène est suspendu sur une électrode métallique et immergé dans l'eau. ADN simple brin, contenant une chaîne de bases (bits de code génétique), est enfilé à travers un trou dans le ruban, qui ne fléchit que lorsqu'une base d'ADN s'apparie puis se sépare d'une base complémentaire fixée au trou. Le mouvement de la membrane est détecté comme un signal électrique. Des simulations numériques et des estimations théoriques montrent que la membrane serait précise de 79 à 86 pour cent pour identifier les bases d'ADN en une seule mesure à des vitesses allant jusqu'à environ 70 millions de bases par seconde. Crédit :NIST
Des chercheurs du National Institute of Standards and Technology (NIST) et des collaborateurs ont proposé une conception pour le premier séquenceur d'ADN basé sur un nanocapteur électronique capable de détecter de minuscules mouvements aussi petits qu'un seul atome.
Le dispositif proposé - un type de condensateur, qui stocke la charge électrique - est un petit ruban de bisulfure de molybdène suspendu au-dessus d'une électrode métallique et immergé dans l'eau. Le ruban mesure 15,5 nanomètres (nm, milliardièmes de mètre) de long et 4,5 nm de large. ADN simple brin, contenant une chaîne de bases (bits de code génétique), est enfilé à travers un trou de 2,5 nm de large dans le ruban mince. Le ruban ne fléchit que lorsqu'une base d'ADN s'apparie puis se sépare d'une base complémentaire fixée au trou. Le mouvement de la membrane est détecté comme un signal électrique.
Comme décrit dans un nouvel article, l'équipe du NIST a effectué des simulations numériques et des estimations théoriques pour montrer que la membrane aurait une précision de 79 à 86 pour cent pour identifier les bases d'ADN en une seule mesure à des vitesses allant jusqu'à environ 70 millions de bases par seconde. Les circuits intégrés détecteraient et mesureraient les signaux électriques et identifieraient les bases. Les résultats suggèrent qu'un tel appareil pourrait être un séquenceur d'ADN précis et économique, selon le papier.
Séquençage conventionnel, développé dans les années 1970, implique la séparation, copier, étiqueter et réassembler des morceaux d'ADN pour lire l'information génétique. Les méthodes plus récentes incluent le séquençage automatisé de nombreux fragments d'ADN à la fois - encore coûteux - et de nouveaux concepts de "séquençage nanopore". Par exemple, le même groupe du NIST a récemment démontré l'idée de séquencer l'ADN en le faisant passer à travers un nanopore de graphène, et mesurer comment les propriétés électroniques du graphène réagissent à la contrainte.
La dernière proposition du NIST repose sur un film mince de bisulfure de molybdène, un matériau stratifié qui conduit l'électricité et est souvent utilisé comme lubrifiant. Entre autres avantages, ce matériau ne colle pas à l'ADN, ce qui peut être un problème avec le graphène. L'équipe du NIST suggère que la méthode pourrait même fonctionner sans nanopore - une conception plus simple - en faisant passer l'ADN à travers le bord de la membrane.
"Cette approche résout potentiellement le problème de l'adhérence de l'ADN au graphène s'il est mal inséré, car cette approche n'utilise pas de graphène, période, " Le théoricien du NIST et auteur principal Alex Smolyanitsky a déclaré. " Une autre différence majeure est qu'au lieu de s'appuyer sur les propriétés du graphène ou de tout matériau particulier utilisé, nous lisons les mouvements électriquement d'une manière plus facile en formant un condensateur. Cela rend toute membrane électriquement conductrice adaptée à l'application."
L'expert en nanomatériaux Boris Yakobson de l'Université Rice, un co-auteur sur le papier, suggéré l'idée du condensateur. Le support informatique a été fourni par l'Université de Groningen aux Pays-Bas.
L'ADN a quatre bases. Pour les simulations, cytosine (C), qui s'associe naturellement à la guanine (G), est attaché à l'intérieur du pore. Lorsqu'un morceau d'ADN traverse le pore, tout G dans le brin s'attache temporairement au C incorporé, tirant sur le nanoruban et signalant l'électrode. La séquence d'ADN est déterminée en mesurant comment et quand les impulsions électriques varient dans le temps. Pour détecter les quatre bases, quatre nanorubans, chacun avec une base différente attachée au pore, pourraient être empilés verticalement pour créer un capteur d'ADN intégré.
Le ruban de bisulfure de molybdène est suffisamment flexible pour se déformer de manière mesurable en réponse aux forces nécessaires pour rompre une paire d'ADN, mais assez rigide pour en avoir moins en cours, mouvement sans signification que le graphène, potentiellement réduire le bruit indésirable dans les signaux de séquençage. La déviation du ruban est extrêmement faible, de l'ordre d'un angström, la taille d'un atome d'hydrogène. Sa force de traction est de l'ordre de 50 piconewtons, ou des billions de newton, assez pour rompre les liaisons chimiques délicates entre les bases de l'ADN.
Les chercheurs ont estimé les performances de l'appareil dans un circuit intégré et ont découvert que les courants de crête à travers le condensateur étaient mesurables (50 à 70 picoampères), même pour les petits nanorubans étudiés. Les pics actuels devraient être encore plus importants dans les systèmes physiques. La taille de l'appareil pourrait être modifiée pour rendre encore plus facile la mesure des signaux de séquençage.
Les auteurs du NIST espèrent construire une version physique de l'appareil à l'avenir. Pour des applications pratiques, la technologie microfluidique de séquençage de l'ADN de la taille d'une puce pourrait être combinée à l'électronique dans un seul appareil suffisamment petit pour être tenu dans la main.