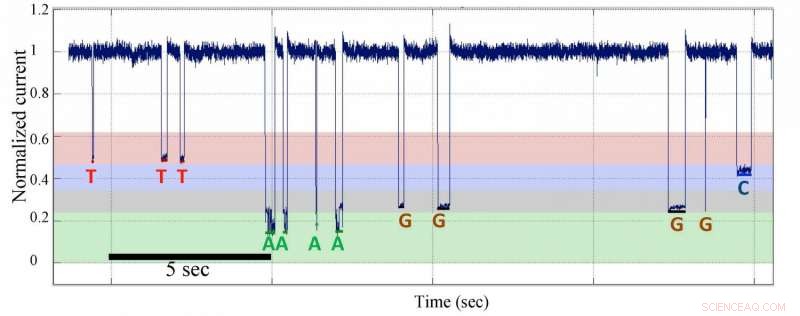
Séquençage d'ADN nanopore à molécule unique par des données de synthèse à partir d'un modèle avec des séquences d'homopolymères. Crédits :Jingyue Ju, Britannique Ingénierie
Des chercheurs de l'Université de Columbia, avec des collègues de Genia Technologies (Roche), L'Université de Harvard et le National Institute of Standards and Technology (NIST) rapportent avoir réalisé un séquençage électronique d'ADN à molécule unique en temps réel à une résolution de base unique à l'aide d'un réseau de nanopores de protéines.
Le séquençage de l'ADN est la technologie clé pour les initiatives de médecine personnalisée et de précision, permettant des découvertes rapides en science biomédicale. La séquence complète du génome d'un individu fournit des marqueurs et des lignes directrices importants pour le diagnostic médical, soins de santé, et le maintien d'une vie saine. À ce jour, le coût et la vitesse impliqués dans l'obtention de séquences d'ADN très précises ont été un défi majeur. Bien que divers progrès aient été réalisés au cours de la dernière décennie, les instruments de séquençage à haut débit largement utilisés aujourd'hui dépendent de l'optique pour la détection de quatre éléments constitutifs de l'ADN :A, C, G et T. Pour explorer des capacités de mesure alternatives, le séquençage électronique d'un ensemble de modèles d'ADN a été développé pour l'analyse génétique. Séquençage de brins nanopores, dans lequel un ADN simple brin est enfilé à travers les pores nanométriques sous une tension électrique appliquée pour produire des signaux électroniques pour la détermination de séquence au niveau d'une seule molécule, a été récemment développé; cependant, parce que les quatre nucléotides sont très similaires dans leurs structures chimiques, ils ne peuvent pas être facilement distingués à l'aide de cette méthode. Les chercheurs poursuivent donc activement la recherche et le développement d'une plate-forme précise de séquençage d'ADN électronique à molécule unique, car elle a le potentiel de produire un séquenceur d'ADN miniaturisé capable de déchiffrer le génome pour faciliter une médecine de précision personnalisée.
Une équipe de chercheurs de Columbia Engineering, dirigé par Jingyue Ju (Samuel Ruben-Peter G. Viele professeur d'ingénierie, Professeur de génie chimique et de pharmacologie, directeur du Center for Genome Technology &Biomolecular Engineering), avec des collègues de la Harvard Medical School, dirigé par George Church (professeur de génétique); Génie Technologies, dirigé par Stefan Roever (PDG de Genia); et John Kasianowicz, le chercheur principal au NIST, ont développé un système complet pour séquencer électroniquement l'ADN dans les nanopores au niveau d'une seule molécule avec une résolution à base unique. Ce travail, intitulé, "Séquençage d'ADN électronique à molécule unique en temps réel par synthèse à l'aide de nucléotides marqués par un polymère sur un réseau de nanopores, " est publié dans la revue, Actes de l'Académie nationale des sciences ( PNAS ) Première édition.
Précédemment, des chercheurs des laboratoires Ju à Columbia et Kasianowicz au NIST ont rapporté le principe général du séquençage des nanopores par synthèse (SBS), la faisabilité de la conception et de la synthèse de nucléotides marqués par un polymère comme substrats pour l'ADN polymérase, la détection et la différenciation des tags polymères par nanopore au niveau de la molécule unique [ Rapports scientifiques 2, 684 (2012) DOI :10.1038/srep00684]. Le courant PNAS L'article décrit la construction du système SBS nanopore complet pour produire des données de séquençage électronique à molécule unique avec une résolution à base unique. Cette stratégie SBS distingue avec précision quatre bases d'ADN en détectant et en différenciant électroniquement quatre étiquettes polymères différentes attachées au 5'-phosphate des nucléotides lors de leur incorporation dans un brin d'ADN en croissance catalysé par la polymérase, une enzyme de synthèse d'ADN. Les chercheurs ont conçu et synthétisé de nouveaux nucléotides marqués au niveau du phosphate terminal avec des polymères à base d'oligonucléotides pour effectuer des nanopores SBS sur une plate-forme de matrice de nanopores de protéine α-hémolysine. Les balises sur les nucléotides marqués par polymère, qui se sont avérés être des substrats actifs pour l'ADN polymérase, produire différents niveaux de blocage de courant électrique. Ils ont construit un réseau de nanopores sur une puce électronique portant plusieurs électrodes; le réseau est composé de canaux protéiques couplés à une ADN polymérase liée à une matrice d'ADN amorcée. L'ajout de nucléotides marqués par un polymère distincts et personnalisés à la matrice de nanopores déclenche la synthèse d'ADN. En bloquant le courant ionique du canal à différents niveaux, les balises distinctes fournissent une lecture de la séquence de modèles en temps réel avec une résolution à base unique.
Comme Carl Fuller, auteur principal, Chercheur principal adjoint au laboratoire Ju du département de génie chimique de Columbia et directeur de chimie à Genia, fait remarquer, "La nouveauté de notre approche SBS nanopore commence par la conception, synthèse, et la sélection de quatre nucléotides différents marqués par un polymère. Nous utilisons une ADN polymérase attachée de manière covalente au nanopore et aux nucléotides marqués pour effectuer le SBS. Lors de la réplication de l'ADN lié à la polymérase, l'étiquette de chaque nucléotide complémentaire est capturée dans le pore pour produire un signal électrique unique. Quatre étiquettes polymères distinctes produisant des signatures distinctes qui sont reconnues par le détecteur électronique dans la puce à matrice de nanopores sont utilisées pour la détermination de la séquence. Ainsi, Les séquences d'ADN sont obtenues pour de nombreuses molécules uniques en parallèle et en temps réel. Les quatre étiquettes en polymère sont conçues pour offrir de bien meilleures distinctions entre elles, contrairement aux petites différences entre les quatre nucléotides d'ADN natifs, surmontant ainsi le défi majeur auquel sont confrontées les autres méthodes de séquençage direct des nanopores. les balises peuvent être encore optimisées en termes de taille, charger, et structure pour fournir une résolution optimale dans le système SBS nanopore.
"Ce projet passionnant rassemble des scientifiques et des ingénieurs du monde universitaire et de l'industrie avec une expertise combinée en ingénierie moléculaire, nanotechnologie, génomique, l'électronique et la science des données pour produire des révolutionnaires, des plateformes de diagnostic génétique rentables avec un potentiel sans précédent pour la médecine de précision, " dit Ju. " Nous sommes extrêmement reconnaissants pour le soutien généreux du NIH qui nous a permis de faire des progrès rapides dans la recherche et le développement de la technologie nanopore SBS, et les contributions exceptionnelles de tous les membres de notre consortium de recherche."
Selon Ju, les chercheurs ont déjà poussé au-delà de ce qui a été démontré dans le PNAS étude où les données de séquençage ont été obtenues sur un premier prototype de séquenceur basé sur le nanopore SBS. Le débit et les performances du séquenceur actuel ont progressé au-delà de ce qui était indiqué dans le PNAS papier. La faisabilité d'atteindre des longueurs de lecture de plus de 1000 bases d'ADN a récemment été atteinte. Aller de l'avant, l'équipe de recherche collaborative continuera à optimiser les balises en peaufinant les linkers, structure, et charge au niveau moléculaire, et affiner la polymérase et l'électronique pour le système SBS nanopore dans le but de séquencer avec précision un génome humain entier rapidement et à faible coût, lui permettant ainsi d'être utilisé dans les diagnostics médicaux de routine.