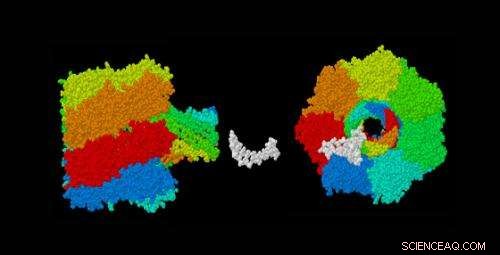
Il s'agit d'une image du pore d'alpha-hémolysine (constitué de 7 sous-unités identiques en 7 couleurs) et d'un ADN simple brin 12-mère (en blanc) à la même échelle pour illustrer les effets de l'ADN sur la conductance lors du déplacement à travers un nanopore. Ci-dessous, une vue orthogonale des mêmes molécules. Image créée du 5 octobre 2008 par George Church à l'aide de Rasmol_2.6 et des coordonnées de 7AHL.pdb et 1BNA.pdb Crédit :George Church
Il n'est pas facile de faire passer de longues chaînes de molécules - telles que l'ADN - à travers un "nanopore" (un pore qui ne fait que quelques millionièmes de millimètre de large) car elles ont tendance à s'emmêler. Une simulation réalisée par un groupe international de scientifiques - parmi lesquels des chercheurs de SISSA - a suggéré une solution :il vaut mieux "tirer" doucement sans appliquer trop de force, sinon, la molécule pourrait être arrêtée en raison d'un frottement excessif. C'est une observation importante pour créer des méthodes de séquençage de l'ADN innovantes.
Au fur et à mesure que la nanotechnologie progresse, il devient de plus en plus important de connaître en détail la dynamique du nanomonde (le monde à l'échelle du millionième de millimètre). Ce qui se produit, par exemple, quand on essaie de piloter un polyélectrolyte (une longue chaîne de molécules chargées électriquement, comme l'ADN) à travers un nanopore si des nœuds provoquent un blocage du processus de translocation ? Ce n'est pas une question inutile, car maintenant une nouvelle méthode de séquençage de l'ADN pour analyser électrochimiquement chaque brin en le faisant passer à travers un nanopore, en développement. Étant donné que ces brins ont tendance à s'emmêler s'ils sont très longs, Angelo Rosa de l'International School for Advanced Studies et ses collègues ont entrepris d'étudier théoriquement la dynamique de cette translocation, en réalisant une simulation.
Le modèle choisi par les scientifiques a montré que le coincement n'est pas causé par la simple présence du nœud, mais par la relation entre le frottement et la force appliquée pour entraîner la molécule dans l'espace. "Le résultat n'est pas si évident si on le compare à ce qui se passe à un niveau macro, " a expliqué Cristian Micheletti, chercheur à SISSA et l'un des auteurs de l'article publié dans Lettres d'examen physique . "Les nœuds introduisent un frottement efficace qui augmente avec la force appliquée et tire le polymère de l'autre côté du nanopore. La translocation n'est arrêtée qu'au-dessus d'une force seuil".
« D'après ce que nous avons observé dans la simulation, pour éviter l'obstruction du pore et l'arrêt de la translocation, la force appliquée doit être contrôlée, sans trop tirer" explique Rosa.
Cette étude n'est qu'une première étape. Pour des détails quantitatifs sur ce processus (quel est ce seuil et comment la force doit être mesurée pour maximiser l'efficacité de cette méthode de séquençage) des examens plus approfondis seront nécessaires tant au niveau théorique (le modèle développé par Rosa, Di Ventra et Micheletti est mésoscopique, non atomistique) et au niveau expérimental.
Plus en détail…
Le nanoporeséquençage est une technique innovante, une alternative aux méthodes plus traditionnelles telles que l'ACP. Cette méthode consiste à séparer les deux brins de nucléobase qui composent la double hélice de l'ADN et à les analyser un à un. Chaque brin est conduit à travers un nanopore pendant que les variations électriques de la translocation sont enregistrées. C'est une méthode électrochimique :les altérations du champ électrique renseignent sur la composition chimique de la molécule entraînée à travers le pore et la composition est ainsi reconstituée. Jusqu'à présent, cette méthode a donné de bons résultats avec de courts fragments d'ADN, alors que des difficultés ont été rencontrées pendant des périodes plus longues, à cause des nœuds. C'est pourquoi des études telles que Rosa, Di Ventra et Micheletti's sont une étape importante pour augmenter son efficacité.