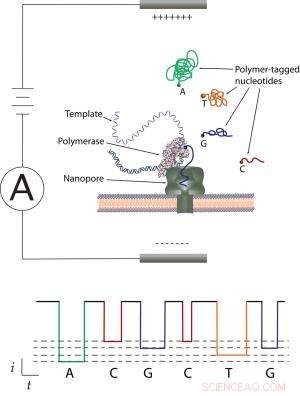
Schéma d'une membrane artificielle, à travers laquelle une tension force un fluide ionisé à travers le nanopore. Les nucléotides sur un brin d'ADN sont d'abord marqués avec des polymères de différentes tailles, puis le brin est passé près de l'ouverture des nanopores, où une polymérase clive les polymères et les fait passer un par un à travers le nanopore. Au passage, le pore produit une signature de blocage de courant ionique unique en raison de la structure chimique distincte de l'étiquette, déterminant ainsi la séquence d'ADN. Crédit :NIST
(Phys.org)—rapide, un séquençage génétique précis pourrait bientôt être à la portée de chaque médecin si les recherches récentes du National Institute of Standards and Technology (NIST) et de la Columbia University School of Engineering and Applied Science peuvent être commercialisées efficacement. L'équipe a démontré un coût potentiellement faible, moyen fiable d'obtenir les séquences d'ADN complètes de tout individu à l'aide d'une sorte de lecteur de bande magnétique moléculaire, permettant potentiellement une détection facile des marqueurs de la maladie dans l'ADN d'un patient.
Alors que le séquençage du génome d'une espèce animale pour la première fois est si courant qu'il ne fait presque plus parler d'eux, il est moins connu que le séquençage de l'ADN d'un seul individu est une affaire coûteuse, coûtant plusieurs milliers de dollars en utilisant la technologie d'aujourd'hui. Le génome d'un individu porte des marqueurs qui peuvent avertir à l'avance du risque de maladie, mais tu as besoin d'un jeûne, moyen fiable et économique de séquencer les gènes de chaque patient pour en tirer pleinement parti. Il est tout aussi important de séquencer continuellement l'ADN d'un individu tout au long de sa vie, car le code génétique peut être modifié par de nombreux facteurs.
La nouvelle méthode détermine les séquences d'ADN en attachant des « étiquettes » moléculaires distinctes à chacun des quatre éléments constitutifs chimiques, ou "bases, " qui comprennent l'information génétique dans un brin d'ADN - abrégé en A, G, C et T. Chacune de ces étiquettes polymères peut ensuite être coupée du brin et passée, un par un, à travers un trou de taille nanométrique dans une membrane. Un flux constant de fluide et d'ions traverse ce "nanopore, " qui est assez grand pour ne contenir qu'une seule étiquette à la fois. Comme les étiquettes en polymère sont de tailles différentes, le changement de courant électrique causé par un écoulement de fluide modifié montre laquelle des quatre bases se trouve à chaque point du brin d'ADN.
Les nanopores et leur interaction avec les molécules polymères sont depuis longtemps un objectif de recherche du scientifique du NIST, John Kasianowicz. Son groupe a collaboré avec une équipe dirigée par Jingyue Ju, directeur du Columbia's Center for Genome Technology and Biomolecular Engineering, qui a eu l'idée de marquer des blocs de construction d'ADN pour le séquençage d'une seule molécule par détection de nanopores. La capacité à discriminer entre les étiquettes polymères a été démontrée par Kasianowicz, son collègue du NIST Joseph Robertson, et d'autres. L'Université Columbia a déposé des brevets pour la commercialisation de la technologie.
Kasianowicz estime que la technique pourrait identifier un élément constitutif de l'ADN avec une précision extrêmement élevée à un taux d'erreur inférieur à un sur 500 millions, et l'équipement nécessaire serait à la portée de n'importe quel fournisseur de soins de santé. "Le cœur du séquenceur serait un amplificateur opérationnel qui coûterait bien moins de 1 $, 000 pour un achat unique, " il dit, "et le coût des matériaux et des logiciels devrait être insignifiant."
Kasianowicz ajoute qu'une entreprise privée pourrait créer un large éventail de nanopores capables d'analyser le génome d'un seul individu découpé en plusieurs brins courts d'ADN, chacun pouvant être séquencé rapidement. Une telle matrice pourrait potentiellement fournir le séquençage à faible coût nécessaire pour une utilisation médicale de routine.