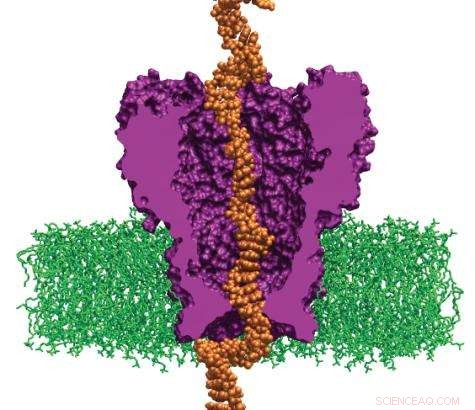
Les scientifiques simulent l'interaction de l'ADN avec une protéine modifiée. Le système peut ralentir suffisamment les brins d'ADN traversant les pores pour lire le génome individuel d'un patient. (Image avec l'aimable autorisation d'Aleksei Aksimentiev)
(PhysOrg.com) -- Le projet du génome humain a ouvert la voie à la génomique, l'étude du génome d'un organisme. La génomique personnalisée peut établir la relation entre les variations de séquence d'ADN chez les individus et leurs conditions de santé et leurs réponses aux médicaments et aux traitements. Faire du séquençage du génome une procédure de routine, cependant, le temps doit être réduit à moins d'une journée et le coût à moins de 1 $, 000 -- un exploit impossible avec les connaissances et les technologies actuelles.
En 2008, une équipe de recherche dirigée par Aleksei Aksimentiev, professeur assistant au département de physique de l'Université de l'Illinois-Urbana-Champaign, a lancé un projet de création de machines pour le séquençage du génome personnel qui seront plus accessibles aux hôpitaux. En utilisant le Jaguar du Laboratoire national d'Oak Ridge, l'un des supercalculateurs les plus rapides au monde, Aksimentiev et son équipe développent une approche nanopore, qui promet une réduction drastique du temps et des coûts pour le séquençage de l'ADN. Leurs recherches révèlent la forme de l'ADN se déplaçant à travers un seul nanopore - un pore de protéine d'un milliardième de mètre de large qui traverse une membrane. Lorsque l'ADN traverse le pore, la séquence de nucléotides (éléments constitutifs de l'ADN) est lue par un détecteur.
« Le principal obstacle au séquençage utilisant les anciennes générations de nanopores biologiques et synthétiques était l'incapacité d'identifier la séquence d'ADN à la résolution d'un seul nucléotide, " a déclaré Aksimentiev. " Les nucléotides sont passés trop rapidement à travers le nanopore pour que les scientifiques puissent séquencer l'ADN. "
Le groupe d'Aksimentiev utilise le nanopore MspA, une protéine modifiée. Sa séquence doit être modifiée pour se lier plus fortement au brin d'ADN en mouvement. MspA est une plateforme idéale pour le séquençage de l'ADN car les scientifiques peuvent désormais mesurer les barrages dans le pore, ce qui pourrait ralentir le voyage de l'ADN à travers la protéine. La modification de la protéine MspA pour optimiser les barrages est à la fois longue et coûteuse en laboratoire mais simple sur un ordinateur. Par exemple, modifier la protéine de quelque manière que ce soit, les scientifiques doivent déterminer si la mutation particulière qu'ils introduisent est stable et si l'idée est raisonnable. Par conséquent, les scientifiques simulent d'abord MspA pour décider d'une mutation à induire et tester des idées à haut risque avant de les mettre en œuvre dans une expérience.
L'équipe de recherche utilise le code NAMD, qui calcule les états d'énergie minimum des atomes dans un grand système biomoléculaire et est un indicateur des formes que les molécules seraient les plus à l'aise de supposer. L'équipe construit d'abord un modèle de la protéine MspA immergée dans une solution de bicouche lipidique et d'électrolyte. Un brin d'ADN d'une séquence nucléotidique souhaitée est ensuite enfilé à travers le nanopore MspA. Ensuite, les scientifiques simulent l'effet d'un champ électrique entraînant des ions et de l'ADN à travers le nanopore MspA.
La simulation utilise la dynamique moléculaire, ou des calculs du mouvement de chaque atome dans un système moléculaire suivant les lois physiques de la nature, pour imiter le système expérimental. Les résultats des simulations peuvent être directement comparés à ceux des expériences car les deux approches mesurent le courant ionique, selon Aksimentiev. En connaissant les positions de chaque atome et ion d'ADN, les scientifiques obtiennent un avantage :ils peuvent optimiser le séquençage des nanopores en utilisant une conception rationnelle pour produire un pore qui épouse plus étroitement l'ADN, ralentir le voyage de la molécule à travers le pore à une vitesse permettant la résolution d'un seul nucléotide.
Le travail de séquençage est financé par le National Human Genome Research Institute des National Institutes of Health. Le développement de la méthode du projet est financé en partie par la National Science Foundation. Les collaborateurs du projet comprennent deux groupes expérimentaux :l'un dirigé par Jens Gundlach à l'Université de Washington-Seattle et l'autre par Michael Niederweis à l'Université d'Alabama-Birmingham.
La recherche a reçu 10 millions d'heures de processeur sur Jaguar grâce à l'impact informatique innovant et nouveau sur la théorie et l'expérience, ou INCITE, programme, qui attribue des allocations considérables sur certains des supercalculateurs les plus puissants du monde à des projets abordant les grands défis de la science et de l'ingénierie. Avec l'allocation INCITE, les scientifiques ont pu reproduire les barrages dans le nanopore MspA pour le type de nucléotides d'ADN qui y sont confinés, ralentir le mouvement de la séquence à travers le nanopore.
"Nous avons mené une étude pilote sur plusieurs variantes du nanopore MspA et observé une réduction considérable de la vitesse des brins d'ADN, " a déclaré Aksimentiev. " Ces résultats très préliminaires suggèrent que l'obtention d'une réduction de 100 fois de la vitesse de l'ADN, qui devrait être suffisant pour lire la séquence d'ADN avec une résolution d'un seul nucléotide, est à portée de main. Les études futures seront orientées vers cet objectif. »
L'équipe espère atteindre l'objectif de ce projet d'ici 2013 et prévoit de poursuivre un certain nombre de projets dérivés passionnants, dit Aksimentiev. La capacité de rendre le séquençage du génome abordable permettra à des programmes tels que le Cancer Genome Project, qui caractérise les mutations de l'ADN dans les cellules cancéreuses de divers tissus à tous les stades du développement du cancer.