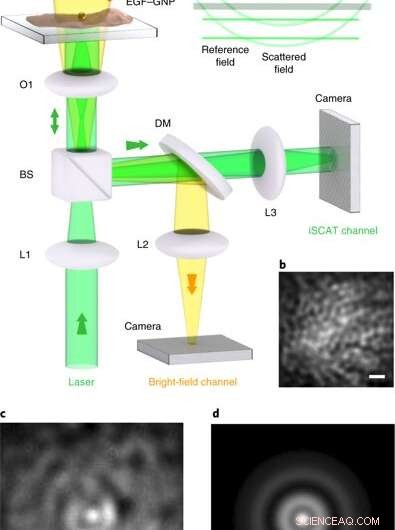
Microscopie iSCAT sur cellules vivantes. une, Disposition expérimentale du microscope iSCAT pour l'imagerie des cellules vivantes. Les cellules sont étalées dans une coupelle à fond de verre sous milieu Leibowitz. (a) la micropipette délivre les sondes EGF-GNP directement sur la culture cellulaire, où ils ciblent spécifiquement la protéine EGFR dans la membrane cellulaire. Le canal d'éclairage à fond clair d'en haut aide à inspecter la culture mais n'est pas requis pour l'imagerie iSCAT. L1–L3, lentilles; O1, objectif ×100 ; BS, diviseur de faisceau 90:10 ; DM, Miroir dichroïque passe-court de 590 nm. L'imagerie iSCAT a été réalisée avec des intensités d'éclairage de 1–8 kW cm-2, qui sont connus pour être viables pour HeLa à la longueur d'onde d'intérêt. Encart, fronts d'onde des champs contribuant au signal iSCAT. (b), Une section de la membrane de la cellule HeLa avant marquage, visualisé par réflexion iSCAT. (c), Image iSCAT de la membrane cellulaire comprenant une sonde EGF-GNP liée. (ré), Le PSF extrait de c. Les barres d'échelle en b–d sont de 1 m. Crédit: Photonique de la nature , doi:10.1038/s41566-019-0414-6
Les fonctions cellulaires sont dictées par le mouvement complexe des protéines dans les membranes qui s'étendent sur une échelle de nanomètres à micromètres, dans un laps de temps de quelques microsecondes à quelques minutes. Cependant, ce riche paramètre de l'espace est inaccessible en microscopie à fluorescence, bien qu'il soit à la portée du suivi des particules par diffusion interférométrique (iSCAT). La nouvelle technique iSCAT est, cependant, très sensible aux protéines simples et non marquées, provoquant ainsi une coloration de fond non spécifique comme un défi substantiel lors de l'imagerie cellulaire.
Dans une étude récente, Richard W. Taylor et ses collègues des départements interdisciplinaires de physique et de biologie en Allemagne ont développé une nouvelle approche de traitement d'images pour surmonter cette difficulté. Ils ont utilisé la méthode pour suivre le récepteur du facteur de croissance épidermique transmembranaire (EGFR) avec une précision à l'échelle nanométrique en trois dimensions (3-D). La technique a permis l'imagerie de quelques microsecondes à quelques minutes. Les scientifiques ont fourni des exemples de mouvement et de confinement à l'échelle nanométrique en utilisant la méthode pour imager des processus omniprésents tels que la diffusion dans les membranes plasmiques, transport dans les filopodes et mouvement de rotation pendant l'endocytose. Les résultats sont maintenant publiés dans Photonique de la nature .
Alors que les progrès constants de la microscopie à fluorescence ont permis aux scientifiques de surveiller les événements cellulaires à l'échelle nanométrique, il reste encore beaucoup à faire avec des systèmes d'imagerie avancés. Les défis de la microscopie à fluorescence sont dus au taux d'émission fini d'une source fluorescente (molécule de colorant ou point quantique semi-conducteur), où trop peu d'émissions de photons pendant une très courte période de temps ont empêché une imagerie efficace ou prolongée. La difficulté centrale de la microscopie par diffusion est relative à la sonde nanoscopique, qui rivalise avec le bruit de fond et un faible rapport signal sur bruit (SNR); limiter le potentiel de l'imagerie à quelques nanomètres seulement dans les expériences de suivi à grande vitesse.

Vidéo brute d'une nanoparticule de facteur de croissance épidermique-or (EGFR-GNP) diffusant sur une membrane cellulaire HeLa. Crédit: Photonique de la nature , doi:10.1038/s41566-019-0414-6
Dans le travail present, Taylor et al. utilisé la microscopie à diffusion interférométrique (iSCAT) pour suivre les protéines dans les membranes cellulaires vivantes. La méthode pourrait visualiser les interactions sonde-cellule pour comprendre la dynamique entre la diffusion et la topologie locale. Lors des expérimentations, les scientifiques ont utilisé des nanoparticules d'or (GNP) pour marquer les récepteurs du facteur de croissance épidermique (EGFR) dans les cellules HeLa. Les EGFR sont des protéines transmembranaires de type I capables de détecter et de répondre aux signaux extracellulaires, dont la signalisation aberrante est liée à une variété de maladies. Taylor et al. a montré la protéine marquée GNP comme un « nano-rover » qui a cartographié la nano-topologie des caractéristiques cellulaires telles que les terrains membranaires, structures filopodes et clathrine. Ils ont fourni des exemples de mouvement de sous-diffusion et de confinement nanoscopique d'une protéine en 3D à haute résolution temporelle et à des points de temps longs.
Dans les expériences, Taylor et al. a introduit les sondes de nanoparticules d'or de facteur de croissance épidermique (EGF-GNP) dans la chambre d'échantillon du microscope à l'aide d'une micropipette pour marquer les EGFR (récepteurs de facteur de croissance épidermique) sur les cellules HeLa et a vérifié que les sondes stimulaient les EGFR. Des études antérieures avaient déjà indiqué que la taille de la sonde pouvait influencer les taux de diffusion des lipides dans les membranes synthétiques, bien qu'ils n'affectent pas le mode de diffusion. En outre, dans les cellules vivantes, l'encombrement moléculaire était négligeable pour les particules égales ou inférieures à 50 nm.
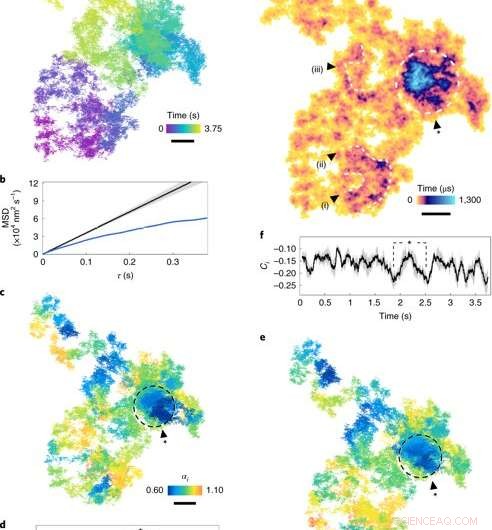
Diffusion sur la membrane plasmique. (une), Une trajectoire diffusionnelle latérale (temps d'exposition de 17,5 s, voir échelle de couleurs pour la chronologie). (b), MSD (déplacement quadratique moyen) en fonction de τ. La courbe bleue montre le MSD de a. La courbe noire est une diffusion normale simulée (α= 1), avec l'enveloppe grise indiquant l'incertitude. (c), L'exposant de diffusion des fenêtres roulantes (échelle de couleurs) sur la trajectoire. Les régions de sous-diffusion (α<1) sont indiquées par des nuances plus foncées. (ré), i à travers le temps. L'ombrage gris représente une incertitude moyenne de 7 ± 4%, correspondant à un intervalle de confiance à 95% pour une fenêtre de 100µms (1, 000 images) et τ= 250 μs. Les points marqués d'un astérisque correspondent au cercle en c. (e), La direction de pas Ci pour les fenêtres roulantes le long de la trajectoire. (F), La direction du pas Ci tracée dans le temps, avec l'ombrage indiquant l'incertitude. (g), Tracé d'occupation ATOM avec temps de résidence (échelle de couleurs). La taille du bac correspond à l'erreur de localisation. Régions remarquables d'occupation prolongée, marqués comme des boucles et des tourbillons (i)–(iii), sont révélateurs de structures nanoscopiques persistantes. La région fermée représente une zone dense de subdiffusion notable. Barres d'échelle, 100 nm. Crédit: Photonique de la nature , doi:10.1038/s41566-019-0414-6
Taylor et al. vérifié ces deux cas concrets dans le présent travail en comparant des PNB de diamètres variables à 48 nm et 20 nm. Les scientifiques ont ensuite mené des études fluorescentes et biochimiques pour suggérer que les GNP recouverts d'EGF activaient la signalisation EGFR, tout comme les EGF disponibles gratuitement, indiquant que le marqueur n'entrave pas les fonctions biologiques. Pour surmonter le bruit de fond lié à l'imagerie moléculaire, les scientifiques ont mis en place un nouvel algorithme, qui a extrait la fonction complète d'étalement des points iSCAT (iSCAT-PSF) directement de chaque image pour plus de clarté. Étant donné que les techniques existantes sont incapables de visualiser les caractéristiques à haute résolution spatiale et temporelle, de nombreux détails sur l'activité intracellulaire restent un sujet de débat. En réponse, la nouvelle méthode de Taylor et al. a offert une multitude d'hétérogénéités dynamiques en 3-D pour faire la lumière sur le mouvement des protéines intracellulaires.
Les scientifiques ont d'abord étudié quantitativement la sous-diffusion dans la membrane plasmique en considérant un exemple en 2D du trajet de l'EGFR sur la membrane d'une cellule HeLa vivante. Pour ça, ils ont calculé le déplacement quadratique moyen (MSD) pour toute la trajectoire du mouvement. Taylor et al. n'a pas eu besoin de faire d'hypothèses sur la nature de la diffusion ou son paysage géographique lors du calcul. Ils ont mesuré l'apparition de barrières diffractives et de confinements biologiques en observant le degré de corrélation directionnelle entre deux étapes vectorielles sur une période de temps.

Diffusion sur filopode. Crédit: Photonique de la nature , doi:10.1038/s41566-019-0414-6
Les scientifiques ont ensuite évalué la popularité de chaque pixel de trajectoire dans l'espace en introduisant une carte d'occupation temporelle accumulée (ATOM). Dans cette technique, ils ont divisé le plan latéral de la trajectoire en bacs de taille nanométrique et ont compté l'occurrence de la particule dans chaque bac. Les résultats ont indiqué l'agencement des nanostructures en boucles et tourbillons dans une durée de vie minimale de 250 nanosecondes (5000 images) pour représenter potentiellement une étape pré-endocytaire. Au total, les observations simulées ont montré comment la diffusion des protéines était affectée par la sous-structure de la cellule.
La technique de microscopie iSCAT a permis aux scientifiques d'enregistrer les effets sur une très longue période de temps, qu'ils ont utilisé avec des capacités d'imagerie 3D pour suivre les EGFR sur un filopode. Les filopodes sont des protubérances cellulaires en forme de bâtonnets biologiques contenant des faisceaux de filaments d'actine d'un diamètre allant jusqu'à 100 à 300 nm et d'une longueur de 100 µm. Les nanostructures peuvent détecter des stimuli mécaniques pour la chimioattraction ou la répulsion dans le microenvironnement cellulaire tout en fournissant des sites d'attachement cellulaire. La liaison au ligand et l'activation de l'EFGR sur les filopodes se sont produites à de faibles concentrations d'EGF, suivi de son association avec les filaments d'actine et le transport rétrograde de l'EFGR vers le corps cellulaire.
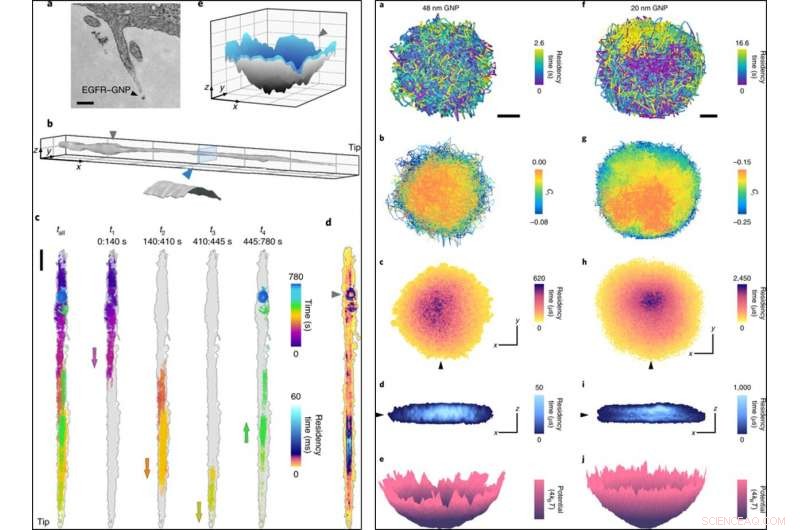
À GAUCHE :(a) Une image MET (microscope électronique à transmission) d'un filopode comprenant un EGFR-GNP. (b), Une surface de filopode reconstruite à partir de 780, 000 points de trajectoire avec une erreur de localisation de σx, y = 2 nm enregistré à 1, 000 fps. Encart, coupe transversale qui représente une surface cylindrique de diamètre 150 nm après avoir pris en compte la taille du PNB. (c), Une trajectoire brute de 13 min (à gauche) divisée en quatre morceaux successifs qui révèlent le trajet vers et depuis la pointe, avec des flèches indiquant la direction du mouvement du filet. (ré), Une parcelle ATOM de c, corrigé de la dérive du filopode. (e), Une interpolation de surface des dernières années 80. Le confinement en forme d'anneau dans la phase finale (marqué d'un triangle) est une fosse 3D. Les barres d'échelle sont 200 nm (a), 1 μm (x, y) et 200 nm (z) (b), 1, 000 nm (c) et 100 nm (x, y) et 50 nm (z) (e). DROITE :(a), Trajectoire latérale d'une sonde GNP 48 nm. Barre d'échelle, 100 nm. Un échantillonnage temporel plus faible de ce confinement aurait sous-estimé l'étendue de la délimitation. (b), Ci de la trajectoire (en utilisant un décalage de cinq images), qui montre une diffusion partiellement entravée avec une propension à une diffusion plus libre au centre. (c), Une parcelle ATOM de a. (ré), Une coupe à travers le tracé 3D-ATOM le long de la ligne du triangle noir en c montre que l'occupation favorise une région semblable à un disque la plus interne. Les axes désignent 100 nm à la fois en c et en d. (e), Conversion de l'occupation temporelle 2D de c en une distribution d'énergie potentielle effective. (f–j), Équivalent à a–e, mais pour une sonde GNP de 20 nm. Crédit: Photonique de la nature , doi:10.1038/s41566-019-0414-6
Les scientifiques ont ainsi obtenu un aperçu des détails nanoscopiques de la diffusion le long du filopode et ont enregistré les données pendant 13 minutes. Ils ont analysé la trajectoire 3D pour créer la topographie du filopode en utilisant des nanoparticules d'or comme « nano rover » et ont cartographié la topologie de surface des structures cellulaires pour un examen plus approfondi. Ils ont tracé la trajectoire ATOM (carte d'occupation temporelle accumulée) et ont constaté que la représentation 3-D était cohérente avec l'étape biologique de l'invagination membranaire pré-endocytaire.
Des techniques de microscopie à grande vitesse telles que iSCAT sont nécessaires pour obtenir des informations temporelles à haute résolution et éviter les effets de flou lors de l'imagerie basée sur la localisation des nanoparticules. Les scientifiques ont démontré cette caractéristique en enregistrant la diffusion confinée à 30, 000 fps (images par seconde) avec des PNB de 48 nm et 20 nm. Ils ont suivi les expériences avec un suivi 3D ultra-rapide des protéines à 66, 000 fps en utilisant un temps d'exposition court de 10 µs sur une durée de 3,5 secondes. L'imagerie par microscopie iSCAT rapide a fourni des preuves supplémentaires pour révéler les caractéristiques complexes des événements endocytaires par rapport à l'endocytose médiée par la clathrine dans les cellules HeLa lorsqu'elles sont simulées par de faibles concentrations d'EGF.
De cette façon, Taylor et al. ont noté que la nouvelle technique pouvait enregistrer fidèlement les informations nano-topographiques. Les résultats correspondent aux observations enregistrées par microscopie électronique à transmission (MET) sans différences significatives sur la réduction de la taille de la sonde de 48 nm à 20 nm, tout en offrant de nouvelles perspectives. Les nouvelles informations comprenaient des détails sur la subdiffusion, confinement nanoscopique, Contours 3-D des structures de filopodes et de clathrine à l'échelle nanométrique. Les scientifiques ont l'intention de combiner iSCAT avec la microscopie à fluorescence à super-résolution in situ pour comprendre les trajectoires des protéines, virus et autres entités biologiques nanoscopiques. Taylor et al. visent à faire progresser les méthodes d'analyse d'images pour suivre les PNB inférieurs à 20 nm à l'avenir et pensent que la nouvelle technologie et l'optimisation supplémentaire leur permettront de comprendre spécifiquement le cycle de vie des virus sans utiliser d'étiquette externe pour le suivi.
© 2019 Réseau Science X