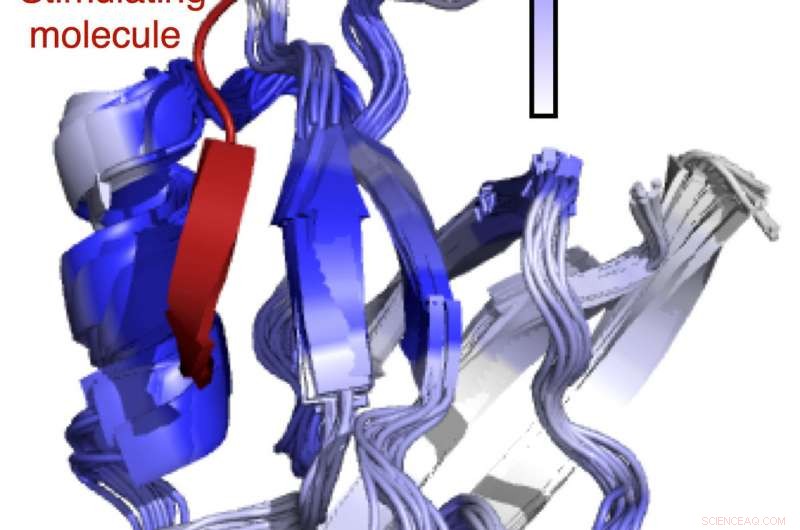
Analyse basée sur l'apprentissage automatique des voies de signalisation trouvées dans les acides aminés présents dans les protéines humaines. Crédit :Navli Duro/Université de Floride du Sud
Les algorithmes d'apprentissage automatique excellent à trouver des modèles complexes dans les mégadonnées, les chercheurs les utilisent donc souvent pour faire des prédictions. Les chercheurs poussent cette technologie émergente au-delà de la recherche de corrélations pour aider à découvrir des relations de cause à effet cachées et à conduire des découvertes scientifiques.
À l'Université de Floride du Sud, les chercheurs intègrent des techniques d'apprentissage automatique dans leurs travaux d'étude des protéines. Comme ils rapportent dans Le Journal de Physique Chimique , l'un de leurs principaux défis a été le manque de méthodes pour identifier les relations de cause à effet dans les données obtenues à partir de simulations de dynamique moléculaire.
"Les protéines peuvent être considérées comme des machines nanoscopiques qui effectuent un ensemble de tâches. Mais le moment et l'endroit où les protéines effectuent leurs tâches spécifiques sont contrôlés par les cellules via divers stimuli, comme les petites molécules, " dit Sameer Varma, professeur agrégé de biophysique à l'USF. "Ces stimuli interagissent avec les protéines pour les activer et les désactiver, ' et peut même modifier leurs vitesses et leurs forces."
Dans la plupart des protéines, les stimuli biologiques interagissent avec un site sur la protéine qui est relativement éloigné de la partie qui effectue sa tâche correspondante, nécessitant une voie de signalisation. "Cette manière télécommandée de commuter les protéines est connue sous le nom de" signalisation allostérique ". De nombreuses protéines d'importance pharmaceutique ont maintenant été identifiées où la dynamique ou le « soulèvement et ondulation » de leurs atomes constitutifs sont connus pour être essentiels à la signalisation allostérique, " dit Varma. " Les détails, cependant, restent sommaires."
Varma et ses collègues pensent que les approches d'apprentissage automatique peuvent faire la différence. « Le développement et l'utilisation de techniques d'apprentissage automatique nous permettront de trouver des relations de cause à effet dans les données sur la dynamique des protéines et de commencer à répondre à certaines des questions très fondamentales de l'allostère des protéines, " at-il dit. " L'une de nos principales découvertes était que le signal initié au site de stimulation de la protéine semblait s'affaiblir à mesure qu'il s'éloignait du site de stimulation. C'est venu comme une surprise, car aucune dépendance à la distance n'a été observée pour le couplage des mouvements thermiques entre les sites protéiques."
Les travaux du groupe montrent comment les approches d'apprentissage automatique peuvent être utilisées pour identifier les relations de cause à effet au sein des données. Au delà de ça, « ces techniques nous permettent de combler des lacunes critiques dans l'allostère protéique, " dit Varma. " En fin de compte, lorsque nos méthodes sont appliquées aux nombreuses protéines d'intérêt pharmaceutique, nous nous attendons à ce que les détails mécanistiques révèlent de nouvelles stratégies d'intervention indispensables pour restaurer les activités des protéines dans les états pathologiques. Les connaissances biophysiques générales que nous acquérons devraient également aider à inspirer de nouvelles solutions biomimétiques pour de nombreux problèmes de nano-ingénierie, tels que la conception de nanocapteurs pour l'administration ciblée de médicaments."
Les chercheurs envisagent de nouveaux travaux passionnants qui découleront de leurs récentes découvertes. "Jusque là, nous nous sommes concentrés sur les données d'équilibre, mais le processus de signalisation a une composante critique de non-équilibre que nous n'avons pas encore explorée, " a déclaré Varma. Le groupe prévoit également d'explorer plus en détail le rôle des eaux environnantes dans la signalisation, ainsi que d'appliquer leurs techniques d'apprentissage automatique à un large éventail de familles de protéines pour déterminer dans quelle mesure leurs nouvelles découvertes biophysiques sont généralisables.