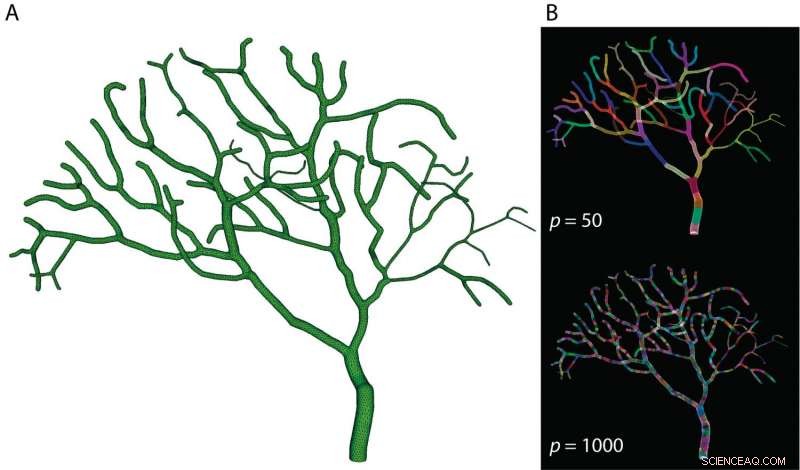
En décomposant le modèle cellulaire partiel de Purkinje (A) en 50 sections (B-top) ou 1000 sections (B-bottom) et en exécutant les calculs de chaque section en parallèle sur un superordinateur, Les chercheurs de l'OIST ont considérablement réduit le temps de simulation du modèle. Crédit :OIST
Contrairement aux neuroscientifiques expérimentaux qui s'occupent des neurones de la vie réelle, les neuroscientifiques computationnels utilisent des simulations de modèles pour étudier le fonctionnement du cerveau. Alors que de nombreux neuroscientifiques computationnels utilisent des modèles mathématiques simplifiés de neurones, des chercheurs de l'unité de neurosciences computationnelles de l'Okinawa Institute of Science and Technology Graduate University (OIST) développent un logiciel qui modélise les neurones dans le détail des interactions moléculaires dans le but d'obtenir de nouvelles informations sur la fonction neuronale. Les applications du logiciel étaient jusqu'à présent limitées en raison de la puissance de calcul intense requise pour des modèles neuronaux aussi détaillés, mais récemment le Dr Weiliang Chen, Dr Iain Hepburn, et le professeur Erik De Schutter ont publié deux articles connexes dans lesquels ils décrivent la précision et l'évolutivité de leur nouveau logiciel de calcul à grande vitesse, "PAS PARALLÈLES". Les résultats combinés suggèrent que Parallel STEPS pourrait être utilisé pour révéler de nouvelles informations sur la façon dont les neurones individuels fonctionnent et communiquent entre eux.
Le premier papier, Publié dans Le Journal de Physique Chimique en août 2016, se concentre sur la garantie que la précision de Parallel STEPS est comparable aux méthodes conventionnelles. Dans les approches conventionnelles, les calculs associés aux réactions chimiques neuronales et à la diffusion des molécules sont tous calculés séquentiellement sur une unité de traitement informatique ou « noyau ». Cependant, Le Dr Iain Hepburn et ses collègues ont introduit une nouvelle approche pour effectuer des calculs de réaction et de diffusion en parallèle qui peuvent ensuite être répartis sur plusieurs cœurs d'ordinateur, tout en maintenant la précision de la simulation à un degré élevé. La clé était de développer un algorithme original séparé en deux parties - une qui calculait les événements de réaction chimique et l'autre les événements de diffusion.
« Nous avons testé une gamme de simulations de modèles allant des modèles de diffusion simples aux modèles biologiques réalistes et avons constaté que nous pouvions obtenir des performances améliorées en utilisant une approche parallèle avec une perte de précision minimale. Cela a démontré la pertinence potentielle de la méthode à plus grande échelle, " dit le Dr Hepburn.
Dans un article connexe publié dans Frontières en neuroinformatique ce février, Le Dr Weiliang Chen a présenté les détails de la mise en œuvre de Parallel STEPS et a étudié ses performances et ses applications potentielles. En décomposant un modèle partiel d'une cellule de Purkinje - l'un des plus gros neurones du cerveau - en 50 à 1000 sections et en simulant des événements de réaction et de diffusion pour chaque section en parallèle sur le supercalculateur Sango de l'OIST, Le Dr Chen et ses collègues ont constaté une augmentation spectaculaire des vitesses de calcul. Ils ont testé cette approche à la fois sur des modèles simples et des modèles plus compliqués d'explosions de calcium dans les cellules de Purkinje et ont démontré que la simulation parallèle pouvait accélérer les calculs plusieurs centaines de fois par rapport aux méthodes conventionnelles.
"Ensemble, nos résultats montrent que la mise en œuvre de STEPS parallèle permet d'améliorer considérablement les performances, et une bonne évolutivité, " explique le Dr Chen. " Des modèles similaires qui nécessitaient auparavant des mois de simulation peuvent maintenant être complétés en quelques heures ou minutes, ce qui signifie que nous pouvons développer et simuler des modèles plus complexes, et apprenez-en plus sur le cerveau en moins de temps."
Dr Hepburn et Dr Chen de l'unité de neurosciences computationnelles de l'OIST, dirigé par le professeur Erik De Schutter, collaborent activement avec le Human Brain Project, une initiative mondiale basée à l'École Polytechnique Fédérale de Lausanne (EPFL) en Suisse, développer une version plus robuste de Parallel STEPS qui intègre la simulation du champ électrique des membranes cellulaires.
Jusqu'à présent, STEPS n'est capable de modéliser de manière réaliste que des parties de neurones, mais avec le soutien de Parallel STEPS, l'unité Computational Neuroscience espère développer un modèle grandeur nature d'un neurone entier et par la suite des interactions entre les neurones d'un réseau. En collaborant avec l'équipe de l'EPFL et en utilisant le supercalculateur IBM 'Blue Gene/Q' qui s'y trouve, ils visent à atteindre ces objectifs dans un avenir proche.
"Grâce aux superordinateurs modernes, nous pouvons étudier les événements moléculaires au sein des neurones de manière beaucoup plus transparente qu'auparavant, ", déclare le professeur De Schutter. "Nos recherches ouvrent des voies intéressantes en neurosciences computationnelles qui relient pour la première fois la biochimie à l'électrophysiologie."