Une nouvelle technologie de séquençage de protéines avec une sensibilité et un débit améliorés révolutionnera la protéomique et le diagnostic clinique.
Dans une étude publiée dans Nature Methods , une équipe de recherche dirigée par le professeur Wu Haichen de l'Institut de chimie de l'Académie chinoise des sciences (CAS) et le professeur Liu Lei de l'Institut de physique des hautes énergies du CAS, ainsi que leurs collaborateurs, ont développé une nouvelle méthode pour le séquençage de peptides basé sur la détection de nanopores assistée par interaction hôte-invité.
L’histoire du séquençage des protéines pourrait remonter à la détermination de la séquence complète des acides aminés de l’insuline par Sanger dans les années 1950. Cependant, jusqu'à présent, il n'existe que deux méthodes principales pour le séquençage des protéines, à savoir la spectrométrie de masse et la dégradation d'Edman.
Au cours des dernières décennies, la détection des nanopores est apparue comme la dernière technique « perturbatrice » à molécule unique et a remporté un grand succès dans la nouvelle génération de développement du séquençage de l’ADN. Cela a incité les scientifiques à transplanter la technologie vers le séquençage de protéines à molécule unique. Cependant, le séquençage nanopore des protéines fait face à d'énormes défis, tels que la réalisation d'un transport unidirectionnel de chaînes peptidiques chargées de manière hétérogène à travers un nanopore et l'identification électrique de 20 acides aminés individuels ou de leurs combinaisons.
Dans cette étude, les chercheurs ont proposé une stratégie de séquençage alternative basée sur une technique améliorée de détection des nanopores assistée par interaction hôte-invité.
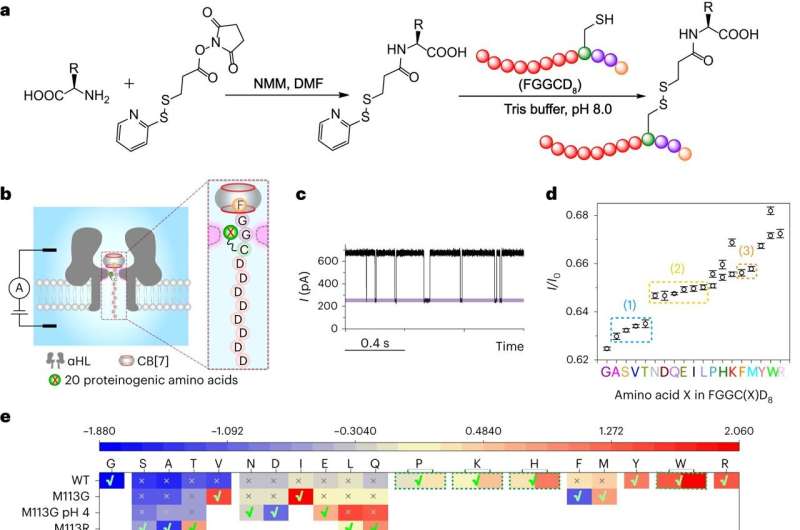
Les peptides modèles ont d’abord été digérés avec des carboxypeptidases pour obtenir un mélange d’acides aminés. L'étape clé suivante consistait à coupler les acides aminés libérés à une sonde peptidique FGGCD8⊂CB[7] via un lieur covalent, puis à soumettre le complexe à des expériences de translocation via l'α-hémolysine de type sauvage ou ses mutants.
Enfin, le blocage actuel de chaque peptide FGGC(X)D8⊂CB[7] a été utilisé pour identifier l'acide aminé X et leur abondance relative a été utilisée pour déterminer l'ordre du clivage enzymatique, c'est-à-dire la séquence du peptide. /P>
Cette étude sert de démonstration de validation de principe pour une nouvelle méthode capable de déterminer avec précision la séquence d’acides aminés d’un peptide. Bien que des limites notables persistent, cela marque un progrès significatif et dévoile une voie prometteuse pour l'avenir du séquençage des protéines.
Plus d'informations : Yun Zhang et al, Séquençage peptidique basé sur la détection de nanopores assistée par interaction hôte-invité, Nature Methods (2023). DOI :10.1038/s41592-023-02095-4
Informations sur le journal : Méthodes naturelles
Fourni par l'Académie chinoise des sciences