Si E. coli est l'un des organismes les plus étudiés, la fonction de 30 % des protéines qui composent E. coli n'a pas encore été clairement révélée. Pour cela, une intelligence artificielle a été utilisée pour découvrir 464 types d'enzymes à partir de protéines inconnues, et les chercheurs ont ensuite vérifié les prédictions de trois types de protéines qui ont été identifiées avec succès grâce à un test enzymatique in vitro.
Une équipe de recherche conjointe, comprenant Gi Bae Kim, Ji Yeon Kim, le Dr Jong An Lee et le professeur distingué Sang Yup Lee du Département de génie chimique et biomoléculaire du KAIST, ainsi que le Dr Charles J. Norsigian et le professeur Bernhard O. Palsson du le département de bio-ingénierie de l'UCSD a développé DeepECtransformer, une intelligence artificielle capable de prédire les fonctions enzymatiques à partir de la séquence protéique. De plus, l'équipe a établi un système de prédiction en utilisant l'IA pour identifier rapidement et précisément la fonction de l'enzyme.
Le travail de l’équipe est décrit dans l’article intitulé « Annotation fonctionnelle des gènes codant pour des enzymes utilisant l’apprentissage profond avec des couches de transformateurs ». L'article a été publié le 14 novembre dans Nature Communications. .
Les enzymes sont des protéines qui catalysent les réactions biologiques, et l'identification de la fonction de chaque enzyme est essentielle pour comprendre les diverses réactions chimiques qui existent dans les organismes vivants et les caractéristiques métaboliques de ces organismes.
Le numéro de la Commission des enzymes (EC) est un système de classification des fonctions enzymatiques conçu par l'Union internationale de biochimie et de biologie moléculaire. Afin de comprendre les caractéristiques métaboliques de divers organismes, il est nécessaire de développer une technologie capable d'analyser rapidement les enzymes et les numéros EC. des enzymes présentes dans le génome.
Diverses méthodologies basées sur l'apprentissage profond ont été développées pour analyser les caractéristiques des séquences biologiques, y compris la prédiction de la fonction des protéines, mais la plupart d'entre elles rencontrent le problème d'une boîte noire, dans laquelle le processus d'inférence de l'IA ne peut pas être interprété.
Divers systèmes de prédiction utilisant l'IA pour prédire la fonction enzymatique ont également été rapportés, mais ils ne résolvent pas ce problème de boîte noire ou ne peuvent pas interpréter le processus de raisonnement à un niveau plus fin (par exemple, le niveau de résidus d'acides aminés dans la séquence enzymatique). ).
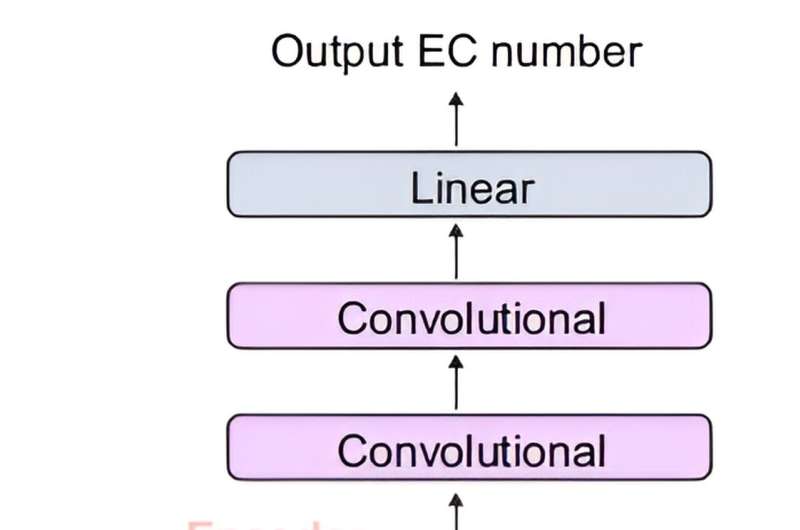
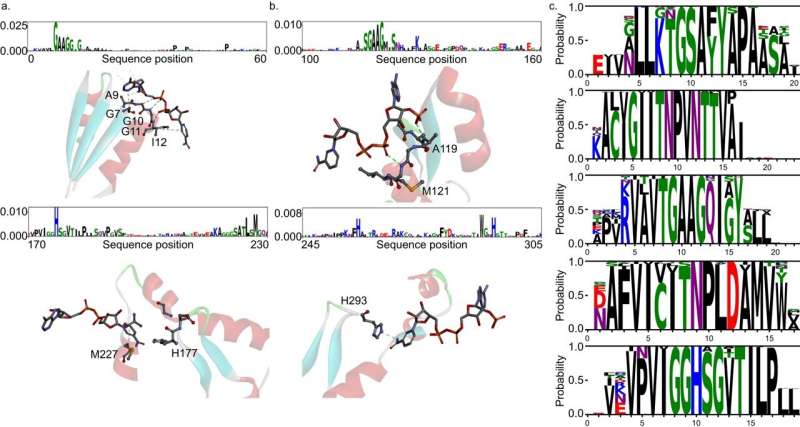
L'équipe commune a développé DeepECtransformer, une IA qui utilise l'apprentissage profond et un module d'analyse d'homologie protéique pour prédire la fonction enzymatique d'une séquence protéique donnée.
Pour mieux comprendre les caractéristiques des séquences protéiques, l'architecture du transformateur, couramment utilisée dans le traitement du langage naturel, a également été utilisée pour extraire des caractéristiques importantes sur les fonctions enzymatiques dans le contexte de l'ensemble de la séquence protéique, ce qui a permis à l'équipe de prédire avec précision la CE. numéro de l’enzyme. Le DeepECtransformer développé peut prédire un total de 5 360 nombres EC.
L'équipe commune a ensuite analysé l'architecture du transformateur pour comprendre le processus d'inférence de DeepECtransformer et a découvert que dans le processus d'inférence, l'IA utilise des informations sur les sites actifs catalytiques et/ou les sites de liaison des cofacteurs qui sont importants pour le fonctionnement de l'enzyme. En analysant la boîte noire de DeepECtransformer, il a été confirmé que l'IA était capable d'identifier seule les caractéristiques importantes pour le fonctionnement de l'enzyme au cours du processus d'apprentissage.
"En utilisant le système de prédiction que nous avons développé, nous avons pu prédire les fonctions d'enzymes qui n'avaient pas encore été identifiées et les vérifier expérimentalement", a déclaré Gi Bae Kim, le premier auteur de l'article.
"En utilisant DeepECtransformer pour identifier des enzymes jusqu'alors inconnues dans les organismes vivants, nous serons en mesure d'analyser plus précisément diverses facettes impliquées dans les processus métaboliques des organismes, telles que les enzymes nécessaires à la biosynthèse de divers composés utiles ou les enzymes nécessaires à la biodégradation des plastiques", il a ajouté.
"DeepECtransformer, qui prédit rapidement et précisément les fonctions des enzymes, est une technologie clé en génomique fonctionnelle, nous permettant d'analyser la fonction d'enzymes entières au niveau des systèmes", a déclaré le professeur Sang Yup Lee.
Il a ajouté :"Nous pourrons l'utiliser pour développer des usines microbiennes respectueuses de l'environnement basées sur des modèles métaboliques complets à l'échelle du génome, minimisant potentiellement les informations manquantes sur le métabolisme."
Plus d'informations : Gi Bae Kim et al, Annotation fonctionnelle de gènes codant pour des enzymes à l'aide de l'apprentissage profond avec des couches de transformateur, Nature Communications (2023). DOI :10.1038/s41467-023-43216-z
Informations sur le journal : Communications naturelles
Fourni par l'Institut supérieur coréen des sciences et technologies (KAIST)