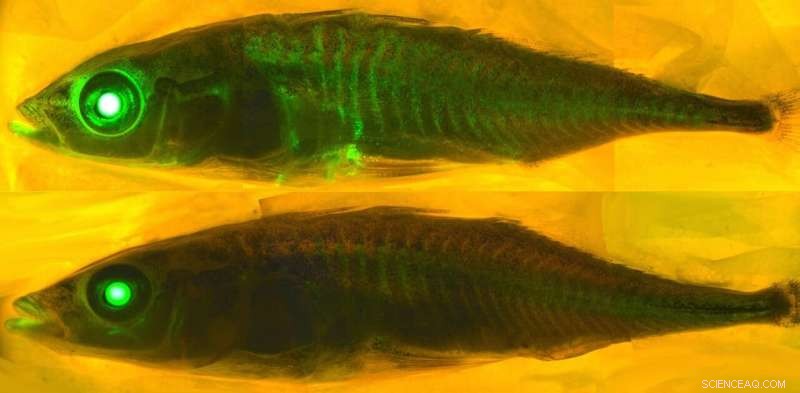
Comprendre ce qui fait briller certaines protéines nécessite une compréhension de la chimie. Crédit :eLife - la revue, CC BY-SA
L'intelligence artificielle a changé la façon dont la science est pratiquée en permettant aux chercheurs d'analyser les quantités massives de données générées par les instruments scientifiques modernes. Il peut trouver une aiguille dans un million de meules de foin d'informations et, grâce à l'apprentissage en profondeur, il peut apprendre des données elles-mêmes. L'IA accélère les progrès dans la recherche de gènes, la médecine, la conception de médicaments et la création de composés organiques.
L'apprentissage en profondeur utilise des algorithmes, souvent des réseaux de neurones formés sur de grandes quantités de données, pour extraire des informations à partir de nouvelles données. Il est très différent de l'informatique traditionnelle avec ses instructions étape par étape. Au contraire, il apprend à partir des données. L'apprentissage en profondeur est beaucoup moins transparent que la programmation informatique traditionnelle, ce qui laisse des questions importantes :qu'est-ce que le système a appris, que sait-il ?
En tant que professeur de chimie, j'aime concevoir des tests qui comportent au moins une question difficile qui étend les connaissances des étudiants pour déterminer s'ils peuvent combiner différentes idées et synthétiser de nouvelles idées et concepts. Nous avons conçu une telle question pour l'affiche des défenseurs de l'IA, AlphaFold, qui a résolu le problème du repliement des protéines.
Repliement des protéines
Les protéines sont présentes dans tous les organismes vivants. Ils structurent les cellules, catalysent les réactions, transportent de petites molécules, digèrent les aliments et bien plus encore. Ils sont constitués de longues chaînes d'acides aminés comme des perles sur une ficelle. Mais pour qu'une protéine fasse son travail dans la cellule, elle doit se tordre et se plier en une structure tridimensionnelle complexe, un processus appelé repliement des protéines. Les protéines mal repliées peuvent entraîner des maladies.
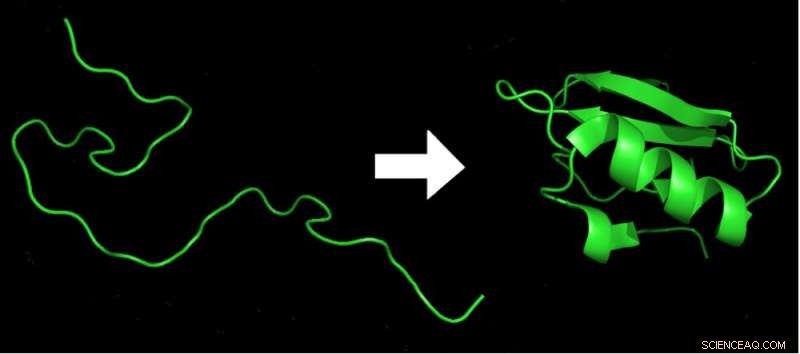
Quelques millisecondes après la sortie d'une chaîne d'acides aminés (à gauche) du ribosome, elle est repliée dans la forme 3D à plus faible énergie (à droite), nécessaire au fonctionnement de la protéine. Crédit :Marc Zimmer, CC BY-ND
Dans son discours d'acceptation du prix Nobel de chimie en 1972, Christiaan Anfinsen a postulé qu'il devrait être possible de calculer la structure tridimensionnelle d'une protéine à partir de la séquence de ses éléments constitutifs, les acides aminés.
Tout comme l'ordre et l'espacement des lettres dans cet article lui donnent un sens et un message, l'ordre des acides aminés détermine l'identité et la forme de la protéine, ce qui se traduit par sa fonction.
En raison de la flexibilité inhérente des éléments constitutifs des acides aminés, une protéine typique peut adopter environ 10 à la puissance de 300 formes différentes. C'est un nombre énorme, plus que le nombre d'atomes dans l'univers. Pourtant, en une milliseconde, chaque protéine d'un organisme se repliera dans sa propre forme spécifique - l'arrangement le plus énergétique de toutes les liaisons chimiques qui composent la protéine. Changez un seul acide aminé parmi les centaines d'acides aminés que l'on trouve généralement dans une protéine et il peut mal se replier et ne plus fonctionner.
Pli Alpha
Pendant 50 ans, les informaticiens ont essayé de résoudre le problème du repliement des protéines, avec peu de succès. Puis, en 2016, DeepMind, une filiale d'intelligence artificielle d'Alphabet, la société mère de Google, a lancé son programme AlphaFold. Il a utilisé la banque de données sur les protéines comme ensemble d'apprentissage, qui contient les structures déterminées expérimentalement de plus de 150 000 protéines.
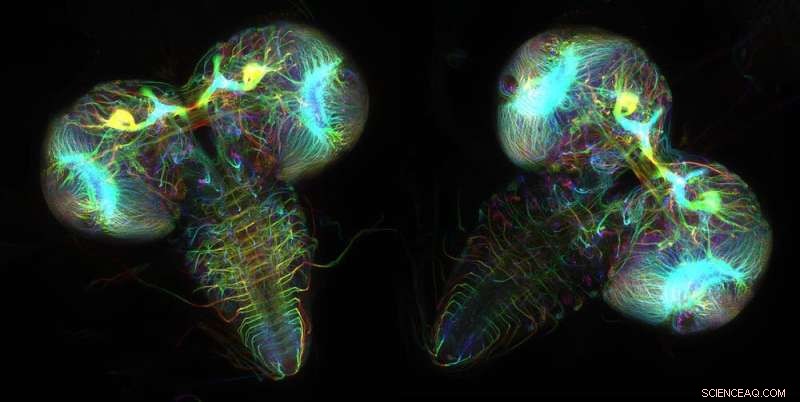
Des neurones exprimant des protéines fluorescentes révèlent les structures cérébrales de deux larves de mouches des fruits. Crédit :Wen Lu et Vladimir I. Gelfand, Feinberg School of Medicine, Northwestern University
En moins de cinq ans, AlphaFold a surmonté le problème du repliement des protéines, du moins la partie la plus utile, à savoir la détermination de la structure de la protéine à partir de sa séquence d'acides aminés. AlphaFold n'explique pas comment les protéines se replient si rapidement et avec précision. Il s'agissait d'une victoire majeure pour l'IA, car elle n'a pas seulement valu un énorme prestige scientifique, c'est aussi une avancée scientifique majeure qui pourrait affecter la vie de chacun.
Aujourd'hui, grâce à des programmes comme AlphaFold2 et RoseTTAFold, des chercheurs comme moi peuvent déterminer la structure tridimensionnelle des protéines à partir de la séquence d'acides aminés qui composent la protéine, sans frais, en une heure ou deux. Avant AlphaFold2, nous devions cristalliser les protéines et résoudre les structures à l'aide de la cristallographie aux rayons X, un processus qui prenait des mois et coûtait des dizaines de milliers de dollars par structure.
Nous avons désormais également accès à la base de données de structure des protéines AlphaFold, où Deepmind a déposé les structures 3D de presque toutes les protéines présentes chez l'homme, la souris et plus de 20 autres espèces. À ce jour, ils ont résolu plus d'un million de structures et prévoient d'ajouter 100 millions de structures supplémentaires cette année seulement. La connaissance des protéines a explosé. La structure de la moitié de toutes les protéines connues devrait être documentée d'ici la fin de 2022, parmi lesquelles de nombreuses nouvelles structures uniques associées à de nouvelles fonctions utiles.
Penser comme un chimiste
AlphaFold2 n'a pas été conçu pour prédire comment les protéines interagiraient les unes avec les autres, mais il a été capable de modéliser la façon dont les protéines individuelles se combinent pour former de grandes unités complexes composées de plusieurs protéines. Nous avions une question difficile pour AlphaFold :son ensemble d'entraînement structurel lui avait-il appris un peu de chimie ? Pourrait-il dire si les acides aminés réagiraient les uns avec les autres - un événement rare mais important ?
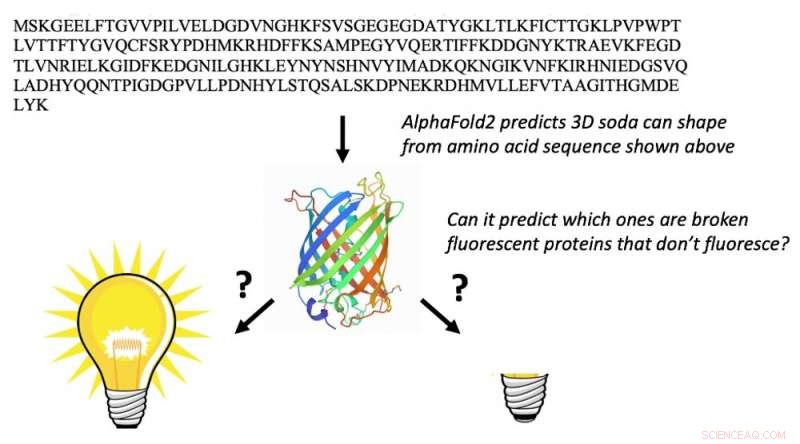
AlphaFold2 peut prendre la séquence d'acides aminés des protéines fluorescentes (lettres en haut) et prédire leurs formes de tonneau 3D (au milieu). Ce n'est pas surprenant. Ce qui est totalement inattendu, c'est qu'il peut également prédire quelles protéines fluorescentes sont « cassées » et ne peuvent pas être fluorescentes. Crédit :Marc Zimmer, CC BY-ND
Je suis un chimiste informaticien intéressé par les protéines fluorescentes. Ce sont des protéines présentes dans des centaines d'organismes marins comme les méduses et les coraux. Leur lueur peut être utilisée pour éclairer et étudier les maladies.
Il y a 578 protéines fluorescentes dans la banque de données de protéines, dont 10 sont "cassés" et ne sont pas fluorescents. Les protéines s'attaquent rarement elles-mêmes, un processus appelé modification post-traduction autocatalytique, et il est très difficile de prédire quelles protéines réagiront avec elles-mêmes et lesquelles ne le feront pas.
Seul un chimiste possédant une quantité significative de connaissances sur les protéines fluorescentes serait en mesure d'utiliser la séquence d'acides aminés pour trouver les protéines fluorescentes qui ont la bonne séquence d'acides aminés pour subir les transformations chimiques nécessaires pour les rendre fluorescentes. Lorsque nous avons présenté AlphaFold2 avec les séquences de 44 protéines fluorescentes qui ne sont pas dans la banque de données de protéines, il a replié les protéines fluorescentes fixées différemment de celles cassées.
Le résultat nous a stupéfaits :AlphaFold2 avait appris un peu de chimie. Il avait découvert quels acides aminés dans les protéines fluorescentes font la chimie qui les fait briller. Nous soupçonnons que l'ensemble d'apprentissage de la banque de données sur les protéines et les alignements de séquences multiples permettent à AlphaFold2 de "penser" comme des chimistes et de rechercher les acides aminés nécessaires pour réagir les uns avec les autres pour rendre la protéine fluorescente.
Un programme de pliage apprenant de la chimie à partir de son ensemble de formation a également des implications plus larges. En posant les bonnes questions, que peut-on encore gagner d'autres algorithmes d'apprentissage en profondeur ? Les algorithmes de reconnaissance faciale pourraient-ils trouver des marqueurs cachés de maladies ? Les algorithmes conçus pour prédire les habitudes de dépenses des consommateurs pourraient-ils également trouver une propension au vol mineur ou à la tromperie ? Et le plus important, est-ce que cette capacité - et des sauts de capacité similaires dans d'autres systèmes d'IA - est souhaitable ?
Cet article est republié de The Conversation sous une licence Creative Commons. Lire l'article d'origine. Les protéines se souviennent-elles ?
