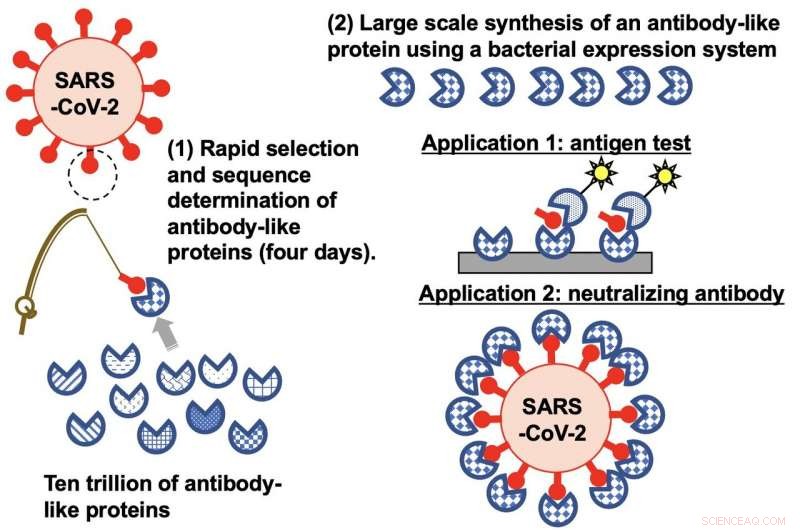
La méthode d'affichage TRAP « pêche » des protéines synthétiques parmi une bibliothèque de milliers de milliards pour celles qui peuvent cibler le SRAS-CoV-2. L'approche a permis d'identifier des protéines qui peuvent être utilisées pour tester le virus et potentiellement traiter les personnes infectées par COVID-19. Crédit :Hiroshi Murakami
Une équipe de recherche dirigée par des scientifiques de l'Université de Nagoya au Japon a développé une approche qui permet de trouver rapidement des protéines synthétiques qui se lient spécifiquement à des cibles importantes, tels que les composants du virus SARS-CoV-2. La méthode a été publiée dans la revue Avancées scientifiques et pourrait être utilisé pour développer des kits de test ou pour trouver des traitements.
"Nous avons développé une technique de laboratoire pour la sélection rapide de protéines synthétiques qui se lient fortement au SARS-CoV-2, ", explique Hiroshi Murakami, ingénieur biomoléculaire de l'Université de Nagoya. "Les protéines synthétiques de haute affinité peuvent être utilisées pour développer des tests antigéniques sensibles pour le SRAS-CoV-2 et pour une utilisation future comme anticorps de neutralisation chez les patients infectés."
Murakami et ses collègues avaient déjà développé un test de laboratoire de sélection de protéines appelé TRAP display, qui signifie "transcription-traduction couplée à l'association d'un linker puromycine". Leur approche saute deux étapes chronophages dans une autre technique couramment utilisée pour rechercher dans les bibliothèques de protéines synthétiques. Mais leurs investigations ont indiqué qu'il y avait un problème avec le linker de puromycine.
Dans l'étude actuelle, l'équipe a amélioré sa technique en modifiant le linker puromycine. Finalement, ils ont pu utiliser leur écran TRAP pour identifier neuf protéines synthétiques qui se lient à la protéine de pointe sur la membrane externe du SRAS-CoV-2. L'approche n'a pris que quatre jours par rapport aux semaines qu'il faudrait avec la technologie d'affichage de l'ARN messager couramment utilisée.
L'affichage TRAP implique l'utilisation d'un grand nombre de matrices d'ADN qui codent et synthétisent des milliards de protéines portant des séquences peptidiques aléatoires. Les protéines synthétiques sont liées à l'ADN à l'aide du linker puromycine modifié, puis exposées à une protéine cible. Lorsque tout l'échantillon est lavé, seules les protéines synthétiques qui se lient à la cible restent. Celles-ci sont ensuite replacées dans l'affichage TRAP pour d'autres tours jusqu'à ce qu'il ne reste plus qu'un petit nombre de protéines synthétiques de liaison à la cible très spécifiques.
Les chercheurs ont étudié les neuf protéines synthétiques qui se sont avérées se lier au SRAS-CoV-2. Certains ont été spécifiquement capables de détecter le SRAS-CoV-2 dans des écouvillonnages nasaux de patients COVID-19, indiquant qu'ils pourraient être utilisés dans des kits de test. On s'attache également au virus pour l'empêcher de se lier aux récepteurs qu'il utilise pour accéder aux cellules humaines. Cela suggère que cette protéine pourrait être utilisée comme stratégie de traitement.
"Notre grande vitesse, l'affichage amélioré de TRAP pourrait être utile pour mettre en œuvre des réponses rapides aux sous-espèces du SRAS-CoV-2 et à d'autres nouveaux virus potentiels provoquant de futures pandémies, " dit Murakami.
Cette étude, "Protéines de type anticorps qui capturent et neutralisent le SARS-CoV-2, " a été publié en ligne dans Avancées scientifiques le 18 septembre, 2020.