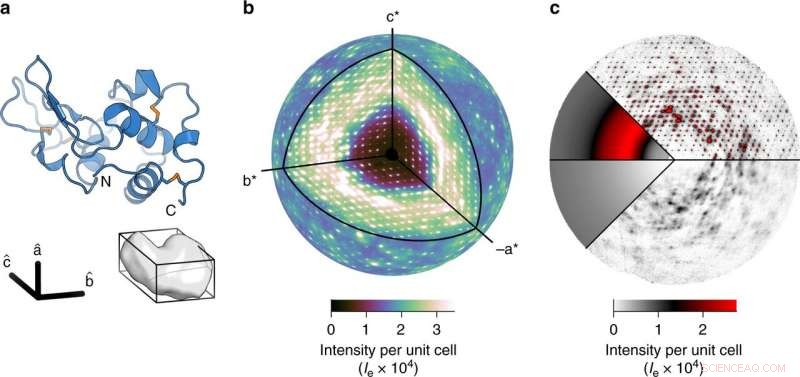
a Diagramme en ruban du lysozyme (en haut) et de la cellule unitaire triclinique contenant une protéine (en bas). b Une carte tridimensionnelle très détaillée de la diffusion diffuse a été obtenue. La sphère extérieure est dessinée à une résolution de 2 Å. c La diffusion totale est constituée de trois composantes :diffusion Compton inélastique (en bas à gauche), un large anneau isotrope qui domine le signal diffus (en haut à gauche), et les caractéristiques variationnelles de la diffusion diffuse (à droite). Des halos intenses sont visibles dans les couches contenant des pics de Bragg (plan l = 0, En haut à droite). La diffusion nuageuse est mieux visualisée dans les plans à mi-chemin entre les pics de Bragg (l = 1∕2 plan, en bas à droite). Crédit: Communication Nature (2020). DOI :10.1038/s41467-020-14933-6
Les biologistes structurels de Cornell ont adopté une nouvelle approche en utilisant une méthode classique d'analyse aux rayons X pour capturer quelque chose que la méthode conventionnelle n'avait jamais pris en compte :le mouvement collectif des protéines. Et ils l'ont fait en créant un logiciel pour assembler minutieusement les fragments de données qui sont généralement ignorés dans le processus.
Leur papier, "Diffusion diffuse des rayons X à partir de mouvements corrélés dans un cristal de protéine, " publié le 9 mars dans Communication Nature .
En tant que biologiste structurale, Nozomi Ando, MME. '04, doctorat '08, professeur assistant de chimie et biologie chimique, s'intéresse à la cartographie du mouvement des protéines, et leurs parties internes, pour mieux comprendre la fonction des protéines. Ce type de mouvement est bien connu mais a été difficile à documenter car la technique standard pour l'imagerie des protéines est la cristallographie aux rayons X, qui produit des instantanés essentiellement statiques.
« Parce que nous étudions des systèmes biologiques vraiment difficiles, le groupe doit aussi souvent être pionnier de nouvelles méthodes structurelles, " a déclaré le chercheur postdoctoral Steve Meisburger, doctorat '14, l'auteur principal du journal. "L'une des questions qui nous intéresse depuis le début est de savoir comment les mouvements respiratoires subtils d'une protéine dirigent la fonction biochimique."
Les chercheurs ont apporté leur projet à la Cornell High Energy Synchrotron Source (CHESS), où ils ont profité du détecteur à matrice de pixels Pilatus 6M de l'installation, ce qui leur a permis de faire des images à très haute résolution.
Pour ce travail, comme en cristallographie ordinaire, Des rayons X ont été émis sur un échantillon de cristal. Le détecteur à matrice de pixels a enregistré l'intensité des rayons X diffractés par les protéines du cristal, codant ainsi la structure atomique. Tout trouble, c'est-à-dire mouvement - à l'intérieur du cristal a fait rebondir des photons supplémentaires, créant un signal de fond très faible appelé diffusion diffuse. Ces informations ont été traditionnellement ignorées lors du traitement des données.
"Les photons vont partout, et le signal semble extrêmement faible parce qu'il est étalé, " dit Ando, l'auteur principal de l'article. "Depuis des décennies, les gens ne pouvaient pas le mesurer avec précision, et ils ne savaient pas comment l'interpréter.
Meisburger a créé un logiciel pour traiter les quelque 50 millions de points de données uniques, résultant en une carte tridimensionnelle de haute qualité. A la grande surprise des chercheurs, la carte a révélé qu'une composante importante de ce modèle de diffusion diffuse était en fait le résultat de la vibration du réseau protéique. Ce mouvement saccadé était si dominant, il semblait masquer tout mouvement à l'intérieur des protéines, ce qui a d'abord été une déception pour les chercheurs.
Mais après avoir pris en compte ces vibrations de réseau dans les simulations, les chercheurs ont également identifié les mouvements internes des protéines. Ces mouvements comprenaient l'ouverture et la fermeture du site actif de la protéine.
"Imaginez que le cristal soit comme une rangée de personnes essayant de marcher ensemble tout en se tenant la main, mais en même temps, chaque individu peut faire quelque chose de légèrement différent, " Ando a dit. " Le signal de tout le monde se déplaçant ensemble est dominant, nous ne pouvions donc pas discerner le signal subtil qui venait des individus. C'était quelque chose qui n'avait jamais été expliqué."
Cette nouvelle approche de la diffusion diffuse pourrait aider les chercheurs à obtenir une image plus claire de la structure et de la dynamique des protéines et, finalement, une meilleure compréhension des réactions biochimiques.
"Nous voulons vraiment pousser cela dans une direction où beaucoup de gens peuvent utiliser la technique et apprendre quelque chose de nouveau sur leur protéine, " Meisburger a dit. " Une grande chose à ce sujet est que vous obtenez gratuitement la diffusion diffuse chaque fois que vous faites une expérience de cristallographie régulière. Cette technique ajoute vraiment des informations à ce que vous obtiendriez normalement."