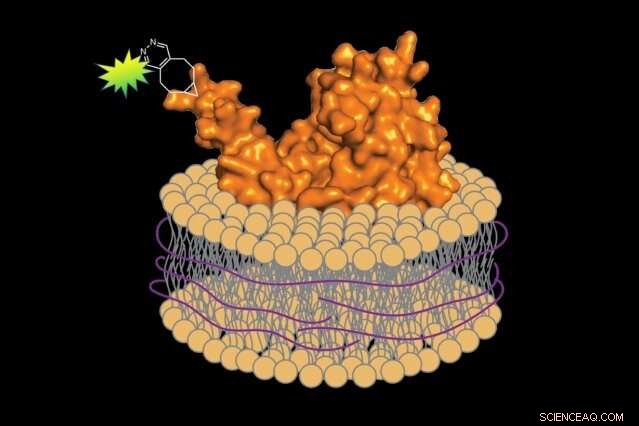
En utilisant une méthode sans détergent, les biologistes peuvent préparer des protéines marquées par fluorescence avec un petit segment de la membrane cellulaire associée, préserver l'environnement natif de la protéine. Crédit :Jean-Marie Swiecicki
Toutes les cellules ont une membrane lipidique qui entoure leurs composants internes, formant une barrière protectrice pour contrôler ce qui entre et ce qui reste à l'extérieur. Les protéines intégrées dans ces membranes sont essentielles à la vie; ils aident à faciliter le transport des nutriments, conversion et stockage d'énergie, et la communication cellulaire. Ils sont également importants dans les maladies humaines, et représentent environ 60 pour cent des cibles médicamenteuses approuvées. Afin d'étudier ces protéines membranaires en dehors de la complexité de la cellule, les chercheurs doivent utiliser un détergent pour enlever la membrane et les extraire. Cependant, déterminer le meilleur détergent pour chaque protéine peut impliquer de nombreux essais et erreurs. Et, retirer une protéine de son environnement naturel risque de déstabiliser la structure repliée et de perturber la fonction.
Dans une étude publiée le 9 décembre dans Biologie Chimique Cellulaire , des scientifiques du MIT ont mis au point un moyen rapide et généralisable d'extraire, purifier, et marquer les protéines membranaires pour l'imagerie sans aucun détergent, en amenant une partie de la membrane environnante pour protéger la protéine et simuler son environnement naturel. Leur approche combine des techniques chimiques et biochimiques bien établies d'une manière nouvelle, isoler efficacement la protéine afin qu'elle puisse être marquée par fluorescence et examinée au microscope.
"Je plaisante toujours en disant que ce n'est pas très réaliste d'étudier les protéines dans le savon, " dit l'auteur principal Barbara Imperiali, professeur de biologie et de chimie. "Nous avons créé un flux de travail qui permet aux protéines membranaires d'être imagées tout en conservant leurs identités et interactions natives. Espérons que maintenant moins de personnes hésiteront à étudier les protéines membranaires, étant donné leur importance dans de nombreux processus physiologiques.
En tant que membre du laboratoire Imperiali, l'ancien postdoctorant et auteur principal Jean-Marie Swiecicki a étudié les protéines membranaires du pathogène d'origine alimentaire Campylobacter jejuni. Dans cette étude, Swiecicki s'est concentré sur PglC et PglA, deux protéines membranaires qui jouent un rôle en permettant aux bactéries d'infecter les cellules humaines. Ses expériences nécessitaient de marquer PglC et PglA avec des marqueurs fluorescents afin de les suivre. Cependant, il n'était pas satisfait des méthodes existantes pour le faire.
Dans certains cas, les balises fluorescentes qui doivent être incorporées dans la protéine afin de la visualiser sont trop grandes pour être placées à des positions définies. Dans d'autres cas, ces balises ne brillent pas assez, ou interférer avec la structure et la fonction de la protéine.
Pour éviter de tels problèmes, Swiecicki a décidé d'utiliser une méthode connue sous le nom de « mutagenèse non naturelle des acides aminés ». Les acides aminés sont les unités qui composent la protéine, et la mutagenèse non naturelle d'acides aminés implique l'ajout d'un nouvel acide aminé contenant un groupe chimique modifié dans la séquence protéique. Ce groupe chimique peut ensuite être étiqueté avec une étiquette brillante.
Swiecicki a inséré le code génétique des protéines membranaires de C. jejuni dans une bactérie différente, Escherichia coli. À l'intérieur d'E. coli, il pourrait incorporer l'acide aminé non naturel, qui pourrait être modifié chimiquement pour ajouter le marqueur fluorescent.
Au moment de retirer les protéines de la membrane, il a substitué une substance différente au détergent :un polymère d'acide styrène-maléique (SMA). Contrairement au détergent, SMA enveloppe la protéine extraite et un petit segment de la membrane associée dans une enveloppe protectrice, préserver son environnement d'origine. Imperiali explique, "C'est comme une écharpe protégeant votre cou du froid."
Swiecicki a ensuite pu surveiller les protéines incandescentes au microscope pour vérifier que sa technique était suffisamment sélective pour isoler les protéines membranaires individuelles. L'ensemble du processus, il dit, ne prend que quelques jours, et est généralement beaucoup plus rapide et plus fiable que les méthodes d'extraction à base de détergent, qui peut prendre des mois et nécessiter l'expertise de biochimistes hautement qualifiés pour être optimisée.
"Je ne dirais pas que c'est une solution miracle qui fonctionnera pour chaque protéine, " dit-il. " Mais c'est un outil très efficace qui pourrait faciliter l'étude de nombreux types différents de protéines membranaires. " Finalement, il dit, cela peut même aider à faciliter les criblages de médicaments à haut débit.
"En tant que personne qui travaille sur les complexes de protéines membranaires, Je peux témoigner du grand besoin de meilleures méthodes pour les étudier, " dit Suzanne Walker, un professeur de microbiologie à la Harvard Medical School qui n'a pas participé à l'étude. Elle espère étendre l'approche décrite dans l'article aux complexes protéiques qu'elle étudie dans son propre laboratoire. « J'ai apprécié les nombreux détails inclus dans le texte sur la façon d'appliquer la stratégie avec succès, " Elle ajoute.
Les prochaines étapes seront de tester la technique sur des protéines de mammifères, et isoler plusieurs protéines à la fois dans la coque SMA pour observer leurs interactions. Et, bien sûr, chaque nouvelle technique mérite un nom. "Nous travaillons toujours sur un acronyme accrocheur, " dit Imperiali. " Des idées ? "
Cette histoire est republiée avec l'aimable autorisation de MIT News (web.mit.edu/newsoffice/), un site populaire qui couvre l'actualité de la recherche du MIT, innovation et enseignement.