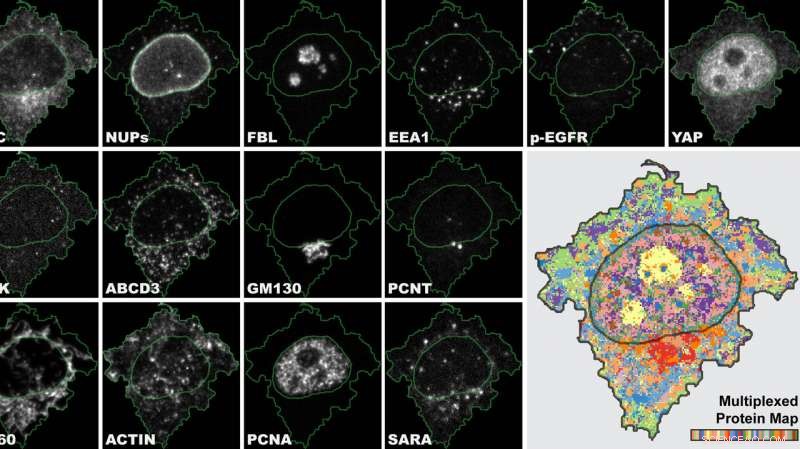
4i est la première technique d'imagerie qui nous donne une vue multiplexée tissu-organelle d'échantillons biologiques et relie plusieurs informations dans une seule et même expérience. Crédit :UZH
Des chercheurs de l'Université de Zurich ont développé une nouvelle méthode d'analyse des cellules et de leurs composants appelée imagerie d'immunofluorescence indirecte itérative (4i). Cette innovation affine considérablement la technique d'imagerie par immunofluorescence standard utilisée en biomédecine et fournit aux cliniciens une énorme quantité de données à partir de chaque échantillon individuel. 4i permet d'observer la répartition spatiale d'au moins 40 protéines et leurs modifications dans une même cellule pour des centaines de milliers de cellules simultanément à différents niveaux, du tissu jusqu'au niveau des organites.
Dix fois plus de protéines visualisées en même temps
"4i est la première technique d'imagerie qui nous donne une vue multiplexée tissu-organelle d'échantillons biologiques. Nous pouvons, pour la première fois, lier les informations multiplexées dérivées au niveau du tissu, niveau cellulaire et subcellulaire dans une même expérience, " dit Gabriele Gut, auteur principal de l'étude et chercheur postdoctoral à l'Institut des sciences de la vie moléculaire de l'UZH.
L'immunofluorescence (IF) utilise des anticorps pour visualiser et localiser des protéines dans des échantillons biologiques. Alors que la méthode IF standard marque généralement trois protéines, 4i utilise des anticorps du commerce et des microscopes à fluorescence conventionnels pour visualiser dix fois plus de protéines par hybridation itérative et élimination des anticorps de l'échantillon. "Imaginez que les biologistes cellulaires soient des journalistes. Chaque expérience est un entretien avec nos cellules. Avec la FI conventionnelle, Je peux poser trois questions, alors qu'avec 4i, Je peux avoir une discussion sur plus de 40 sujets, " explique Gabriele Gut.
La carte fournit une étude systématique du paysage cellulaire
Une fois acquis, l'énorme quantité de données doit ensuite être analysée, le prochain obstacle pour les chercheurs. "Nous avons généré des images avec une résolution subcellulaire pour des milliers de cellules pour 40 canaux pour plus de 10 conditions de traitement. L'œil et le cerveau humains ne peuvent pas traiter la complexité biologique collectée par 4i."
Pour utiliser pleinement les données 4i, Gabriele Gut a développé un nouveau programme informatique pour la visualisation et l'analyse appelé cartes de protéines multiplexées. Il extrait le signal de fluorescence multiplexé pour des millions de pixels et génère une carte abstraite mais représentative de la distribution des protéines multiplexées dans les cellules.
Les chercheurs ont ainsi pu générer une étude systématique du paysage cellulaire :ils ont réussi à visualiser l'organisation spatiale intracellulaire de la plupart des organites de mammifères tout au long du cycle cellulaire et dans différents microenvironnements.
Faire progresser la médecine de précision
Les applications des cartes de protéines 4i et multiplexées sont multiples, allant de la recherche fondamentale à la médecine de précision. "Nous espérons que les cartes de protéines 4i et multiplexées aideront les chercheurs à mieux comprendre les processus qui sont au centre de la recherche biologique depuis des décennies, " dit Gut. En même temps, les chercheurs prévoient d'utiliser ces technologies pour faire avancer les frontières de la médecine de précision, en particulier dans le diagnostic du cancer et la sélection des thérapies.
Une nouvelle méthode déjà appliquée en thérapie tumorale
La nouvelle méthode d'analyse itérative d'imagerie par immunofluorescence indirecte (4i) peut également être utilisée pour déterminer les effets de substances pharmacologiques sur l'organisation et la physiologie des cellules. Il est actuellement utilisé dans une collaboration de recherche translationnelle avec des cliniciens et une société pharmaceutique dans le but d'améliorer les résultats du traitement des patients atteints de cancer. Lucas Pelkmans, professeur à l'Institut des sciences de la vie moléculaire de l'UZH, et son équipe de recherche visent à caractériser les cellules tumorales de patients qui ont été traités avec différents médicaments anticancéreux. Les scientifiques espèrent que les résultats de laboratoire fourniront des informations pour soutenir la prise de décision clinique pour le traitement individuel des patients. De plus, les chercheurs prévoient de mettre en œuvre des cartes de protéines 4i et multiplexées sur des coupes tissulaires de tumeurs pour identifier des biomarqueurs pertinents et ainsi améliorer les diagnostics et les pronostics des patients atteints de cancer.