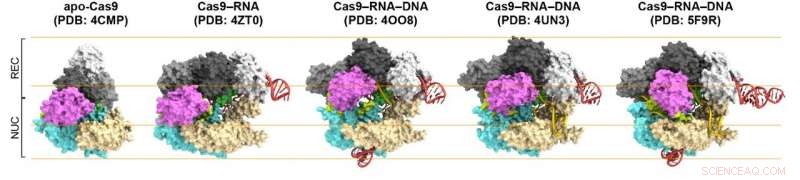
Figure 1. Structures de Cas9. De gauche à droite :Cas9 seul (apo-Cas9), Cas9 lié à l'ARN (Cas9-ARN), Cas9-ARN lié à sa cible d'ADN simple brin (Cas9-ARN-ADN), Cas9-ARN lié à un duplex d'ADN partiel (Cas9-ARN-ADN) et Cas9-ARN lié à sa cible d'ADN double brin (Cas9-ARN-ADN). Crédit :Université de Kanazawa
Des chercheurs de l'Université de Kanazawa et de l'Université de Tokyo rapportent dans Communication Nature la visualisation de la dynamique des « ciseaux moléculaires », le principal mécanisme de la technique de génie génétique CRISPR-Cas9.
L'une des techniques utilisées en génie génétique - le processus de modification artificielle du génome d'un organisme vivant - implique le système de nucléase CRISPR-Cas9. En utilisant ce système, l'ADN d'une cellule peut être coupé à un site souhaité, où les gènes peuvent être supprimés ou ajoutés. La sélection du site à couper se fait par une molécule « ARN guide » liée à la protéine Cas9. Maintenant, une équipe de chercheurs dirigée par Mikihiro Shibata de l'université de Kanazawa et Osamu Nureki de l'université de Tokyo a visualisé la dynamique du complexe CRISPR-Cas9, en particulier comment il coupe l'ADN, fournissant des informations précieuses sur le mécanisme de clivage de l'ADN médié par CRISPR-Cas9.
Pour leurs études de visualisation, les scientifiques ont utilisé la microscopie à force atomique à grande vitesse (HS-AFM), une méthode d'imagerie de surfaces. Une surface est sondée en déplaçant un petit porte-à-faux dessus ; la force subie par la sonde peut être convertie en une mesure de hauteur. Un balayage de toute la surface donne alors une carte de hauteur de l'échantillon. La configuration expérimentale à grande vitesse de Shibata et de ses collègues a permis de scans répétés - convertibles en films - des biomolécules participant à l'action de cisaillement moléculaire.
D'abord, les scientifiques ont comparé Cas9 sans et avec ARN attaché (Cas9-ARN). Ils ont constaté que le premier était capable d'adopter avec souplesse diverses conformations, tandis que ce dernier a un fixe, structure à deux lobes, mettant en évidence la capacité de stabilisation conformationnelle de l'ARN guide. Puis, Shibata et ses collègues ont examiné comment le complexe Cas9-ARN stabilisé cible l'ADN. Ils ont confirmé qu'il se lie à un site de motif adjacent de protospacer (PAM) présélectionné dans l'ADN. Un PAM est une courte séquence de nucléotides située à côté du site cible de l'ADN, qui est complémentaire de l'ARN guide.
Les films à grande vitesse de l'équipe de recherche ont en outre révélé que le ciblage (« interrogation de l'ADN ») est obtenu grâce à la diffusion en 3D du complexe Cas9-ARN. Finalement, les chercheurs ont réussi à visualiser la dynamique du processus de clivage lui-même :ils ont observé comment la région des « ciseaux moléculaires » subit des fluctuations conformationnelles après que Cas9-ARN a déroulé localement l'ADN double brin (film 1[URL]).
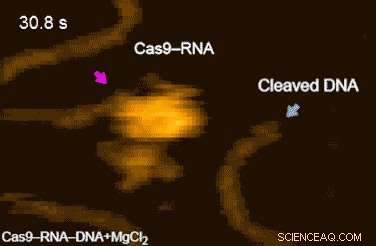
Figure 2. Films HS-AFM du clivage de l'ADN par Cas9-ARN. Les fluctuations du domaine de la nucléase sont indiquées par des flèches magenta. Les produits de clivage libérés de Cas9-ARN sont indiqués par des flèches bleues. Crédit :Université de Kanazawa
Les travaux de Shibata font progresser notre compréhension du mécanisme d'édition du génome CRISPR-Cas9. Selon les mots des chercheurs :« … cette étude fournit des détails sans précédent sur la dynamique fonctionnelle de CRISPR-Cas9, et met en évidence le potentiel de la HS-AFM pour élucider les mécanismes d'action des nucléases effectrices guidées par l'ARN à partir de systèmes CRISPR-Cas distincts."
CRISPR-Cas9
CRISPR, abréviation de « répétitions palindromiques courtes regroupées régulièrement espacées », désigne un ensemble de séquences d'ADN bactérien contenant des fragments d'ADN de virus ayant précédemment attaqué les bactéries. Ces fragments sont utilisés par les bactéries pour empêcher d'autres attaques par les mêmes virus. "Cas" fait référence aux gènes associés à CRISPR; "Cas9" est une protéine associée à CRISPR avec deux domaines de nucléase (Une nucléase est une enzyme capable de cliver les acides nucléiques, molécules organiques présentes dans l'ADN et l'ARN).
Dans les années récentes, une technique de génie génétique dans laquelle un complexe CRISPR-Cas9 agit comme « ciseaux moléculaires » a été mise au point ; la nucléase Cas9 se lie à une molécule d'ARN guide qui contient des informations sur le site d'ADN à cibler. En utilisant la microscopie à force atomique à grande vitesse, Mikihiro Shibata de l'Université de Kanazawa et ses collègues ont maintenant étudié en détail la dynamique du complexe CRISPR-Cas9.
Microscopie à force atomique
La microscopie à force atomique (AFM) est une technique d'imagerie dans laquelle l'image est formée en balayant une surface avec une très petite pointe. Le mouvement de balayage horizontal de la pointe est contrôlé via des éléments piézoélectriques, while vertical motion is converted into a height profile, resulting in a height distribution of the sample's surface. As the technique does not involve lenses, its resolution is not restricted by the so-called diffraction limit. In a high-speed setup, AFM can be used to produce movies of a sample's evolution in real time. High-speed AFM has been used successfully to study protein dynamics, for example myosin V walking on an actin filament, the photo-induced conformational change of bacteriorhodopsin, and the degradation of cellulose. Shibata and colleagues have now applied the high-speed AFM technique for visualizing the dynamics of DNA cleavage by CRISPR-Cas9.