
Chaque sous-unité protéique est de couleur différente. Le domaine V1 est le premier, Vo en bas, tiges périphériques gauche et droite. L'arrière-plan montre une pompe à eau éolienne. Crédit :IST Autriche, 2019
Les cellules dépendent de complexes protéiques appelés ATP synthases ou ATPases pour leurs besoins énergétiques. Les molécules d'adénosine triphosphate (ATP) alimentent la plupart des processus qui maintiennent la vie. Le professeur Leonid Sazanov, biologiste structuraliste et son groupe de recherche de l'Institut des sciences et technologies d'Autriche (IST Austria) à Klosterneuburg, L'Autriche a maintenant déterminé la première structure atomique du représentant de la famille V/A-ATPase, combler le vide dans l'arbre évolutif de ces machines moléculaires essentielles. Ces résultats obtenus à l'aide des dernières méthodes de cryomicroscopie électronique ont révélé une structure similaire de turbine ou de moulin à eau de l'enzyme et ont maintenant été publiés dans la revue Science .
Puissance rotative
Les ATP synthases/ATPases sont de grands complexes de protéines membranaires qui partagent des plans de construction bruts globaux et des mécanismes de catalyse rotative. Cette famille de protéines comprend l'enzyme de type F présente dans les mitochondries (usines énergétiques de la cellule), les chloroplastes (organites qui effectuent la photosynthèse chez les plantes) et les bactéries; Le type V (vacuolaire) trouvé dans les compartiments intracellulaires chez les eucaryotes (organismes supérieurs avec un noyau) et le type A (archéal) trouvé chez les procaryotes - les archées (micro-organismes anciens) et certaines bactéries.
Différentes saveurs d'ATPases
Les enzymes de type F et A fonctionnent généralement pour produire de l'ATP, entraîné par le flux de protons à travers la membrane. Les enzymes de type V fonctionnent généralement en sens inverse, en utilisant l'ATP pour pomper des protons. Les V- et A-ATPases sont structurellement similaires, mais elles diffèrent du type F en ayant deux ou trois tiges périphériques et des sous-unités protéiques de connexion supplémentaires entre V1 et Vo. Les enzymes de type V ont probablement évolué à partir du type A et en raison de ces similitudes, le type A est également appelé V/A-ATPase. Certaines bactéries, comprenant Thermus thermophilus , acquis une enzyme de type A. Long Zhou, post-doctorant dans le groupe de recherche Sazanov de l'IST Autriche, a purifié et étudié cette enzyme (ThV1Vo) par cryo-EM. Contrairement au type F, pour les ATPases de type V, seules les structures des domaines V1 et Vo isolés ont été déterminées précédemment. Comment V1 est couplé à Vo n'était donc pas connu, et la connaissance du cycle catalytique complet manquait.
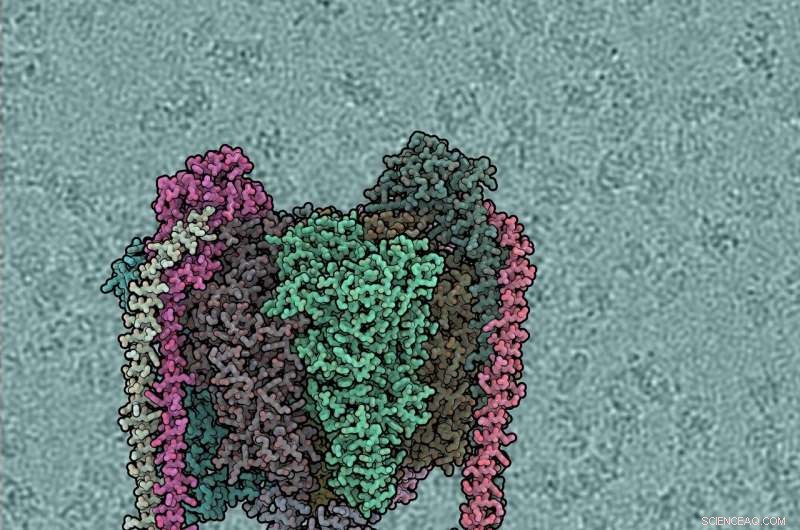
Chaque sous-unité protéique est de couleur différente. Le domaine V1 est le premier, Vo en bas, tiges périphériques gauche et droite. L'arrière-plan montre une micrographie cryo-EM brute, avec des molécules d'ATPase individuelles visibles. Crédit :IST Autriche, 2019
Plasticité et compétition
Les scientifiques n'en ont déterminé aucun, mais au total cinq structures de l'ensemble de l'enzyme ThV1Vo, utilisant des méthodes de cryomicroscopie électronique développées récemment dans la soi-disant « révolution de résolution » de cette technique. Les structures représentent plusieurs états conformationnels de l'enzyme différant par la position du rotor à l'intérieur du stator. La plasticité conformationnelle globale de ThV1Vo est révélée par une oscillation substantielle de V1 dans l'espace lors de la transition d'un état à un autre. C'est le résultat d'une compétition mécanique entre la rotation du rotor central courbé et la rigidité du stator. Le couplage V1-Vo est obtenu via une correspondance structurelle et électrostatique étroite entre l'arbre et la sous-unité spécifique de type V le reliant à l'anneau en C. La visualisation du trajet du proton a révélé des différences significatives dans la distribution des résidus de protéines chargées par rapport à celle des F-ATPases, avec un "check-point" plus strict empêchant le "glissement" de l'enzyme.
Pourquoi une complexité supplémentaire ?
Au lieu d'une seule tige périphérique d'enzymes de type F, Les types A tels que ThV1Vo ont deux tiges périphériques, tandis que les types V eucaryotes en ont trois. Mais quel est l'avantage de la complexité supplémentaire dans l'assemblage de protéines déjà très important, ainsi que des sous-unités supplémentaires reliant V1 et Vo? Le domaine F1/V1 a une symétrie triple et donc une molécule d'ATP est produite (ou consommée) pour chaque rotation de 120° du stator à l'intérieur de F1/V1. Le professeur Leonid Sazanov déclare :« Dans les V/A-ATPases, cette étape est une rotation unique de 120°, contrairement à la F-ATP synthase où elle est divisée en plusieurs sous-étapes. Ainsi, une plus grande plasticité peut être requise dans ThV1Vo afin de lier ces pas de 120 ° dans V1 à des pas plus petits par sous-unité dans l'anneau Vo c12. Cette flexibilité supplémentaire peut être offerte dans les types V par les tiges périphériques supplémentaires et les sous-unités de connexion. Nos nouvelles structures montrent comment cela est réalisé, fournissant un cadre pour l'ensemble de la famille V-ATPase".