Une équipe de scientifiques dirigée par l’Université d’Oxford a réalisé une avancée significative dans la détection de modifications sur les structures protéiques. La méthode, publiée dans Nature Nanotechnology , utilise une technologie innovante de nanopores pour identifier les variations structurelles au niveau d'une seule molécule, même au plus profond des longues chaînes protéiques.
Les cellules humaines contiennent environ 20 000 gènes codant pour des protéines. Cependant, le nombre réel de protéines observées dans les cellules est bien plus important, avec plus de 1 000 000 de structures différentes connues. Ces variantes sont générées par un processus appelé modification post-traductionnelle (PTM), qui se produit après qu'une protéine a été transcrite à partir de l'ADN.
Le PTM introduit des changements structurels tels que l’ajout de groupes chimiques ou de chaînes glucidiques aux acides aminés individuels qui composent les protéines. Il en résulte des centaines de variations possibles pour la même chaîne protéique.
Ces variantes jouent un rôle central en biologie, en permettant une régulation précise de processus biologiques complexes au sein de cellules individuelles. Cartographier cette variation permettrait de découvrir une multitude d’informations précieuses qui pourraient révolutionner notre compréhension des fonctions cellulaires. Mais jusqu'à présent, la capacité de produire des inventaires complets de protéines est restée un objectif difficile à atteindre.
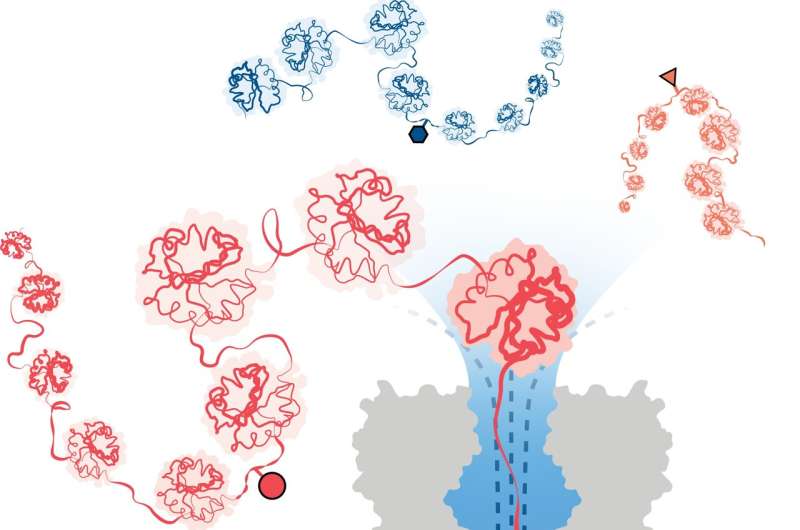
Pour surmonter ce problème, une équipe dirigée par des chercheurs du département de chimie de l'Université d'Oxford a développé avec succès une méthode d'analyse des protéines basée sur la technologie de séquençage de l'ADN/ARN des nanopores. Dans cette approche, un flux d'eau directionnel capture et déplie les protéines 3D en chaînes linéaires alimentées par de minuscules pores, juste assez larges pour qu'une seule molécule d'acide aminé puisse passer à travers.
Les variations structurelles sont identifiées en mesurant les modifications d'un courant électrique appliqué à travers le nanopore. Différentes molécules provoquent différentes perturbations du courant, leur donnant une signature unique.
L'équipe a démontré avec succès l'efficacité de la méthode dans la détection de trois modifications différentes du PTM (phosphorylation, glutathionylation et glycosylation) au niveau d'une seule molécule pour des chaînes protéiques de plus de 1 200 résidus de long. Celles-ci comprenaient des modifications profondes dans la séquence de la protéine. Il est important de noter que la méthode ne nécessite pas l'utilisation de marqueurs, d'enzymes ou de réactifs supplémentaires.
Selon l'équipe de recherche, la nouvelle méthode de caractérisation des protéines pourrait être facilement intégrée aux dispositifs portables de séquençage de nanopores existants pour permettre aux chercheurs de constituer rapidement des inventaires de protéines de cellules et de tissus uniques. Cela pourrait faciliter les diagnostics sur le lieu d'intervention, permettant la détection personnalisée de variantes protéiques spécifiques associées à des maladies telles que le cancer et les troubles neurodégénératifs.
Le professeur Yujia Qing (Département de chimie, Université d'Oxford), auteur collaborateur de l'étude, a déclaré :« Cette méthode simple mais puissante ouvre de nombreuses possibilités. Dans un premier temps, elle permet d'examiner des protéines individuelles, telles que celles impliquées dans des maladies spécifiques. . À plus long terme, la méthode a le potentiel de créer des inventaires étendus de variantes protéiques au sein des cellules, ouvrant ainsi la voie à des informations plus approfondies sur les processus cellulaires et les mécanismes pathologiques. "
Le professeur Hagan Bayley (Département de chimie, Université d'Oxford), auteur collaborateur et co-fondateur d'Oxford Nanopore Technologies, a ajouté :« La capacité d'identifier et d'identifier les modifications post-traductionnelles et autres variations protéiques au niveau d'une seule molécule est extrêmement prometteuse. pour faire progresser notre compréhension des fonctions cellulaires et des interactions moléculaires. Cela peut également ouvrir de nouvelles voies pour la médecine personnalisée, les diagnostics et les interventions thérapeutiques. "
Ce travail a été réalisé en collaboration avec le groupe de recherche du mécanobiologiste Sergi Garcia-Maynes du King's College de Londres et du Francis Crick Institute.
Plus d'informations : Pablo Martin-Baniandres et al, Détection de nanopores sans enzyme de modifications post-traductionnelles au sein de polypeptides longs, Nature Nanotechnology (2023). DOI :10.1038/s41565-023-01462-8
Informations sur le journal : Nanotechnologie naturelle
Fourni par l'Université d'Oxford