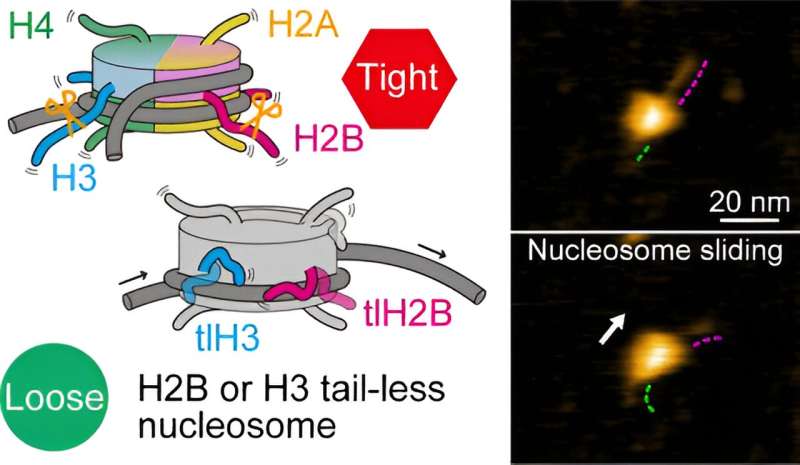
Chez les plantes et les animaux, les unités d'emballage de base de l'ADN, qui transportent l'information génétique, sont ce qu'on appelle les nucléosomes. Un nucléosome est constitué d'un segment d'ADN enroulé autour de huit protéines appelées histones.
Au cours de l’expression des gènes (le processus à la base de la production de protéines), les nucléosomes sont impliqués dans divers changements structurels dynamiques, tels que le glissement des nucléosomes, le déballage de l’ADN et d’autres interactions ADN-histone. Les structures terminales, ou queues, des histones sont particulièrement importantes dans ces processus.
Les queues d'histone subissent des modifications chimiques, modifiant la fonctionnalité de l'histone selon les besoins. Des études détaillées, et en particulier des visualisations, de la dynamique des nucléosomes sont cruciales pour mieux comprendre le rôle des queues d'histone.
Mikihiro Shibata de l'Université de Kanazawa et ses collègues ont réussi à réaliser des enregistrements vidéo de nucléosomes sans queue, prouvant que l'absence de queues d'histone augmente considérablement l'activité dynamique d'un nucléosome. L'étude est publiée dans Nano Letters .
Les scientifiques ont utilisé la microscopie à force atomique à grande vitesse (HS-AFM), un puissant outil de nanoimagerie permettant de visualiser les structures moléculaires et leur dynamique à haute résolution spatiale et temporelle.
Pour cela, les nucléosomes devaient être déposés sur un substrat. Shibata et ses collègues ont utilisé un film de ce qu'on appelle des piliers[5]arènes (molécules à structure tubulaire pentagonale) comme substrat, formant une surface idéale car les nucléosomes y sont facilement adsorbés sans que les processus dynamiques soient supprimés.
Les chercheurs ont d’abord examiné les nucléosomes pour lesquels les huit histones manquaient de queue. Sur la base de leurs observations HS-AFM, ils ont conclu que le glissement des nucléosomes et le déballage/réemballage de l'ADN se produisaient plus souvent que pour les nucléosomes normaux (canoniques). Cela suggère que sans queues, l'interaction histone-ADN est affaiblie, conduisant à une situation dans laquelle l'ADN peut se détacher plus facilement des histones.
Pour mieux comprendre les rôles de queues d'histone spécifiques, Shibata et ses collègues ont préparé des nucléosomes dans lesquels un type d'histone était sans queue. Il existe quatre types différents d'histones, appelés H2A, H2B, H3 et H4. Les expériences HS-AFM sur les nucléosomes ont révélé que les nucléosomes sans queue H2B et H3 présentaient une fréquence de dynamique accrue. À l'inverse, cela signifie que les histones canoniques H2B et H3 sont essentielles à la stabilité des nucléosomes.
Les scientifiques soulignent qu’ils n’ont pu observer aucun mouvement réel des queues d’histone – il est fort probable que la résolution temporelle de l’étude, 0,3 seconde, était beaucoup plus lente que la vitesse de la dynamique d’enroulement/déballage des queues. Malgré cette limitation, les travaux de Shibata et ses collègues prouvent clairement que les queues des histones H2B et H3 sont les principaux contributeurs à la dynamique des nucléosomes.
Concernant les travaux futurs, citant les chercheurs, "une technique de marquage des extrémités des queues d'histone pourrait permettre au HS-AFM de capturer les mouvements des queues d'histone elles-mêmes."
Microscopie à force atomique à grande vitesse
Le principe général de la microscopie à force atomique (AFM) est de faire scanner par une très petite pointe la surface d'un échantillon. Au cours de ce balayage horizontal (xy), la pointe, qui est attachée à un petit porte-à-faux, suit le profil vertical (z) de l'échantillon, induisant une force sur le porte-à-faux qui peut être mesurée.
L'ampleur de la force à la position xy peut être liée à la valeur z ; les données xyz générées lors d'une analyse aboutissent ensuite à une carte de hauteur fournissant des informations structurelles sur l'échantillon étudié. Dans l'AFM à grande vitesse (HS-AFM), le principe de fonctionnement est légèrement plus complexe :le cantilever est amené à osciller près de sa fréquence de résonance.
Lorsque la pointe est déplacée autour d'une surface, les variations de l'amplitude (ou de la fréquence) de l'oscillation du cantilever, résultant de l'interaction de la pointe avec la surface de l'échantillon, sont enregistrées, car elles fournissent une mesure de la valeur z locale. L'AFM n'implique pas de lentilles, sa résolution n'est donc pas limitée par ce que l'on appelle la limite de diffraction comme dans la diffraction des rayons X, par exemple.
HS-AFM donne une vidéo, où l'intervalle de temps entre les images dépend de la vitesse à laquelle une seule image peut être générée (en balayant l'échantillon par xy).
Des chercheurs du Nano Life Science Institute (WPI-NanoLSI) de l’Université de Kanazawa ont développé ces dernières années le HS-AFM, afin qu’il puisse être appliqué à l’étude de molécules biochimiques et de processus biomoléculaires en temps réel. Mikihiro Shibata et ses collègues ont maintenant appliqué la méthode pour étudier en détail la dynamique des nucléosomes, et en particulier le rôle des terminaisons moléculaires des histones, des protéines qui jouent un rôle crucial dans l'accessibilité de l'ADN.
Plus d'informations : Shin Morioka et al, La microscopie à force atomique à grande vitesse révèle le glissement des nucléosomes et la dynamique de déballage/emballage de l'ADN des nucléosomes sans queue, Nano Letters (2024). DOI :10.1021/acs.nanolett.4c00801
Informations sur le journal : Lettres nano
Fourni par l'Université de Kanazawa