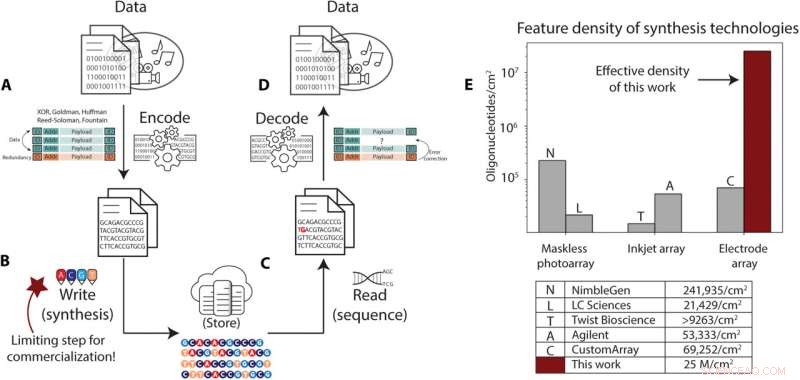
Le stockage des données ADN nécessite un débit de synthèse plus élevé que ce qui est possible avec les techniques actuelles. (A à D) Aperçu du pipeline de stockage des données ADN. (A) Les données numériques sont codées à partir de leur représentation binaire dans des séquences de bases d'ADN, avec un identifiant qui les corrèle avec un objet de données, des informations d'adressage utilisées pour réorganiser les données lors de la lecture et des informations redondantes utilisées pour la correction d'erreurs. (B) Ces séquences sont synthétisées en oligonucléotides d'ADN et stockées. (C) Au moment de la récupération, les molécules d'ADN sont sélectionnées et copiées par PCR ou d'autres méthodes et séquencées dans des représentations électroniques des bases de ces séquences. (D) Le processus de décodage prend cet ensemble bruyant et parfois incomplet de lectures de séquençage, corrige les erreurs et les séquences manquantes, et décode les informations pour récupérer les données. (E) Résumé des procédés de synthèse commerciaux et des densités d'oligonucléotides estimées correspondantes, telles que rapportées dans la littérature ou par les entreprises elles-mêmes. La densité de notre méthode électrochimique est surlignée en rouge foncé. Crédit :Progrès scientifiques , 10.1126/sciadv.abi6714
Les généticiens peuvent stocker des données dans de l'ADN synthétique comme support de stockage à long terme en raison de sa densité, de sa facilité de copie, de sa longévité et de sa durabilité. La recherche dans le domaine avait récemment avancé avec de nouveaux algorithmes d'encodage, d'automatisation, de préservation et de séquençage. Néanmoins, l'obstacle le plus difficile dans le déploiement du stockage ADN reste le débit d'écriture, qui peut limiter la capacité de stockage des données. Dans un nouveau rapport, Bichlien H. Nguyen et une équipe de scientifiques de Microsoft Research et d'informatique et d'ingénierie de l'Université de Washington, Seattle, États-Unis, ont développé le premier graveur de stockage d'ADN à l'échelle nanométrique. L'équipe avait l'intention de faire passer la densité d'écriture de l'ADN à 25 x 10 6 séquences par centimètre carré, une capacité de stockage améliorée par rapport aux puces de synthèse d'ADN existantes. Les scientifiques ont écrit et décodé avec succès un message dans l'ADN pour établir un système pratique de stockage des données ADN. Les résultats sont maintenant publiés dans Science Advances .
Archives ADN à long terme
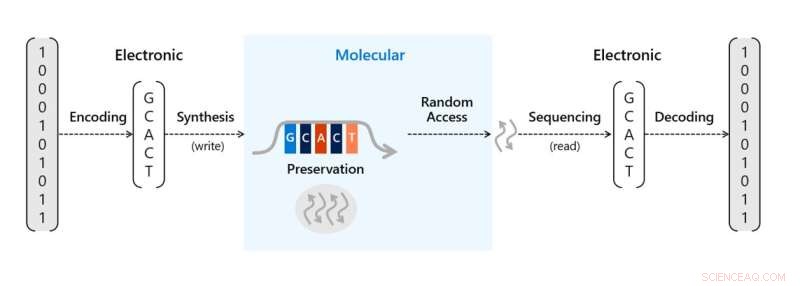
Le rythme actuel de génération de données dépasse les capacités de stockage existantes, l'ADN est une solution prometteuse à ce problème à une densité pratique attendue de plus de 60 pétaoctets par centimètre cube. Le matériau est durable dans une gamme de conditions, pertinent et facile à copier, avec la promesse d'être plus durable ou plus écologique que les supports commerciaux. Au cours du processus, des données numériques sous forme de séquences de bits peuvent être codées dans des séquences des quatre bases naturelles de l'ADN - guanine, adénine, thiamine et cytosine, bien que des bases supplémentaires soient également possibles. L'équipe peut ensuite écrire les séquences sous forme moléculaire via une synthèse d'oligonucléotides d'ADN de novo pour créer des molécules spécifiques basées sur un ensemble d'étapes chimiques répétitives. Les oligonucléotides résultants peuvent être conservés et stockés après synthèse. Pour accéder aux données, le stockage d'ADN peut être amplifié à l'aide de réactions en chaîne par polymérase et séquencé pour renvoyer les séquences de bases d'ADN dans le domaine numérique, puis les séquences de bases d'ADN peuvent être décodées pour récupérer la séquence de bits d'origine.
Vue d'ensemble d'un réseau de 650 nm avec un pas de 2 μm. (A) L'analyse par éléments finis de la génération et de la diffusion d'acide anodique à une électrode de 650 nm de diamètre avec un puits de 200 nm est représentée avec une vue en coupe le long du plan y =x et (B) vue de haut en bas sur le z =0 plan. Les couleurs bleu et jaune représentent des régions avec des concentrations d'acide relativement faibles et élevées, respectivement. (C) Un aperçu du réseau de synthèse d'ADN à l'échelle nanométrique avec des images de microscopie électronique à balayage du réseau d'électrodes de 650 nm et une vue agrandie d'une électrode. (D) Une image fluorescente dans laquelle le puits entourant chaque anode activée est modelé avec AAA-fluorescéine. Le diagramme de bande dessinée montre quelles électrodes de la disposition ont été activées. (E) Illustration des puits à motifs avec AAA-fluorescéine et AAA-AquaPhluor et (F) superposition d'image correspondante des deux fluorophores à la fin de l'ADN synthétisé sur le même réseau d'électrodes de 650 nm. Crédit :Progrès scientifiques , 10.1126/sciadv.abi6714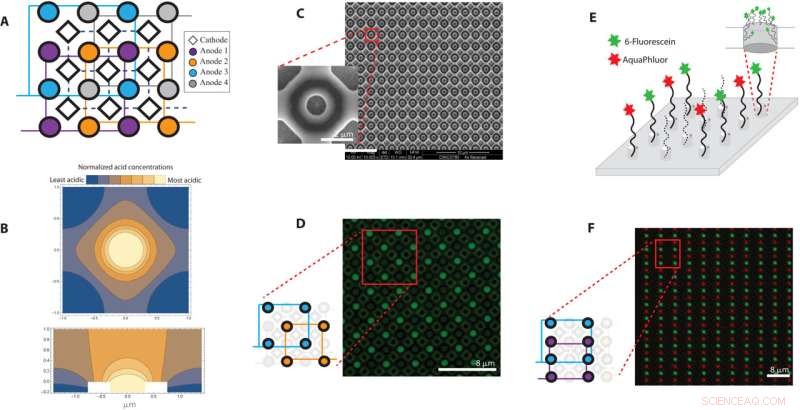
Dans cette étude, Nguyen et al. a produit un réseau d'électrodes qui a démontré un contrôle indépendant spécifique à l'électrode de la synthèse d'ADN avec des tailles et des pas d'électrode pour établir une densité de synthèse de 25 millions d'oligonucléotides par cm 2 . Cette valeur est estimée comme la densité d'électrodes requise pour atteindre l'objectif minimum de kilo-octets par seconde de stockage de données dans l'ADN. L'équipe a poussé l'état de l'art en matière de contrôle électronique-chimique et a fourni des preuves expérimentales de la bande passante d'écriture nécessaire au stockage des données ADN.
L'équipe a introduit un contrôleur moléculaire de preuve de concept sous la forme d'un minuscule mécanisme d'écriture de stockage d'ADN sur une puce. La puce pourrait emballer étroitement la synthèse d'ADN à 3 ordres de grandeur plus élevés qu'auparavant pour atteindre un plus grand débit d'écriture d'ADN. Pour stocker des informations dans l'ADN à l'échelle nécessaire à une utilisation commerciale, il a fallu deux processus cruciaux. L'équipe a d'abord dû traduire des bits numériques (uns et zéros) en brins d'ADN synthétique représentant des bits avec un logiciel d'encodage et un synthétiseur d'ADN. Ensuite, ils doivent être capables de lire et de décoder les informations jusqu'à leurs bits pour récupérer ces informations sous forme numérique à nouveau avec un séquenceur d'ADN et un logiciel de décodage.
Développement de réseaux électrochimiques pour des fonctionnalités à l'échelle nanométrique
Au cours de la synthèse traditionnelle des chaînes d'ADN, les scientifiques utilisent une méthode en plusieurs étapes connue sous le nom de chimie des phosphoramidites, dans laquelle une chaîne d'ADN peut être développée séquentiellement par l'ajout de bases d'ADN. Chaque base d'ADN contient un groupe de blocage pour empêcher les ajouts multiples de bases d'ADN à la chaîne en croissance. Lors de la fixation à une chaîne d'ADN, l'acide peut être délivré dans la configuration pour cliver le groupe de blocage et amorcer la chaîne d'ADN pour ajouter la base suivante. Lors de la synthèse électrochimique de l'ADN, chaque point du réseau contient une électrode et lorsqu'une tension est appliquée, de l'acide est généré à l'électrode de travail (anode) pour débloquer les chaînes d'ADN en croissance, tandis qu'une base équivalente est générée à la contre-électrode (cathode) . L'équipe a empêché la diffusion d'acide dans la configuration en concevant un réseau d'électrodes, où chaque électrode de travail autour de laquelle la formation d'acide s'est produite pendant la synthèse de l'ADN était enfoncée dans un puits et entourée de quatre contre-électrodes communes, c'est-à-dire des cathodes qui entraînaient la formation de base, pour confiner l'acide à des régions spécifiques. Nguyen et al. vérifié l'efficacité de la conception à l'aide d'une analyse par éléments finis. Au cours des expériences, lorsqu'il est présenté à une concentration suffisante, l'acide débloque les nucléotides liés à la surface pour permettre au nucléotide suivant de se coupler. En utilisant la configuration de puces contenant des points caractéristiques pour confiner les acides, ils ont développé des réseaux électrochimiques avec quatre électrodes individuelles pour réguler la synthèse d'ADN. L'équipe a ensuite réalisé des expériences avec deux bases marquées par fluorescence en vert et rouge. Comme preuve de concept, ils ont montré la capacité de l'appareil à écrire des données en synthétisant quatre brins d'ADN uniques, chacun long de 100 bases avec un message codé, sans erreur.
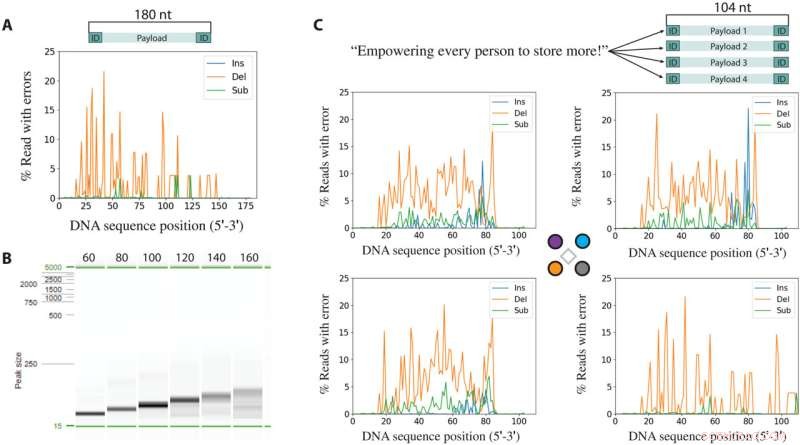
Erreurs issues de la synthèse suivie du séquençage. (A) Insertions (Ins), suppressions (Del) et substitutions (Sub) par position pour une séquence de 180 bases synthétisée et amplifiée par PCR. (B) Image d'électrophorèse des produits de synthèse après amplification par PCR. (C) Message codé en 64 octets divisés en quatre séquences uniques de 104 bases (en haut). Insertions, délétions et substitutions par locus de chacune des quatre séquences dans la synthèse multiplex. Dans chaque graphique d'analyse d'erreurs, les 20 bases terminales aux deux extrémités 3' et 5' proviennent des amorces utilisées en PCR et ne sont pas représentatives des erreurs synthétisées. Crédit :Progrès scientifiques , 10.1126/sciadv.abi6714
Mise à l'échelle du stockage des données ADN avec des puits d'électrodes à l'échelle nanométrique. Minuscule mécanisme d'écriture de stockage d'ADN sur une puce. Crédit :Microsoft Research Blog, Avances scientifiques , 10.1126/sciadv.abi6714
Perspectives :Synthétiser des oligonucléotides courts sur le réseau d'électrodes pour le stockage de données
En utilisant la configuration, Nguyen et al. a également démontré la synthèse spatialement contrôlée d'oligonucléotides courts sur le réseau d'électrodes pour évaluer la longueur maximale d'ADN qui pourrait être formée. Les scientifiques ont créé une seule séquence d'ADN avec 180 nucléotides et des produits de différentes longueurs amplifiés par PCR à partir de la longueur complète des oligonucléotides. Au fur et à mesure que l'amplicon s'allongeait, les produits de PCR attendus semblaient plus faibles et moins bien définis, tandis que les amplicons plus courts montraient des bandes plus fortes et plus bien définies indiquant des erreurs de synthèse plus élevées. Sur la base des résultats, les chercheurs ont sélectionné une longueur de séquence allant jusqu'à 100 bases pour faciliter la purification afin de fournir une démonstration pratique du stockage des données ADN sans autre optimisation. De cette façon, la méthode de preuve de concept démontrée dans ce travail par Bichlien H. Nguyen et ses collègues a ouvert la voie à la génération de séquences d'ADN uniques et à grande échelle en parallèle pour le stockage des données. Les travaux ont dépassé les rapports précédents sur les séquences d'ADN synthétiques denses pour fournir une première indication expérimentale pour atteindre la bande passante d'écriture requise pour le stockage de données à l'échelle nanométrique. Les scientifiques s'attendent à des applications immédiates des appareils dans les technologies de l'information et prévoient leurs applications pratiques dans la science des matériaux, la biologie synthétique et les essais de biologie moléculaire à grande échelle.
© 2021 Réseau Science X La synthèse enzymatique d'ADN voit le jour