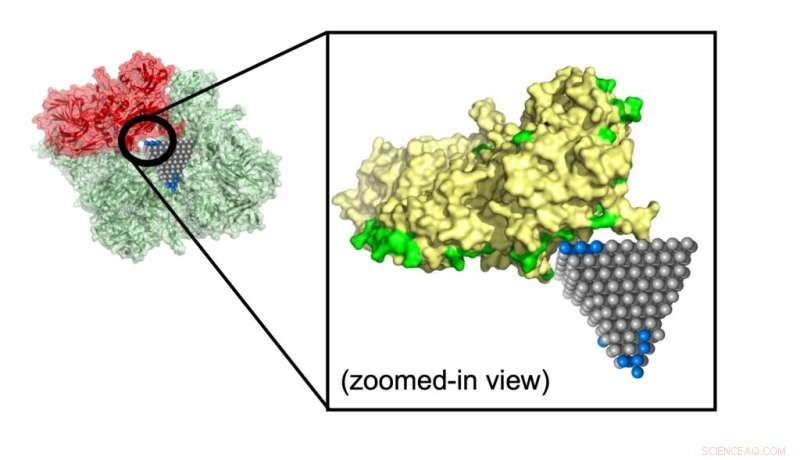
Le modèle a prédit comment une nanopyramide d'oxyde de zinc interrompt une protéine qui contribue au métabolisme du SARM (Staphylococcus aureus résistant à la méthicilline), une souche courante qui provoque des infections résistantes aux antibiotiques. Crédit :Minjeong Cha et Emine Sumeyra Turali Emre, Kotov Lab
Avec l'augmentation des infections résistantes aux antibiotiques et un virus pandémique en constante évolution, il est facile de comprendre pourquoi les chercheurs veulent pouvoir concevoir des nanoparticules artificielles capables de stopper ces infections.
Un nouveau modèle d'apprentissage automatique qui prédit les interactions entre les nanoparticules et les protéines, développé à l'Université du Michigan, nous rapproche un peu plus de cette réalité.
"Nous avons repensé les nanoparticules pour qu'elles soient plus que de simples véhicules d'administration de médicaments. Nous les considérons comme des médicaments actifs en eux-mêmes", a déclaré J. Scott VanEpps, professeur adjoint de médecine d'urgence et auteur de l'étude dans Nature Computational Sciences .
La découverte de médicaments est un processus lent et imprévisible, c'est pourquoi tant d'antibiotiques sont des variantes d'un médicament précédent. Les développeurs de médicaments aimeraient concevoir des médicaments capables d'attaquer les bactéries et les virus de la manière qu'ils choisissent, en tirant parti des mécanismes "serrure et clé" qui dominent les interactions entre les molécules biologiques. Mais il n'était pas clair comment passer de l'idée abstraite d'utiliser des nanoparticules pour perturber les infections à la mise en œuvre pratique du concept.
"En appliquant des méthodes mathématiques aux interactions protéine-protéine, nous avons simplifié la conception de nanoparticules qui imitent l'une des protéines de ces paires", a déclaré Nicholas Kotov, professeur émérite Irving Langmuir de sciences chimiques et d'ingénierie et auteur correspondant de l'étude. .
"Les nanoparticules sont plus stables que les biomolécules et peuvent conduire à des classes entièrement nouvelles d'agents antibactériens et antiviraux."
Le nouvel algorithme d'apprentissage automatique compare les nanoparticules aux protéines en utilisant trois façons différentes de les décrire. Alors que la première était une description chimique conventionnelle, les deux qui concernaient la structure se sont avérées les plus importantes pour prédire si une nanoparticule serait une correspondance parfaite avec une protéine spécifique.
Entre elles, ces deux descriptions structurelles ont capturé la surface complexe de la protéine et comment elle pourrait se reconfigurer pour permettre des ajustements de verrouillage et de clé. Cela inclut les poches dans lesquelles une nanoparticule pourrait s'insérer, ainsi que la taille qu'une telle nanoparticule devrait avoir. Les descriptions incluaient également la chiralité, une torsion dans le sens des aiguilles d'une montre ou dans le sens inverse des aiguilles d'une montre qui est importante pour prédire comment une protéine et une nanoparticule vont se verrouiller.
"Il existe de nombreuses protéines à l'extérieur et à l'intérieur des bactéries que nous pouvons cibler. Nous pouvons utiliser ce modèle comme premier criblage pour découvrir quelles nanoparticules se lieront à quelles protéines", a déclaré Emine Sumeyra Turali Emre, chercheuse postdoctorale en génie chimique et co-première auteur de l'article, avec Minjeong Cha, un Ph.D. étudiant en science et génie des matériaux.
Emre et Cha ont expliqué que les chercheurs pourraient suivre les correspondances identifiées par leur algorithme avec des simulations et des expériences plus détaillées. Une telle correspondance pourrait arrêter la propagation du SARM, une souche courante résistante aux antibiotiques, en utilisant des nanopyramides d'oxyde de zinc qui bloquent les enzymes métaboliques de la bactérie.
"Les algorithmes d'apprentissage automatique comme le nôtre fourniront un outil de conception pour les nanoparticules qui peuvent être utilisées dans de nombreux processus biologiques. L'inhibition du virus qui cause le COVID-19 en est un bon exemple", a déclaré Cha. "Nous pouvons utiliser cet algorithme pour concevoir efficacement des nanoparticules qui ont une activité antivirale à large spectre contre toutes les variantes."
Cette percée a été rendue possible par la Blue Sky Initiative de l'U-M College of Engineering, qui a fourni un soutien à l'équipe interdisciplinaire qui a mené l'exploration fondamentale de l'efficacité d'une approche d'apprentissage automatique lorsque les données sur l'activité biologique des nanoparticules sont si rares.
"Le cœur de l'idée Blue Sky est exactement ce que ce travail couvre :trouver un moyen de représenter les protéines et les nanoparticules dans une approche unifiée pour comprendre et concevoir de nouvelles classes de médicaments qui ont de multiples façons d'agir contre les bactéries", a déclaré Angela Violi, une Professeur Arthur F. Thurnau, professeur de génie mécanique et chef du projet nanobiotique Blue Sky.
Des collaborateurs de l'Université de Californie à Los Angeles ont également contribué à l'algorithme d'apprentissage automatique. Un nouvel outil permet une modélisation sans précédent des nanoparticules magnétiques