
Analyse des déformations élastiques Crédit :POSTECH
C'est des millions de milliards de fois plus lumineux que la lumière du soleil et un énorme 1, 000 trillionième de seconde, appelée à juste titre « lumière instantanée », la lumière du laser à électrons libres à rayons X (XFEL) qui ouvre un nouveau paradigme scientifique. En le combinant avec l'IA, une équipe de recherche internationale a réussi à filmer et à restaurer la structure 3-D de nanoparticules qui partagent des similitudes structurelles avec les virus. Avec la crainte d'une nouvelle pandémie croissante dans le monde en raison de COVID-19, cette découverte attire l'attention des milieux universitaires pour l'imagerie de la structure du virus avec à la fois une précision et une vitesse élevées.
Une équipe internationale de chercheurs de POSTECH, Université nationale de Singapour (NUS), KAIST, ESSENTIEL, et IBS ont analysé avec succès les hétérogénéités structurelles dans les structures 3D de nanoparticules en irradiant des milliers de nanoparticules par heure à l'aide du XFEL au Pohang Accelerator Laboratory (PAL) en Corée et en restaurant des multi-modèles 3D grâce à l'apprentissage automatique. L'équipe de recherche dirigée par le professeur Changyong Song et Ph.D. Le candidat Do Hyung Cho du Département de physique de POSTECH a conduit la collaboration internationale de recherche pour le réaliser.
Les nanoparticules ont une fonction particulière qui peut ne pas être disponible à partir de matériaux en vrac natifs, et on peut contrôler leurs propriétés physiques et chimiques en concevant des structures 3D et des compositions d'éléments constitutifs.
Le point commun entre les nanoparticules et les virus est qu'ils existent sous forme de particules indépendantes, plutôt qu'en cristal-régulier, arrangements périodiques, et, En tant que tel, leurs structures ne sont pas uniformes au niveau du nanomètre. Pour comprendre précisément leurs structures, il est nécessaire d'analyser statistiquement la structure des particules individuelles en utilisant l'ensemble de la distribution des structures allant de milliers à des centaines de milliers de spécimens. Cependant, les microscopes électroniques ne parviennent souvent pas à fournir une pénétration suffisante pour limiter la taille de l'échantillon à sonder; les rayons X conventionnels peuvent endommager l'échantillon par le rayonnement X lui-même, rendant difficile l'obtention d'une résolution suffisante.

PAL-XFEL au laboratoire de l'accélérateur de Pohang Crédit :POSTECH
L'équipe de recherche a surmonté les limitations pratiques de la méthode conventionnelle en utilisant le laser à électrons libres à rayons X et la méthode d'apprentissage automatique pour observer la distribution statistique de la structure 3-D de milliers de nanoparticules au niveau nanométrique. Par conséquent, Des structures 3-D de nanoparticules ayant une taille de 300 nm ont été obtenues avec une résolution meilleure que 20 nm.
Cette réalisation a été particulièrement importante pour restaurer la structure 3D de milliers de nanoparticules à l'aide de l'apprentissage automatique. Étant donné que les techniques conventionnelles d'imagerie à particule unique supposent souvent une structure 3D identique des spécimens, il était difficile de restituer la structure dans des données expérimentales réelles où la structure de l'échantillon n'est pas homogène. Cependant, avec l'introduction du multi-modèle cette fois, les chercheurs ont réussi à restaurer les structures 3D représentatives. Ces recherches ont permis de classer les nanoparticules en quatre formes principales, et a confirmé qu'environ 40 % d'entre eux avaient des structures similaires.
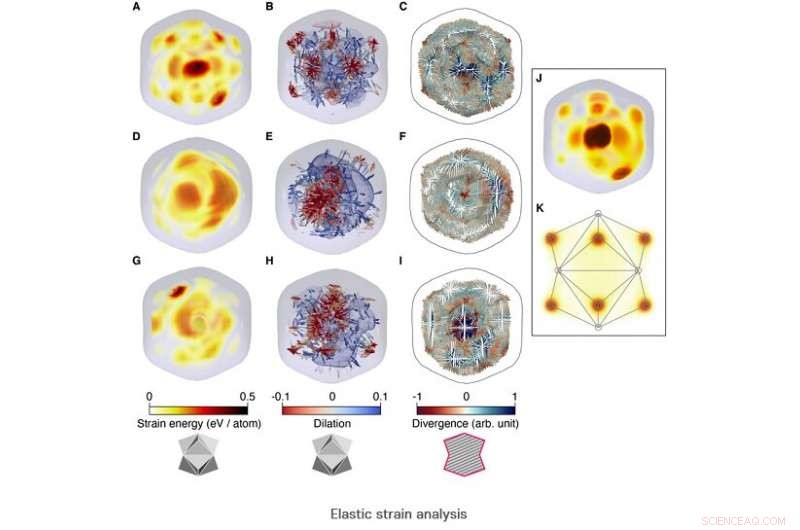
Crédit :Université des sciences et technologies de Pohang (POSTECH)
En outre, grâce à l'analyse quantitative de la structure 3D restaurée, l'équipe de collaboration de recherche internationale a également découvert la distribution des contraintes élastiques internes accompagnée de la structure polyédrique caractéristique des nanoparticules et de la distribution de densité inhomogène.
"Ces résultats permettent l'observation de la structure 3-D d'échantillons viraux non cristallins avec des molécules internes réparties de manière non homogène, " a expliqué le professeur Changyong Song de POSTECH. " L'ajout de l'algorithme de restauration d'images 3D à cela grâce à l'apprentissage automatique semble prometteur pour les études de structures de macromolécules ou de virus dans les organismes vivants. "