
Un résumé graphique de l'axe intestin-cerveau (à gauche) et de la plate-forme in vitro imprimée en 3D (à droite). Crédit :Université du Maryland
Quiconque a déjà rencontré des "papillons dans l'estomac" avant de faire une grande présentation ne sera pas surpris d'apprendre qu'il existe un lien physique réel entre son intestin et son cerveau. Les neuroscientifiques et les professionnels de la santé appellent cela "l'axe intestin-cerveau" (GBA). Une meilleure compréhension de l'ACS pourrait conduire à des traitements et à des remèdes pour les troubles de l'humeur neurologiques comme la dépression et l'anxiété, ainsi que pour une gamme de maladies inflammatoires auto-immunes chroniques comme le syndrome du côlon irritable (SCI) et la polyarthrite rhumatoïde (PR).
Le problème est que jusqu'à présent, les "papillons" ont été tous les médecins avec lesquels les médecins ont dû travailler pour détecter ces troubles liés à l'ACS. Même aujourd'hui, ces maladies sont principalement diagnostiquées par les propres rapports des patients sur leurs symptômes. Trouver des « biomarqueurs » – des mesures objectives d'une substance dans le corps qui indique la présence d'un trouble – pourrait considérablement améliorer le diagnostic et alléger le fardeau des patients pour identifier correctement leurs propres symptômes.
Les scientifiques soupçonnent que la sérotonine, un neurotransmetteur chimique, est le biomarqueur d'une gamme de troubles de l'ACS. La sérotonine stimule le système nerveux via le nerf vague, le connecteur physique entre le cerveau et le côlon. Généré profondément dans la muqueuse de l'intestin, la sérotonine influence finalement tout, de l'humeur et des émotions au sommeil, la digestion et la sécrétion d'hormones. Sa production est en quelque sorte affectée par le « microbiome » bactérien présent dans cet environnement. Les chercheurs espèrent que la création d'outils pour analyser la production et le dysfonctionnement de la sérotonine dans le microbiome intestinal aidera à percer les mystères des troubles liés à la GBA.
Avec 1 million de dollars de financement de la National Science Foundation, Ingénieurs de l'Université du Maryland (UMD), neuroscientifiques, les microbiologistes et les physiciens ont fait des progrès significatifs dans le développement d'une plate-forme qui peut surveiller et modéliser le traitement en temps réel de l'activité de la sérotonine du microbiome intestinal. Leur objectif est de conditionner un jour la plateforme dans une capsule ingérable capable de détecter, le traitement et le suivi des maladies GBA.
La convergence des disciplines est essentielle, dit le professeur Reza Ghodssi, le chercheur principal du projet. "Nous faisons converger les neurosciences, signalisation moléculaire, et les dispositifs et systèmes micro-nano. Cela nous permet de mesurer et d'étudier les données à l'interface de chaque jonction d'une plate-forme GBA simulée - cellule à cellule, cellule à molécule, molécule au nerf et développer des méthodologies d'ingénierie pour l'analyser et l'interpréter.
Le travail s'appuie sur l'expertise des dispositifs médicaux ingérables qui a été développée dans le laboratoire UMD MEMS Sensors and Actuators, le département de bio-ingénierie Fischell, et l'Initiative Cerveau et Comportement.
Trois nouveaux articles publiés détaillent les progrès réalisés dans la détection de la sérotonine, évaluer ses effets neurologiques, et détecter des changements infimes dans l'épithélium intestinal.
Dans "Mesure électrochimique de la sérotonine par des électrodes Au-CNT fabriquées sur des membranes de culture cellulaire poreuses, " l'équipe a développé une plateforme qui donne accès au site spécifique de production de sérotonine, important parce que la sérotonine est sécrétée par le fond des cellules. Une membrane poreuse innovante avec un capteur de sérotonine intégré sur lequel un modèle de la muqueuse intestinale peut être cultivé a permis aux chercheurs d'accéder à la fois aux côtés supérieur et inférieur de la culture cellulaire.
Le document a été publié en ligne le 7 septembre. 2020 dans le La nature journal, Microsystèmes et nano-ingénierie. Les auteurs sont des docteurs en bio-ingénierie. étudiante Ashley Chapin, ancien chercheur postdoctoral ISR Pradeep Ramiah Rajasekaran, ancien élève David N. Quan (Ph.D. BioE 2015), Professeur Liangbing Hu (MSE/MEII), Professeur agrégé Jens Herberholz (Psychologie/NACS), Professeur William Bentley (BioE/Fischell Institute/IBBR), et le professeur Reza Ghodssi (ECE/ISR).
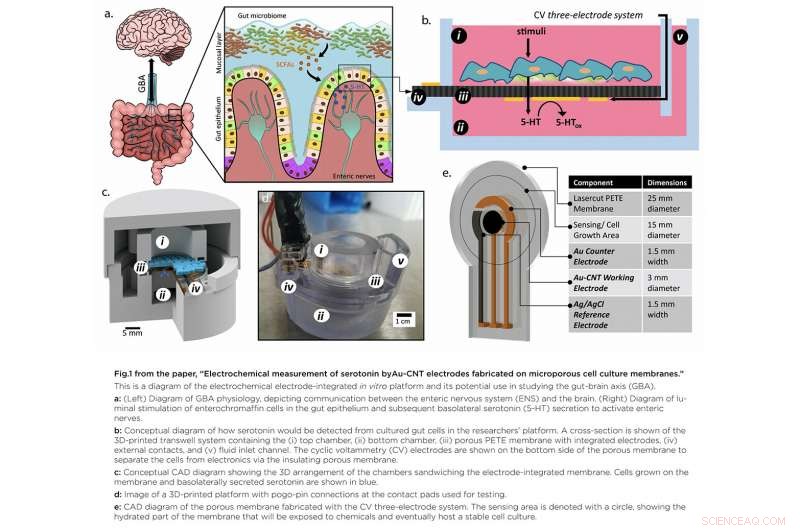
Crédit :Université du Maryland
En utilisant le dépôt de métal, ils ont modelé un système à trois électrodes directement sur une membrane de culture cellulaire poreuse suspendue dans un boîtier personnalisé imprimé en 3D. Les cellules peuvent être cultivées sur le dessus de la membrane avec le capteur de sérotonine orienté vers le bas pour une détection directe. L'équipe a ensuite amélioré la sensibilité de la détection de la sérotonine en augmentant la surface efficace de l'électrode, goutter un petit volume de nanotubes de carbone sur la surface de l'électrode. Les solutions préparées de sérotonine étaient détectables bien dans la plage de concentration physiologique attendue.
Ce travail est le premier à démontrer une méthode réalisable pour détecter des molécules redox, telles que la sérotonine, directement sur un substrat de culture cellulaire poreux et flexible. Il offre un accès supérieur aux molécules libérées par les cellules et crée un environnement intestinal modèle contrôlable sans recourir à des procédures invasives sur les humains ou les animaux.
Le deuxième article de l'équipe, "Un système de biosurveillance hybride pour la communication intestin-neurone, " s'appuie sur les résultats de la première :les chercheurs ont développé davantage la plate-forme de mesure de la sérotonine afin qu'elle puisse évaluer les effets neurologiques de la sérotonine. En ajoutant et en intégrant un modèle de nerf d'écrevisse disséqué avec le modèle de muqueuse intestinale, l'équipe a créé une interface intestin-neurone qui peut évaluer électrophysiologiquement la réponse nerveuse à la sérotonine détectée électrochimiquement. Cette avancée permet l'étude de la signalisation moléculaire entre l'intestin et les cellules nerveuses, rendant possible la surveillance en temps réel des deux tissus GBA pour la première fois.
L'article a été publié en ligne en juin 2020 Journal IEEE des systèmes microélectromécaniques . Il a été écrit par Chapin, Doctorat en génie électrique et informatique étudiant Jinjing Han, Doctorat en neurosciences et sciences cognitives étudiant Ta-Wen Ho, Herberholz et Ghodssi.
Finalement, le concept, design and use for the entire biomonitoring platform is described in a third paper, "3-D Printed Electrochemical Sensor Integrated Transwell Systems, " published online Oct. 5, 2020 in the La nature journal Microsystems and Nanoengineering . The paper was written by Rajasekaran, Chapin, Quan, Herberholz, Bentley and Ghodssi.
This paper delves into the development of the 3-D-printed housing, the maintenance of a healthy lab-on-a-chip gut cell culture, and the evaluation of the two types of sensors integrated on the cell culture membrane. The dual sensors are particularly important because they provide feedback about multiple components of the system—namely, the portions that model the gut lining's permeability (a strong indicator of disease) and its serotonin release (a measure of communication with the nervous system). Alongside the electrochemical sensor—evaluated using a standard redox molecule ferrocene dimethanol—an impedance sensor was used to monitor cell growth and coverage over the membrane. Using both these sensors would allow monitoring of a gut cell culture under various environmental and dietary conditions. It also would enable researchers to evaluate changes to barrier permeability (a strong indicator of disease), and serotonin release (a measure of communication with the nervous system).
"These works represent a big step forward in our understanding of the gut/brain axis, " says Cornell University's John March, Chair of the Department of Biological and Environmental Engineering. "One of the limitations of this field is the inability to perform highly controlled experiments in a 'close to in vivo' system. These papers provide ways around this problem with simple, elegant experiments that are highly accessible. I expect these will be used frequently."
Because the engineering aspects of the platform are well underway, the researchers are working towards culturing multi-tissue interfaces with the help of Jay Pasricha and Subhash Kulkarni at Johns Hopkins University. Eventually multiple platforms will be created, each colonized with a different combination of gut bacteria, to measure the neurophysiological effects of serotonin production in varying microbiome environments.
Avec ces informations, Professor Wolfgang Losert (Physics/IPST/IREAP) will lead a machine learning effort to process the sensor data through a computer model that can simulate the outcomes of the different microbiomes. This will provide the clearest picture yet of how a system as complex and individually unique as the gut microbiome affects both gut and brain health. It also may help researchers better understand the connection between nutrition and mental health.
"Understanding biology at the level of whole organisms is a frontier in biology, and essential to forming a basis for precision medicine, " says the University of California, Berkeley's Amy Herr, the John D. &Catherine T. MacArthur Professor of Bioengineering. "By harnessing hallmarks of engineering—integrated, systems-level design—the new research from the Ghodssi-Bentley-Herberholz team presents an integrated approach to elegantly perturb and then probe the electrons and molecules that are key conduits of information flow in whole organisms."