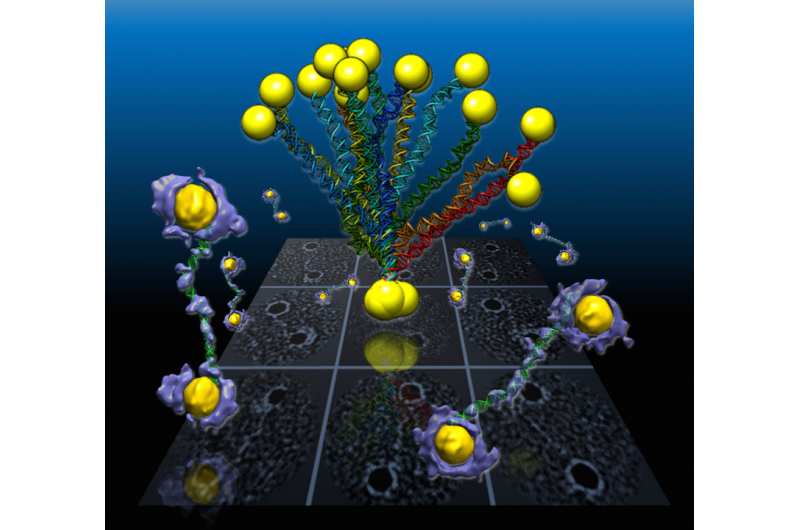
Dans une étude menée par le Berkeley Lab, Des segments d'ADN flexibles à double hélice connectés à des nanoparticules d'or sont révélés à partir des cartes de densité 3D (violet et jaune) reconstruites à partir d'échantillons individuels à l'aide d'une technique développée par Berkeley Lab appelée tomographie électronique à particules individuelles ou IPET. Les projections des structures sont affichées dans la grille d'arrière-plan. Crédit :Berkeley Lab
Une équipe internationale travaillant au Lawrence Berkeley National Laboratory (Berkeley Lab) du ministère de l'Énergie a capturé les premières images 3D haute résolution à partir de segments d'ADN individuels à double hélice attachés à chaque extrémité à des nanoparticules d'or. Les images détaillent la structure flexible des segments d'ADN, qui apparaissent comme des cordes à sauter à l'échelle nanométrique.
Cette capacité d'imagerie unique, mis au point par les scientifiques du Berkeley Lab, pourrait aider à l'utilisation de segments d'ADN comme éléments constitutifs de dispositifs moléculaires qui fonctionnent comme des systèmes d'administration de médicaments à l'échelle nanométrique, marqueurs pour la recherche biologique, et composants pour mémoire d'ordinateur et dispositifs électroniques. Cela pourrait également conduire à des images de protéines importantes liées à la maladie qui se sont révélées insaisissables pour d'autres techniques d'imagerie, et du processus d'assemblage qui forme l'ADN à partir de brins individuels.
Les formes des brins d'ADN enroulés, qui ont été pris en sandwich entre des nanoparticules d'or en forme de polygone, ont été reconstruits en 3-D à l'aide d'une technique de microscopie électronique de pointe couplée à un processus de coloration des protéines et à un logiciel sophistiqué qui a fourni des détails structurels à l'échelle d'environ 2 nanomètres, ou deux milliardièmes de mètre.
"Nous n'avions aucune idée de ce à quoi ressemblerait l'ADN double brin entre les particules de nano-or, " dit Gang "Gary" Ren, un scientifique de Berkeley Lab qui a dirigé la recherche. "C'est la première fois que l'on visualise directement un segment d'ADN double brin individuel en 3-D, ", a-t-il déclaré. Les résultats ont été publiés dans l'édition du 30 mars de Communication Nature .

Gang Ren (debout) et Lei Zhang ont participé à une étude à la fonderie moléculaire de Berkeley Lab qui a produit des reproductions en 3D d'échantillons individuels de segments d'ADN à double hélice attachés à des nanoparticules d'or. Crédit :Roy Kaltschmidt/Laboratoire de Berkeley
La méthode développée par cette équipe, appelée tomographie électronique à particules individuelles (IPET), avait précédemment capturé la structure 3-D d'une seule protéine qui joue un rôle clé dans le métabolisme du cholestérol humain. En saisissant des images 2D du même objet sous différents angles, la technique permet aux chercheurs d'assembler une image 3-D de cet objet. L'équipe a également utilisé la technique pour découvrir la fluctuation d'une autre protéine flexible bien connue, immunoglobuline humaine 1, qui joue un rôle dans notre système immunitaire.
Pour cette dernière étude des nanostructures d'ADN, Ren a utilisé une technique d'étude par faisceau d'électrons appelée cryomicroscopie électronique (cryo-EM) pour examiner des échantillons d'ADN-nanogold congelés, et utilisé IPET pour reconstruire des images 3D à partir d'échantillons colorés avec des sels de métaux lourds. L'équipe a également utilisé des outils de simulation moléculaire pour tester les variations de forme naturelles, appelé "conformations, " dans les échantillons, et comparé ces formes simulées avec des observations.
Ren a expliqué que la dynamique naturellement flexible des échantillons, comme un homme qui agite les bras, ne peut pas être entièrement détaillé par une méthode utilisant une moyenne de nombreuses observations.
Un moyen populaire de visualiser les détails structurels à l'échelle nanométrique d'échantillons biologiques délicats consiste à les transformer en cristaux et à les zapper avec des rayons X, bien que cela ne préserve pas leur forme naturelle et que les échantillons d'ADN-nanoor de cette étude soient incroyablement difficiles à cristalliser. D'autres techniques de recherche courantes peuvent nécessiter une collection de milliers d'objets quasi identiques, vu au microscope électronique, pour en compiler un seul, structure 3-D moyennée. Mais cette image 3D peut ne pas montrer de manière adéquate les fluctuations de forme naturelles d'un objet donné.