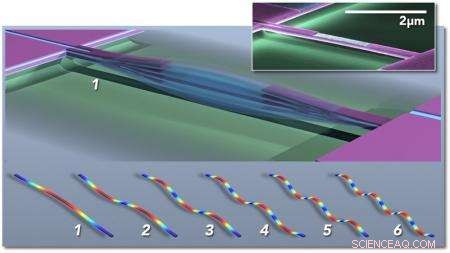
Capteur de masse basé sur des systèmes nanoélectromécaniques multimodes (NEMS) ; la figure principale représente schématiquement une poutre à double pince vibrant en mode fondamental (1). Des « instantanés » conceptuels des six premiers modes de vibration sont présentés ci-dessous (1-6), les couleurs indiquent une tension élevée (rouge) à faible (bleu). L'encart montre une micrographie électronique colorisée d'un résonateur piézoélectrique NEMS fabriqué au Kavli Nanoscience Institute de Caltech. Crédit :M. Matheny, L.G. Villanueva, P. Hung, J. Li et M. Roukes/Caltech
S'appuyant sur leur création du tout premier appareil mécanique capable de mesurer la masse de molécules individuelles, un à la fois, une équipe de scientifiques de Caltech et leurs collègues ont créé des nanodispositifs qui peuvent également révéler leur forme. De telles informations sont cruciales lorsque l'on essaie d'identifier de grosses molécules de protéines ou des assemblages complexes de molécules de protéines.
« Vous pouvez imaginer qu'avec de grands complexes protéiques fabriqués à partir de nombreux, petites sous-unités, il existe de nombreuses façons de les assembler. Ceux-ci peuvent finir par avoir des masses assez similaires tout en étant en réalité des espèces différentes avec des fonctions biologiques différentes. Cela est particulièrement vrai avec les enzymes, protéines qui interviennent dans les réactions chimiques dans le corps, et des protéines membranaires qui contrôlent les interactions d'une cellule avec son environnement, " explique Michael Roukes, le professeur de physique Robert M. Abbey, Physique appliquée, et Bioengineering at Caltech et co-auteur d'un article décrivant la technologie paru le 30 mars dans le numéro en ligne de la revue Nature Nanotechnologie .
L'un des fondements de la révolution génomique a été la capacité de répliquer en masse des molécules d'ADN ou d'ARN en utilisant la réaction en chaîne par polymérase pour créer les millions de copies nécessaires au séquençage et à l'analyse typiques. Cependant, la même technologie de production de masse ne fonctionne pas pour copier des protéines. À l'heure actuelle, si vous voulez bien identifier une protéine en particulier, vous en avez besoin de beaucoup, généralement des millions de copies de la seule protéine d'intérêt, avec très peu d'autres protéines étrangères comme contaminants. La masse moyenne de cette population moléculaire est ensuite évaluée avec une technique appelée spectrométrie de masse, dans lequel les molécules sont ionisées - de sorte qu'elles atteignent une charge électrique - puis autorisées à interagir avec un champ électromagnétique. En analysant cette interaction, les scientifiques peuvent en déduire le rapport masse moléculaire/charge.
Mais la spectrométrie de masse ne peut souvent pas discriminer des différences subtiles mais cruciales dans les molécules ayant des rapports masse/charge similaires. "Avec la spectrométrie de masse aujourd'hui, " explique Roukes, « les grosses molécules et les complexes moléculaires sont d'abord découpés en de nombreux petits morceaux, C'est, en fragments de molécules plus petits que les instruments existants peuvent traiter. Ces différents fragments sont analysés séparément, puis la bioinformatique, impliquant des simulations informatiques, est utilisée pour reconstituer le puzzle. Mais ce processus de réassemblage peut être contrecarré si des morceaux de différents complexes sont mélangés. »
Avec leurs appareils, Roukes et ses collègues peuvent mesurer la masse d'une molécule intacte individuelle. Chaque appareil, qui ne mesure que quelques millionièmes de mètre ou moins, se compose d'une structure vibrante appelée résonateur de système nanoélectromécanique (NEMS). Lorsqu'une particule ou une molécule atterrit sur le nanodispositif, la masse ajoutée modifie la fréquence à laquelle la structure vibre, un peu comme mettre des gouttes de soudure sur une corde de guitare changerait la fréquence de ses vibrations et le son résultant. Les décalages de fréquence induits renseignent sur la masse de la particule. Mais eux aussi, comme décrit dans le nouveau document, peut être utilisé pour déterminer la distribution spatiale tridimensionnelle de la masse :c'est-à-dire, la forme de la particule.
"Une corde de guitare ne vibre pas seulement à une fréquence, " dit Roukes. " Il y a des harmoniques de son ton fondamental, ou modes dits vibratoires. Ce qui distingue une corde de violon d'une corde de guitare, ce sont vraiment les différents mélanges de ces différentes harmoniques du ton fondamental. La même chose s'applique ici. Nous avons tout un tas de tonalités différentes qui peuvent être excitées simultanément sur chacun de nos nanodispositifs, et nous suivons de nombreux tons différents en temps réel. Il s'avère que lorsque la molécule atterrit dans des orientations différentes, ces harmoniques sont décalées différemment. Nous pouvons alors utiliser la théorie de l'imagerie inertielle que nous avons développée pour reconstruire une image dans l'espace de la forme de la molécule."
"La nouvelle technique révèle une capacité auparavant non réalisée de capteurs mécaniques, " déclare le professeur Mehmet Selim Hanay de l'université Bilkent d'Ankara, Turquie, un ancien chercheur postdoctoral au laboratoire Roukes et co-premier auteur de l'article. "Auparavant, nous avons identifié des molécules, comme l'anticorps IgM, basé uniquement sur leurs poids moléculaires. Maintenant, en permettant de déduire simultanément les informations de poids moléculaire et de forme pour une même molécule, la nouvelle technique peut grandement améliorer le processus d'identification, et cela est important à la fois pour la recherche fondamentale et l'industrie pharmaceutique. »
Actuellement, les structures moléculaires sont déchiffrées par cristallographie aux rayons X, une technique souvent laborieuse qui consiste à isoler, purification, puis cristalliser des molécules, puis en évaluant leur forme en fonction des schémas de diffraction produits lorsque les rayons X interagissent avec les atomes qui forment ensemble les cristaux. Cependant, de nombreuses molécules biologiques complexes sont difficiles, voire impossibles à cristalliser. Et, même quand ils peuvent être cristallisés, la structure moléculaire obtenue représente la molécule à l'état cristallin, qui peut être très différente de la structure de la molécule sous sa forme biologiquement active.
"Vous pouvez imaginer des situations où vous ne savez pas exactement ce que vous cherchez, où vous êtes en mode découverte, et vous essayez de comprendre la réponse immunitaire du corps à un agent pathogène particulier, par exemple, " dit Roukes. Dans ces cas, la capacité d'effectuer la détection d'une molécule unique et d'obtenir autant d'informations distinctes que possible sur cette molécule individuelle améliore considérablement les chances de faire une identification unique.
"Nous disons que le cancer commence souvent par une seule cellule aberrante, et ce que cela signifie, c'est que même s'il peut s'agir de l'une d'une multitude de cellules similaires, il y a quelque chose d'unique dans la composition moléculaire de cette cellule. Avec cette technique, nous avons potentiellement un nouvel outil pour comprendre ce qui est unique à ce sujet, " il ajoute.
Jusque là, la nouvelle technique a été validée en utilisant des particules de tailles et de formes connues, comme les nanogouttelettes de polymère. Roukes et ses collègues montrent qu'avec les nanodispositifs de pointe d'aujourd'hui, l'approche peut fournir une résolution à l'échelle moléculaire, c'est-à-dire fournir la possibilité de voir les sous-composants moléculaires de l'individu, assemblages de protéines intacts. Les efforts actuels du groupe se concentrent désormais sur de telles explorations.
Scott Kelber, un ancien étudiant diplômé du laboratoire Roukes, est l'autre co-premier auteur de l'article, intitulé « Imagerie inertielle avec des systèmes nanoélectromécaniques ». Professeur John Sader de l'Université de Melbourne, Australie, et un associé invité en physique à Caltech, est l'auteur co-correspondant. Les coauteurs supplémentaires sont Cathal D. O'Connell et Paul Mulvaney de l'Université de Melbourne. Le travail a été financé par un prix Pioneer du directeur des National Institutes of Health, une chaire de professeur invité distingué du Caltech Kavli Nanoscience Institute, la Fondation pour la Recherche et l'Enseignement Supérieur à Paris, et le programme de subventions du Conseil australien de la recherche.