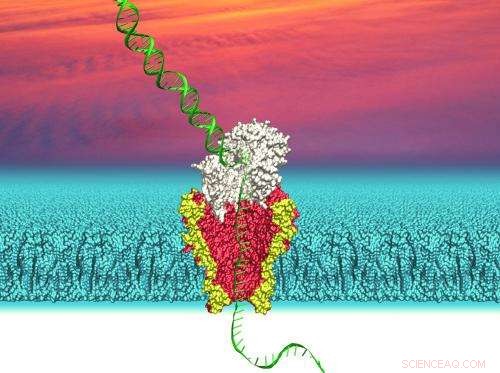
Illustration d'un nanopore dérivé d'un canal membranaire bactérien génétiquement modifié utilisé pour séquencer l'ADN. Crédit :Ian Derrington
(Phys.org) — Une technologie peu coûteuse pourrait permettre de lire de longues séquences d'ADN beaucoup plus rapidement que les techniques actuelles.
La recherche fait progresser une technologie, appelé séquençage d'ADN nanopore. S'il est perfectionné, il pourrait un jour être utilisé pour créer des appareils portables capables d'identifier rapidement des séquences d'ADN à partir d'échantillons de tissus et de l'environnement, ont déclaré les chercheurs de l'Université de Washington qui ont développé et testé l'approche.
« L'une des raisons pour lesquelles les gens sont si enthousiastes à propos du séquençage de l'ADN nanopore est que la technologie pourrait éventuellement être utilisée pour créer des dispositifs de type « tricordeur » pour détecter des agents pathogènes ou diagnostiquer des troubles génétiques rapidement et sur place, " a déclaré Andrew Laszlo, auteur principal et étudiant diplômé du laboratoire de Jen Gundlach, un professeur de physique de l'UW qui a dirigé le projet.
L'article « Decoding long nanopore sequencing reads of natural DNA » décrit la nouvelle technique. Il paraît le 25 juin dans l'édition en ligne avancée de la revue Biotechnologie naturelle .
La plupart des technologies actuelles de séquençage des gènes nécessitent de travailler avec de courts extraits d'ADN, généralement de 50 à 100 nucléotides de long. Ceux-ci doivent être traités par de gros séquenceurs en laboratoire. Le processus fastidieux peut prendre des jours à des semaines.
La technologie Nanopore tire parti du petit structures en forme de tunnel trouvées dans les membranes bactériennes. Dans la nature, ces pores permettent aux bactéries de contrôler le flux de nutriments à travers leurs membranes.
Un chercheur de l'UW a utilisé le nanopore Mycobacterium smegmatis porine A (MspA). Ce pore bactérien a été génétiquement modifié de sorte que la partie la plus étroite du canal a un diamètre d'environ un nanomètre, ou 1 milliardième de mètre. C'est assez grand pour qu'un seul brin d'ADN passe à travers. Le nanopore modifié est ensuite inséré dans une membrane séparant deux solutions salines pour créer un canal reliant les deux solutions.
Pour lire une séquence d'ADN avec ce système, une petite tension est appliquée à travers la membrane pour faire circuler les ions de la solution saline à travers le nanopore. Le flux d'ions crée un courant mesurable. Si un brin d'ADN est ajouté à la solution d'un côté de la membrane et pénètre ensuite dans un pore, les molécules d'ADN volumineuses entraveront le flux de l'ion beaucoup plus petit et modifieront ainsi le courant. Dans quelle mesure les changements actuels dépendent de quels nucléotides - les molécules individuelles d'adénine, guanine, la cytosine et la thymine qui composent la chaîne d'ADN sont à l'intérieur du pore. La détection des changements de courant peut révéler quels nucléotides traversent le canal du nanopore à un instant donné.
Depuis que la technique a été proposée pour la première fois dans les années 1990, les chercheurs espéraient que le séquençage de l'ADN nanopore offrirait un alternative rapide au séquençage génétique actuel. Mais leurs tentatives ont été contrecarrées par plusieurs défis. Il est difficile d'identifier chaque nucléotide un par un lorsqu'ils traversent le nanopore. Au lieu, les chercheurs doivent travailler avec des changements de courant associés à quatre nucléotides à la fois. En outre, certains nucléotides peuvent être manqués ou lus plusieurs fois. Par conséquent, la technologie actuelle de séquençage des nanopores donne une lecture imprécise d'une séquence d'ADN.
Les chercheurs de l'UW décrivent comment ils ont contourné ces problèmes. Les chercheurs ont d'abord identifié les signatures électroniques de toutes les combinaisons de nucléotides possibles avec les quatre nucléotides qui composent l'ADN, soit un total de 256 combinaisons en tout (4 x 4 x 4 x 4).
Ils ont ensuite créé des algorithmes informatiques pour faire correspondre les changements actuels générés lorsqu'un segment d'ADN traverse le pore avec les changements actuels attendus des séquences d'ADN de gènes et de génomes connus stockés dans une base de données informatique. Une correspondance montrerait que la séquence de l'ADN passant à travers le pore était identique ou proche de la séquence d'ADN stockée dans la base de données. L'ensemble du processus prendrait de quelques minutes à quelques heures, au lieu de semaines.
Pour tester cette approche, les chercheurs ont utilisé leur système nanopore pour lire la séquence du bactériophage Phi X 174, un virus qui infecte les bactéries et qui est couramment utilisé pour évaluer les nouvelles technologies de séquençage du génome. Ils ont découvert que l'approche lisait de manière fiable les séquences d'ADN du bactériophage et pouvait lire des séquences aussi longues que 4, 500 nucléotides.
"C'est la première fois que quelqu'un montre que les nanopores peuvent être utilisés pour générer des signatures interprétables correspondant à de très longues séquences d'ADN à partir de génomes du monde réel, " a déclaré le co-auteur Jay Shendure, un professeur agrégé UW des sciences du génome dont le laboratoire développe des applications de technologies de séquençage du génome. "C'est un grand pas en avant."
Parce que la technique repose sur l'appariement des lectures aux bases de données de gènes et de génomes précédemment séquencés, il ne peut pas encore être utilisé pour séquencer un gène ou un génome nouvellement découvert, les chercheurs ont dit, mais avec quelques raffinements, ils ont ajouté, il devrait être possible d'améliorer les performances dans ce domaine. Pour accélérer la recherche sur cette nouvelle technologie, les scientifiques font leurs méthodes, des données et des algorithmes informatiques entièrement accessibles à tous.
"Malgré les obstacles restants, notre démonstration qu'un appareil à faible coût peut lire de manière fiable les séquences d'ADN naturel et peut interpréter des segments d'ADN aussi longs que 4, 500 nucléotides de longueur représente une avancée majeure dans le séquençage de l'ADN nanopore, " a déclaré Gundlach.