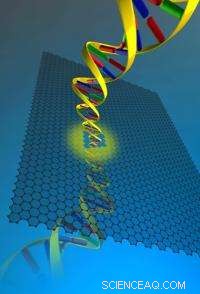
Dans un article de couverture de Nature Nanotechnology, des chercheurs de Harvard et du Massachusetts Institute of Technology ont démontré qu'une longue molécule d'ADN peut être tirée à travers un nanopore de graphène. Crédit :Slaven Garaj, Jene Golovchenko, Jing-Kong, Daniel Branton, W. Hubbard, et A. Reina
Les scientifiques font un pas de plus vers une révolution dans le séquençage de l'ADN, suite au développement dans un laboratoire de Harvard d'un minuscule appareil conçu pour lire les minuscules changements électriques produits lorsque des brins d'ADN sont passés à travers de minuscules trous - appelés nanopores - dans une membrane chargée électriquement.
Comme décrit dans Nature Nanotechnologie le 11 décembre une équipe de recherche dirigée par Charles Lieber, le professeur de chimie Mark Hyman Jr., ont réussi pour la première fois à créer un détecteur nanopore intégré, un développement qui ouvre la porte à la création de dispositifs qui pourraient utiliser des réseaux de millions de trous microscopiques pour séquencer l'ADN rapidement et à moindre coût.
Décrit pour la première fois il y a plus de 15 ans, Le séquençage des nanopores mesure les changements de courant électrique subtils produits lorsque les quatre molécules de base qui composent l'ADN traversent le pore. En lisant ces changements, les chercheurs peuvent séquencer efficacement l'ADN.
Mais la lecture de ces changements subtils de courant est loin d'être facile. Une série de défis - de la façon d'enregistrer les minuscules changements de courant à la façon d'augmenter le processus de séquençage - signifie que le processus n'a jamais été possible à grande échelle. Lieber et son équipe, cependant, croient avoir trouvé une solution unifiée à la plupart de ces problèmes.
« Jusqu'à ce que nous développions notre détecteur, il n'y avait aucun moyen de mesurer localement les changements de courant, ", a déclaré Lieber. « Notre méthode est idéale car elle est extrêmement localisée. On peut utiliser tout le travail existant qui a été fait sur les nanopores, mais avec un détecteur local, nous sommes un peu plus près de révolutionner complètement le séquençage.

"Le transistor nanofil mesure le changement de potentiel électrique au niveau du pore et amplifie efficacement le signal, », a déclaré Charles Lieber. "En plus d'un signal plus important, cela nous permet de lire les choses beaucoup plus rapidement. » Crédit :Kris Snibbe/Harvard Staff Photographer
Le détecteur développé par Lieber et son équipe est né de travaux antérieurs sur les nanofils. En utilisant les fils ultra-minces comme transistor nanométrique, ils sont capables de mesurer les changements de courant plus localement et avec plus de précision que jamais auparavant.
"Le transistor nanofil mesure le changement de potentiel électrique au niveau du pore et amplifie efficacement le signal, ", a déclaré Lieber. "En plus d'un signal plus important, cela nous permet de lire les choses beaucoup plus rapidement. C'est important parce que l'ADN est si gros [que] le débit de toute méthode de séquençage doit être élevé. En principe, ce détecteur peut fonctionner à des fréquences gigahertz.
La mesure très localisée ouvre également la porte au séquençage parallèle, qui utilise des réseaux de millions de pores pour accélérer considérablement le processus de séquençage.
En plus du potentiel d'amélioration considérable de la vitesse de séquençage, le nouveau détecteur promet de réduire considérablement le coût du séquençage de l'ADN, dit Ping Xie, un associé du Département de chimie et de biologie chimique et co-auteur de l'article décrivant la recherche.
Les méthodes de séquençage actuelles commencent souvent par un processus appelé réaction en chaîne par polymérase, ou amplification d'ADN, qui copie une petite quantité d'ADN des milliers de millions de fois, facilitant le séquençage. Bien que d'une importance cruciale pour la biologie, le processus est coûteux, nécessitant des fournitures chimiques et du matériel de laboratoire coûteux.
À l'avenir, Xie a dit, il sera possible de construire la technologie de séquençage des nanopores sur une puce de silicium, permettre aux médecins, des chercheurs, ou même la personne moyenne à utiliser le séquençage de l'ADN comme outil de diagnostic.
La percée de l'équipe de Lieber pourrait bientôt faire la transition du laboratoire au produit commercial. Le Harvard Office of Technology Development travaille sur une stratégie pour commercialiser la technologie de manière appropriée, y compris l'octroi d'une licence à une entreprise qui envisage de l'intégrer dans sa plate-forme de séquençage d'ADN.
« En ce moment, nous sommes limités dans notre capacité à effectuer le séquençage de l'ADN, " dit Xie. « La technologie de séquençage actuelle est là où se trouvaient les ordinateurs dans les années 50 et 60. Il nécessite beaucoup de matériel et coûte très cher. Mais à peine 50 ans plus tard, les ordinateurs sont partout, même dans les cartes de vœux. Notre détecteur ouvre la porte à faire une prise de sang et à savoir immédiatement de quoi un patient est infecté, et prendre très rapidement des décisions de traitement.
Cette histoire est publiée avec l'aimable autorisation de la Harvard Gazette, Journal officiel de l'Université Harvard. Pour des nouvelles universitaires supplémentaires, visitez Harvard.edu.