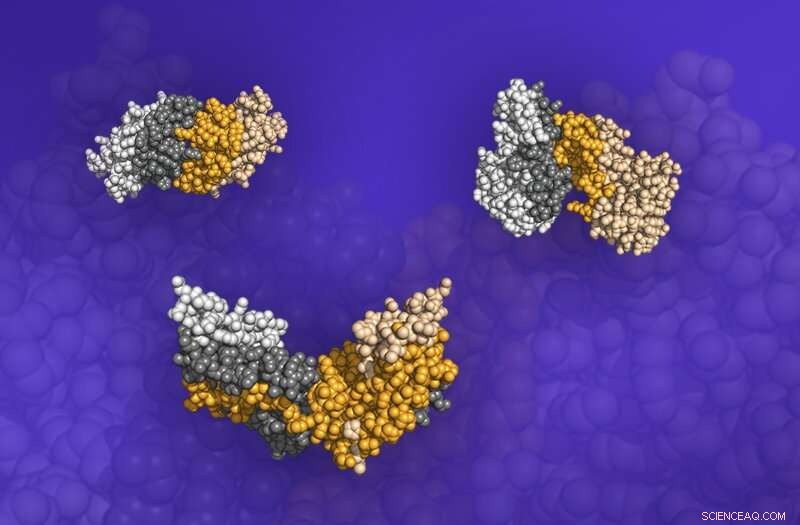
Trois dimères, structures protéiques constituées de deux protéines liées, de la base de données Dockground. Les interfaces auxquelles les protéines se rencontrent sont représentées par les régions sombres. Crédit :ORNL
Quelque chose d'aussi simple que la forme peut-il pleinement déterminer si les protéines se lieront ou non ? Les scientifiques mettent en service des superordinateurs pour le découvrir.
Une équipe dirigée par Sharon Glotzer, professeur distingué et président du département de génie chimique à l'Université du Michigan (UM), utilisé le supercalculateur Summit de 200 pétaflops du laboratoire national d'Oak Ridge (ORNL) du département américain de l'Énergie (DOE) pour modéliser les interactions de verrouillage et de clé entre les protéines afin d'étudier leurs comportements de liaison. Les résultats, Publié dans Matière douce, a révélé que certaines protéines le font, En réalité, lier basé sur la forme seule.
"Nous avons démontré que quelque chose d'aussi simple que la forme est capable de prédire des interactions protéiques parfois très complexes, " a déclaré Jens Glaser, informaticien au sein du groupe Advanced Computing for Chemistry and Materials de l’Oak Ridge Leadership Computing Facility (OLCF). "Cette première démonstration nous a amenés à croire que la forme a été un ingrédient méconnu dans de nombreux processus d'assemblage de protéines."
Les résultats pourraient avoir de nombreuses applications en recherche biologique. Par exemple, l'approche pourrait être utilisée pour cribler des médicaments contre les maladies ou fournir aux scientifiques des informations sur la façon d'utiliser les protéines comme éléments constitutifs pour concevoir de nouveaux matériaux biologiques.
"Cette étude passionnante démontre le pouvoir de la complémentarité des formes dans la prédiction des interfaces protéine-protéine, " a déclaré le Dr Stéphanie McElhinny, responsable de programme au Army Research Laboratory de l'US Army Combat Capabilities Development Command, se référant à la relation spatiale favorable entre deux protéines de forme compatible. "Les modèles informatiques qui prédisent avec précision ces interfaces soutiendront la conception future de matériaux avancés à base de protéines avec des propriétés actives et réactives, tels que les plastiques à base de protéines de récolte de lumière qui pourraient fonctionner comme une feuille artificielle pour la production d'électricité. »
Les superordinateurs révèlent que la forme est la clé de certaines protéines
Pour que les protéines se lient avec succès les unes aux autres, l'un d'eux agit comme un ligand, une molécule qui se fixe à une protéine cible, et l'un d'eux agit comme un récepteur, la molécule qui reçoit le ligand. Ce processus implique des interactions chimiques complexes, dans lequel les molécules partagent des liaisons et changent de configuration lors de la liaison.
L'équipe de Glotzer voulait voir s'ils pouvaient prédire cette liaison moléculaire basée uniquement sur la forme, ignorant les interactions entre les protéines. A partir d'une base de données de plus de 6, 000 paires de protéines, l'équipe a testé 46 paires connues pour se lier les unes aux autres et a simulé leur assemblage sur Summit. L'équipe a effectué les simulations dans le cadre du programme INCITE (Innovative and Novel Computational Impact on Theory and Experiment).
Comme plusieurs balles de tennis lancées sur une seule cible, les simulations ont modélisé plusieurs ligands lancés en un seul, récepteur cible fixe. Sur les 46 paires testées, ils ont trouvé 6 paires qui ont bien fonctionné - plus de 50 pour cent du temps, ils ont réussi à les assembler en se basant uniquement sur leurs formes complémentaires.
"Nous avons examiné les interfaces où les protéines se sont liées pour voir à quel point elles étaient similaires à leurs interfaces réelles, puis nous avons déterminé le seuil pour voir combien de paires étaient de bons prédicteurs des interfaces réelles, " dit Fengyi Gao, doctorat candidat à l'UM. "Nous avons découvert que 13% de ces paires de protéines pouvaient se lier en fonction de la forme seule."
L'équipe a ensuite construit un modèle d'apprentissage automatique qui pourrait déterminer quelles protéines sont capables de s'assembler uniquement en fonction de leur forme. La combinaison de leur modèle initial avec de tels outils d'apprentissage automatique les aidera à comprendre quelles informations sont nécessaires pour les paires de protéines qui ne peuvent pas s'assembler sur la seule base de la complémentarité des formes.