Les régions délimitées par les cases jaunes en pointillés à gauche sont agrandies à droite. Barres d'échelle, 5 m (gauche) et 1 m (droite). Crédit :par Dashan Dong, Xiaoshuai Huang, Liuju Li, Heng Mao, Yanquan Mo, Guangyi Zhang, Zhe Zhang, Jiayu Shen, Wei Liu, Zeming Wu, Guanghui Liu, Yanmei Liu, Hong Yang, Qihuang Gong, Kebin Shi, Liangyi Chen
L'émergence de la microscopie à fluorescence à superrésolution (SR) a rajeuni la recherche de nouvelles sous-structures cellulaires et d'intermédiaires dynamiques. Cependant, limité par le large spectre d'émission des fluorophores et une phototoxicité excessive, La microscopie à fluorescence SR ne peut être utilisée que pour mettre en évidence une poignée de biomolécules simultanément et est incapable de fournir une carte holistique de l'environnement et du paysage cellulaires.
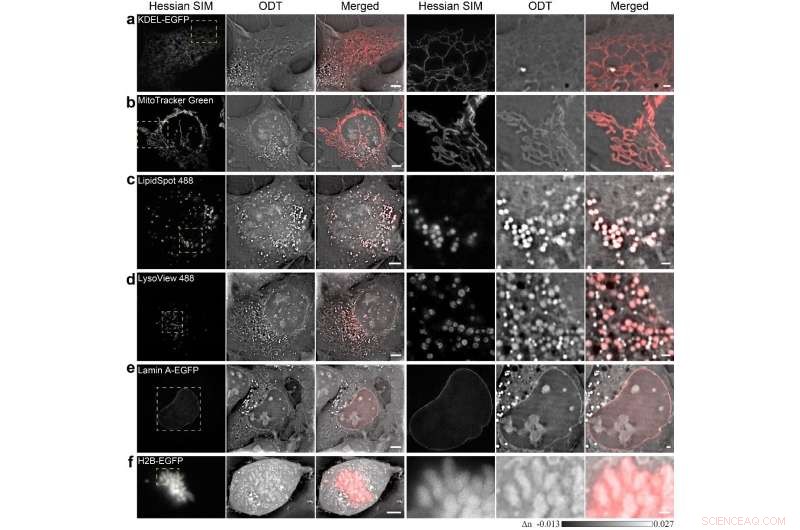
Dans un nouvel article publié dans Science de la lumière et applications , des scientifiques du State Key Laboratory for Mesoscopic Physics and Frontiers Science Center for Nano-optoelectronics, Université de Pékin, Chine, Laboratoire clé d'État de biologie membranaire, Laboratoire clé de Pékin de médecine moléculaire cardiométabolique, Institut de médecine moléculaire, Université de Pékin, La Chine et ses collaborateurs ont développé la tomographie informatique par diffraction assistée par fluorescence SR (SR-FACT), qui combine la tomographie par diffraction optique (ODT) tridimensionnelle sans marquage avec la microscopie à illumination structurée de Hesse à fluorescence bidimensionnelle. Le module ODT est capable de résoudre les mitochondries, gouttelettes lipidiques, la membrane nucléaire, chromosomiques, le réticulum endoplasmique tubulaire et les lysosomes. En utilisant l'imagerie de cellules vivantes corrélées bimode pendant une période prolongée, ils ont observé de nouvelles structures subcellulaires appelées corps à vacuole noire (DB), dont la majorité provient de régions périnucléaires densément peuplées, et interagissent intensément avec les organites tels que les mitochondries et la membrane nucléaire avant de s'effondrer finalement dans la membrane plasmique. Ce travail démontre les capacités uniques de SR-FACT, ce qui suggère sa large applicabilité en biologie cellulaire en général.
SR-FACT visualise à la fois le paysage cellulaire et l'identité moléculaire des cellules vivantes. Un nouvel algorithme appelé algorithme de recherche itérative vectorielle (VISA) a été développé pour minimiser les erreurs de reconstruction d'imagerie 3D sous un schéma de balayage tomographique à haute vitesse de kHz. Par conséquent, SR-FACT peut utiliser simultanément une vitesse d'imagerie maximale pour capturer la dynamique dans les cellules vivantes et maintenir un flux de photons suffisant pour une sensibilité maximale. Dans le système SR-FACT, le module ODT a atteint une résolution latérale d'environ 200 nm à une vitesse d'imagerie volumétrique de 0,8 Hz (40 × 40 × 20 m 3 ). Hesse 2-D-SIM, qui permet une imagerie SR à une fraction de la dose de photons utilisée par le SIM classique, a été utilisé pour guider l'interprétation des structures observées par le module ODT. En réalisant une imagerie corrélée bimode dans des cellules COS-7, ils ont résolu six organites connus sans marquage :le réticulum endoplasmique tubulaire (RE), mitochondries, endosomes/lysosomes tardifs (LEs/LYs), LD, la membrane nucléaire et les chromosomes. Toutes ces données mettent en évidence l'avantage unique de SR-FACT dans l'étude de l'interactome des organites. De plus, ils ont également observé des structures vacuolées à pH neutre qui contenaient principalement du liquide dans la lumière. L'imagerie SR de cellules vivantes d'une heure, combinée à une analyse quantitative, révèle les voies de trafic non conventionnelles et les rôles indispensables des vacuoles dans l'organisation de l'interactome des organites, ce qui suggère qu'ils représentent des organites auparavant méconnus.