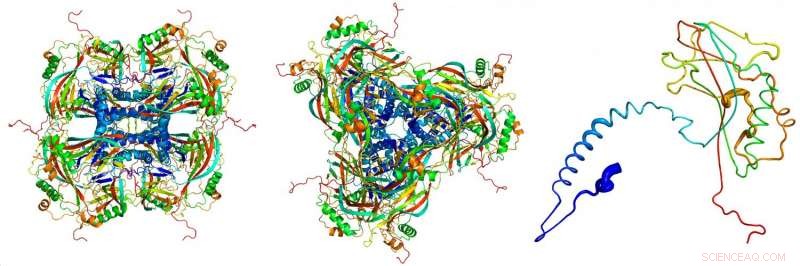
Modèle atomique des corps d'occlusion cristallins, dérivées des images de diffraction des rayons X enregistrées au laser à rayons X à électrons libres LCLS du SLAC National Accelerator Laboratory. Les protéines individuelles (à droite) se collent pour former les blocs de construction (à gauche, vu de côté; centre, vu de dessus) des corps cristallins d'occlusion. Crédit :Dominik Oberthuer, CFEL/DESY
Une équipe internationale de scientifiques a utilisé des impulsions de rayons X à haute intensité pour déterminer la structure de l'enveloppe protéique cristalline d'un virus d'insecte. Leur analyse révèle les détails fins des éléments constitutifs du cocon viral jusqu'à une échelle de 0,2 nanomètre (millionième de millimètre) - approchant la résolution à l'échelle de l'atome. Les minuscules virus avec leur enveloppe cristalline sont de loin les plus petits cristaux de protéines jamais analysés par cristallographie aux rayons X. Cela ouvre de nouvelles perspectives dans l'étude des structures des protéines, comme le rapporte l'équipe dirigée par le scientifique principal de DESY, Henry Chapman du Center for Free-Electron Laser Science, dans le Actes de l'Académie nationale des sciences ( PNAS ).
"Le granulovirus attaque certains insectes et les tue. Cela le laisse d'abord bloqué à l'intérieur de l'hôte en décomposition, il doit donc se protéger, peut-être pendant des années, contre les conditions environnementales défavorables telles que la chaleur, rayonnement ultraviolet et sécheresse, jusqu'à ce qu'il soit à nouveau ingéré par un insecte. Pour y parvenir, le virus s'enveloppe dans un cocon fait de cristaux de protéines, qui ne se dissout à nouveau qu'une fois qu'il atteint l'intestin d'un insecte, " explique Cornelius Gati de DESY, l'auteur principal de l'article. Ces virus intéressent particulièrement Peter Metcalf de l'Université d'Auckland en Nouvelle-Zélande et Johannes Jehle du Julius Kühn Institute de Darmstadt, qui s'est associé à DESY pour cette recherche. Les chercheurs ont examiné le cocon de la Cydia pomonella granulovirus (CpGV), qui infecte les chenilles du carpocapse ( Cydia pomonella ) et est utilisé en agriculture comme pesticide biologique. Le virus est inoffensif pour l'homme.
Les scientifiques s'intéressent à la structure spatiale des protéines et autres biomolécules car cela permet d'éclairer la manière précise dont elles fonctionnent. Cela a conduit à une science spécialisée connue sous le nom de biologie structurale. « Au cours des 50 dernières années, les scientifiques ont déterminé les structures de plus de 100, 000 protéines, " dit Chapman, qui est également professeur de physique à l'Université de Hambourg. "L'outil de loin le plus important pour cela est la cristallographie aux rayons X." Dans cette méthode, un cristal de la protéine étudiée est cultivé et irradié avec des rayons X brillants. Cela produit un motif de diffraction caractéristique, à partir de laquelle la structure spatiale du cristal et de ses blocs de construction peut être calculée.
« L'un des grands défis de cette procédure est, cependant, faire pousser les cristaux, " ajoute Chapman. De nombreuses protéines ne s'alignent pas facilement pour former des cristaux, car ce n'est pas leur état naturel. Plus les cristaux pouvant être utilisés pour l'analyse sont petits, plus il est facile de les cultiver, mais plus il est difficile de les mesurer. "Nous espérons qu'à l'avenir, nous pourrons nous passer complètement de la croissance des cristaux et étudier des molécules individuelles directement à l'aide de rayons X, " dit Chapman, "donc nous aimerions comprendre les limites".
"Ces particules virales nous ont fourni les plus petits cristaux de protéines jamais utilisés pour l'analyse de la structure aux rayons X, " explique Gati. Le corps d'occlusion (le virus "cocon") a un volume d'environ 0,01 micromètre cube, environ cent fois plus petits que les plus petits cristaux de protéines cultivés artificiellement qui ont jusqu'à présent été analysés à l'aide de techniques cristallographiques.
Pour briser cette limite de taille de cristal, un faisceau de rayons X extrêmement brillant était nécessaire, qui a été obtenu à l'aide d'un laser dit à électrons libres (FEL), dans lequel un faisceau d'électrons à grande vitesse est guidé à travers un onduleur magnétique les obligeant à émettre des impulsions de rayons X de type laser.
Les scientifiques ont utilisé le laser à électrons libres LCLS au SLAC National Accelerator Laboratory aux États-Unis, et a utilisé des optiques pour focaliser chaque impulsion de rayons X à une taille similaire à celle de l'une des particules virales. " Diriger toute la puissance du FEL sur un minuscule virus l'a exposé à des niveaux de rayonnement énormes, " rapporte Gati, qui travaille maintenant au SLAC. La dose était de 1,3 milliard de Grays; à titre de comparaison :la dose létale pour l'homme est d'environ 50 Grays.
La dose FEL était certainement mortelle pour les virus aussi - chacun a été complètement vaporisé par une seule impulsion de rayons X. Mais l'impulsion de durée femtoseconde transporte l'information de la structure vierge jusqu'au détecteur et la destruction du virus ne se produit qu'après le passage de l'impulsion. L'analyse de la diffraction enregistrée a montré que même de minuscules cristaux de protéines qui sont bombardés de doses de rayonnement extrêmement élevées peuvent encore révéler leur structure à l'échelle atomique.
"Les simulations basées sur nos mesures suggèrent que notre méthode peut probablement être utilisée pour déterminer la structure de cristaux encore plus petits constitués de seulement des centaines ou des milliers de molécules, " rapporte Chapman, qui est également membre du Hamburg Center for Ultrafast Imaging (CUI). "Cela nous fait faire un pas de plus vers notre objectif d'analyser des molécules individuelles."