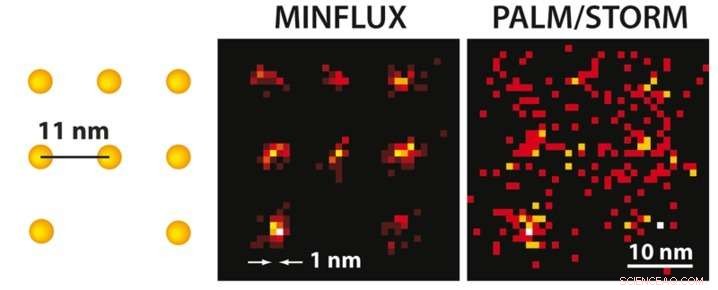
Avec la microscopie MINFLUX on peut, pour la première fois, séparer optiquement des molécules qui ne sont distantes que de quelques nanomètres. À gauche, un schéma des molécules fluorescentes est présenté. Alors que la microscopie PALM/STORM ultra haute résolution à la même luminosité moléculaire (à droite) délivre une image diffuse des molécules (ici en simulation dans des conditions techniques idéales), la position des molécules individuelles peut être facilement discernée avec le MINFLUX réalisé dans la pratique (au milieu). Crédit :MPI f. Chimie biophysique/ K. Gwosch
C'est le Saint Graal de la microscopie optique :améliorer le pouvoir de résolution de cette méthode de telle sorte que l'on puisse discerner individuellement des molécules très proches les unes des autres. Les scientifiques autour du lauréat du prix Nobel Stefan Hell de l'Institut Max Planck de chimie biophysique de Göttingen ont maintenant réalisé ce qui était longtemps considéré comme impossible - ils ont développé un nouveau microscope à fluorescence, appelé MINFLUX, permettant, pour la première fois, séparer optiquement des molécules, qui ne sont distants que de quelques nanomètres (un millionième de millimètre). Ce microscope est plus de 100 fois plus précis que la microscopie optique conventionnelle et surpasse même les meilleures méthodes de microscopie optique à super-résolution à ce jour, à savoir STED développé par Hell et PALM/STORM décrit par le lauréat du prix Nobel Eric Betzig, jusqu'à 20 fois. Pour MINFLUX, Hell a utilisé les avantages de STED et PALM/STORM dans un tout nouveau concept. Cette percée ouvre de nouvelles opportunités pour les chercheurs d'étudier le fonctionnement de la vie au niveau moléculaire.
"Nous avons régulièrement atteint des résolutions du nanomètre avec MINFLUX, qui est le diamètre des molécules individuelles - la limite ultime de ce qui est possible en microscopie à fluorescence, " explique l'Enfer, Directeur à l'Institut Max Planck de chimie biophysique. "Je suis convaincu que les microscopes MINFLUX ont le potentiel pour devenir l'un des outils les plus fondamentaux de la biologie cellulaire. Avec ce concept, il sera possible de cartographier les cellules en détail moléculaire et d'observer les processus rapides à l'intérieur en temps réel. Cela pourrait révolutionner notre connaissance des processus moléculaires se produisant dans les cellules vivantes."
Le physicien de Göttingen, qui travaille également à l'Institut Max Planck de recherche médicale et au Centre allemand de recherche sur le cancer à Heidelberg, est convaincu depuis longtemps que la résolution de la microscopie à fluorescence peut être augmentée jusqu'à la dimension des molécules individuelles - avec l'utilisation classique de la lumière focalisée et des lentilles conventionnelles.
En réalité, le physicien Ernst Abbe avait formulé en 1873 que la résolution des microscopes optiques est limitée à la moitié de la longueur d'onde de la lumière, qui est d'environ 200 nanomètres. Plus de 100 ans plus tard, cette limite d'Abbe est toujours valable. Cependant, Hell a été le premier à montrer que cette limite peut être dépassée avec la microscopie STED, qu'il a conçu en 1994 et établi à titre expérimental cinq ans plus tard.
STED ainsi que PALM/STORM, développé quelques années plus tard, en pratique, atteindre une netteté de séparation d'environ 20 à 30 nanomètres - environ dix fois mieux que la limite d'Abbe. Pour le développement de ces techniques de microscopie optique à ultra haute résolution, Hell et Betzig ainsi que William E. Moerner ont reçu le prix Nobel de chimie 2014.
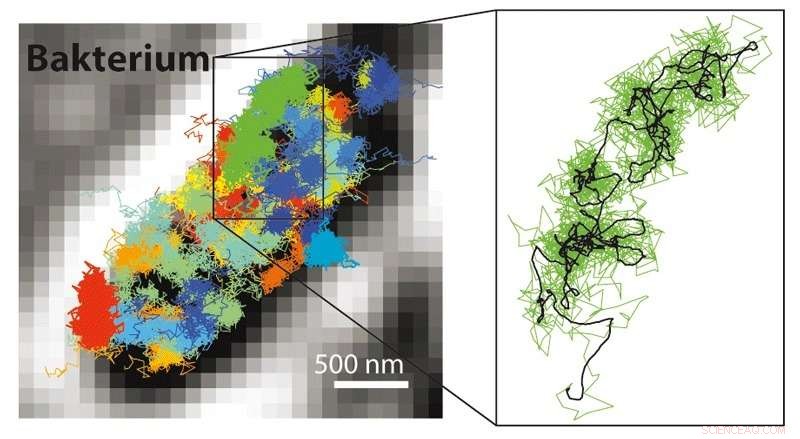
Avec MINFLUX, il est possible de suivre de nombreux mouvements beaucoup plus rapides qu'avec la microscopie STED ou PALM/STORM. Il est donc possible de rendre visibles les mouvements de molécules marquées par fluorescence dans une cellule vivante. À gauche :modèle de mouvement des ribosomes 30S (parties d'usines de protéines, coloré) dans une bactérie E. coli (noir-blanc). À droite :modèle de mouvement d'un seul ribosome 30S (vert) illustré agrandi. Crédit :MPI f. Chimie biophysique/ Y. Eilers
Avantages de STED et PALM/STORM combinés
STED et PALM/STORM séparent les molécules fluorescentes voisines en les allumant et en les éteignant l'une après l'autre afin qu'elles émettent une fluorescence séquentiellement. Cependant, les méthodes diffèrent sur un point essentiel :la microscopie STED utilise un faisceau laser en forme de beignet pour désactiver la fluorescence moléculaire à un endroit fixe dans l'échantillon, c'est-à-dire partout dans la région focale sauf au centre du beignet. L'avantage est que le faisceau de donut définit exactement à quel point de l'espace se trouve la molécule lumineuse correspondante. L'inconvénient est qu'en pratique le faisceau laser n'est pas assez puissant pour confiner l'émission à une seule molécule au centre de l'anneau. Dans le cas de PALM/STORM, d'autre part, l'allumage et l'extinction se font à des emplacements aléatoires et au niveau d'une molécule unique. L'avantage ici est que l'on travaille déjà au niveau d'une molécule unique, mais un inconvénient est que l'on ne connaît pas les positions exactes des molécules dans l'espace. Les positions doivent être découvertes en collectant autant de photons de fluorescence que possible sur une caméra; plus que 50, 000 photons détectés sont nécessaires pour atteindre une résolution inférieure à 10 nanomètres. En pratique, on ne peut donc pas atteindre en routine une résolution moléculaire (un nanomètre).
Hell a eu l'idée de combiner de manière unique les forces des deux méthodes dans un nouveau concept. "Cette tâche était tout sauf triviale. Mais mes collègues Francisco Balzarotti, Yvan Eilers, et Klaus Gwosch ont fait un travail formidable en mettant en œuvre cette idée expérimentalement avec moi. » Leur nouvelle technique, appelé MINFLUX (MINimal Emission FLUXes), est maintenant présenté par Hell avec les trois scientifiques juniors en tant que premiers auteurs de Science.
MINFLUX, comme PALM/STORM, active et désactive aléatoirement des molécules individuelles. Cependant, à la fois, leurs positions exactes sont déterminées avec un faisceau laser en forme de beignet comme dans STED. Contrairement à STED, le faisceau de beignet excite ici la fluorescence. Si la molécule est sur l'anneau, il brillera; si c'est exactement au centre sombre, il ne brillera pas mais on a trouvé sa position exacte. Balzarotti a développé un algorithme intelligent pour que cette position puisse être localisée très rapidement et avec une grande précision. "Avec cet algorithme, il a été possible d'exploiter le potentiel du faisceau d'excitation du donut, " explique le jeune scientifique. Gwosch, qui a obtenu les images à résolution moléculaire, ajoute "C'était un sentiment incroyable alors que nous, pour la première fois, ont pu distinguer des détails avec MINFLUX à l'échelle de quelques nanomètres."
100 fois meilleure résolution
En plus de la résolution moléculaire, la combinaison de STED et PALM/STORM offre un avantage supplémentaire majeur :« MINFLUX est beaucoup plus rapide en comparaison. Comme il fonctionne avec un faisceau laser donut, il nécessite un signal lumineux beaucoup plus faible, c'est-à-dire moins de photons de fluorescence, par molécule par rapport à PALM/STORM pour atteindre la résolution ultime, " Hell States. Déjà avec STED on pouvait enregistrer des vidéos en temps réel de l'intérieur de cellules vivantes. Mais maintenant il était possible de suivre le mouvement des molécules dans une cellule avec une résolution temporelle 100 fois meilleure, comme le souligne Eilers. Il a réussi à filmer pour la première fois le mouvement des molécules dans une bactérie E. coli vivante avec MINFLUX, avec une résolution spatio-temporelle sans précédent. "En ce qui concerne la vitesse, nous n'avons pas exploité au maximum les possibilités de MINFLUX, " dit Eilers. Les chercheurs sont convaincus que même des changements extrêmement rapides dans les cellules vivantes peuvent être étudiés à l'avenir, comme par exemple le mouvement des nanomachines cellulaires ou le repliement des protéines.