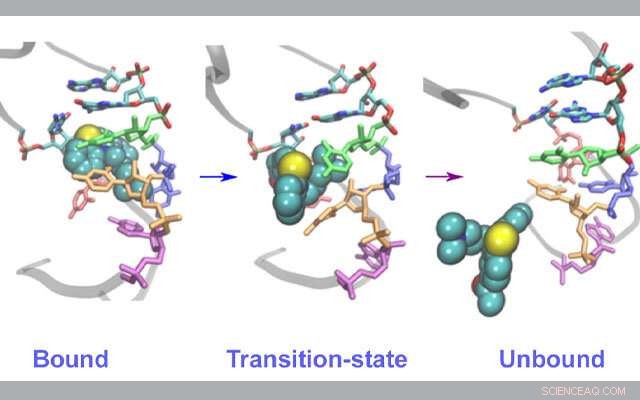
Modifications structurelles de l'ARN au cours de la liaison/déconnexion du médicament. Crédit :Lev Levintov, Université du New Hampshire
Des chercheurs de l'Université du New Hampshire (UNH) ont récemment utilisé Comet au San Diego Supercomputer Center de l'UC San Diego et Stampede2 au Texas Advanced Computing Center pour identifier de nouvelles voies de liaison/déliaison d'inhibiteurs dans un virus à ARN. Les résultats pourraient être bénéfiques pour comprendre comment ces inhibiteurs réagissent et potentiellement aider à développer une nouvelle génération de médicaments pour cibler les virus avec des taux de mortalité élevés, comme le VIH-1, Zika, Ebola, et SARS-CoV2, le virus qui cause le COVID-19.
« Quand nous avons commencé cette recherche, nous n'avions jamais prévu que nous serions au milieu d'une pandémie causée par un virus à ARN, " dit Harish Vashisth, professeur agrégé de génie chimique à l'UNH. « À mesure que ces types de virus émergent, nos découvertes offriront, espérons-le, une meilleure compréhension de la façon dont les ARN viraux interagissent avec les inhibiteurs et seront utilisés pour concevoir de meilleurs traitements. »
Semblable à la façon dont les humains codent leur génome en utilisant l'ADN, de nombreux virus ont une constitution génétique de molécules d'ARN. Ces génomes à base d'ARN contiennent des sites potentiels où les inhibiteurs peuvent se fixer et désactiver le virus. Une partie du défi dans le développement de médicaments est que des variations ou des mutations dans le génome viral peuvent empêcher les inhibiteurs de se fixer.
Dans leur étude, récemment publié dans le Journal des lettres de chimie physique , Vashisth et son équipe ont créé des simulations de dynamique moléculaire à l'aide des superordinateurs Comet et Stampede2 pour examiner spécifiquement un fragment d'ARN du virus VIH-1 et son interaction avec l'acétylpromazine, une petite molécule connue pour interférer avec le processus de réplication du virus.
Les scientifiques se sont concentrés sur les éléments structurels du génome à ARN du VIH-1, car ils sont considérés comme un bon modèle pour étudier les mêmes processus sur un large éventail de virus à ARN. Ces simulations leur ont permis de découvrir les voies de la déliaison de l'inhibiteur de l'ARN viral dans plusieurs événements rares - qui sont souvent difficiles à observer expérimentalement - qui ont montré de manière inattendue un mouvement coordonné dans de nombreuses parties de la poche de liaison qui sont les éléments constitutifs de l'ARN.
Grâce aux allocations de la National Science Foundation (NSF) Extreme Science and Engineering Environment (XSEDE) sur Comet et Stampede2, les chercheurs ont pu exécuter des centaines de simulations en même temps pour observer ce que l'on appelle des événements de retournement de base rares impliqués dans le processus de liaison/déliaison des inhibiteurs qui ont fourni les nouveaux détails du mécanisme sous-jacent de ce processus.
« Notre espoir est que cela ajoute de nouvelles possibilités à un domaine traditionnellement axé sur les structures biomoléculaires statiques et mène à de nouveaux médicaments, " a déclaré Vashisth.