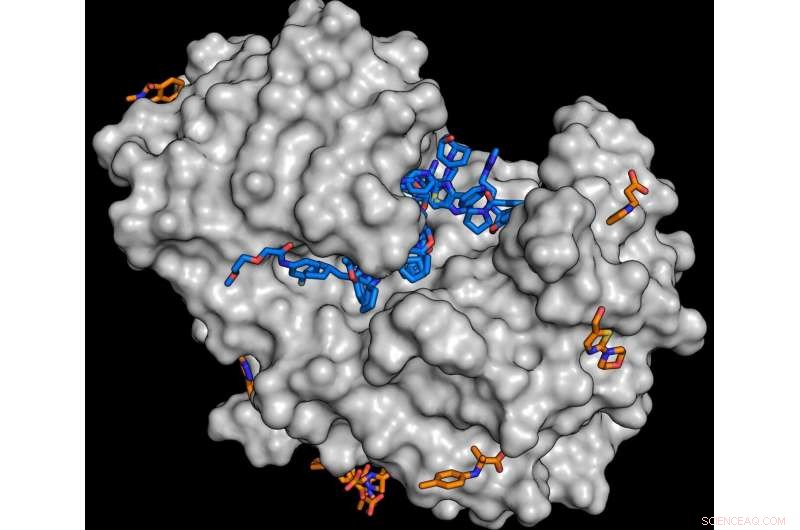
Pour l'étude, l'enzyme endothiapepsine (gris) a été combinée avec des molécules de la banque de fragments. L'analyse montre que de nombreuses substances sont capables de s'arrimer à l'enzyme (molécules bleues et oranges). Chaque substance trouvée est un point de départ potentiel pour le développement de molécules plus grosses. Crédit :J. Wollenhaupt/HZB
Afin d'accélérer le développement systématique de médicaments, l'équipe MX du Helmholtz-Zentrum Berlin (HZB) et le Drug Design Group de l'Université de Marburg ont créé une nouvelle bibliothèque de substances. Il se compose de 1103 molécules organiques qui pourraient être utilisées comme éléments constitutifs de nouveaux médicaments. L'équipe MX a maintenant validé cette bibliothèque en collaboration avec le groupe FragMAX au MAX IV. La bibliothèque de substances du HZB est disponible pour la recherche dans le monde entier et joue également un rôle dans la recherche de substances actives contre le SRAS-CoV-19.
Pour que les médicaments soient efficaces, ils doivent généralement s'arrimer aux protéines d'un organisme. Comme une clé dans une serrure, une partie de la molécule de médicament doit s'insérer dans des évidements ou des cavités de la protéine cible. Depuis plusieurs années maintenant, l'équipe du département de cristallographie macromoléculaire (MX) du HZB dirigée par le Dr Manfred Weiss et le Drug Design Group dirigé par le professeur Gerhard Klebe (Université de Marburg) ont donc travaillé à la constitution de ce que l'on appelle des bibliothèques de fragments. Ceux-ci sont constitués de petites molécules organiques (fragments) avec lesquelles les cavités fonctionnellement importantes à la surface des protéines peuvent être sondées et cartographiées. Les cristaux de protéines sont saturés avec les fragments puis analysés à l'aide d'une puissante lumière à rayons X. Cela permet d'obtenir des informations structurelles tridimensionnelles à des niveaux de résolution atomique. Entre autres, il est possible de découvrir dans quelle mesure un fragment de molécule spécifique s'arrime à la protéine cible. Le développement de ces bibliothèques de substances a eu lieu dans le cadre du projet de recherche conjoint Frag4Lead et a été financé par le ministère fédéral allemand de l'Éducation et de la Recherche (BMBF).
L'équipe MX (MX signifie Macromolecular Crystallography) a maintenant publié la conception d'une bibliothèque de fragments chimiquement diversifiée appelée bibliothèque "F2X-Universal", qui se compose de 1, 103 composés. Une sélection représentative de 96 composés a été extraite de cette bibliothèque, qui est appelé l'écran de saisie F2X. Dans le cadre de l'édition de la bibliothèque, cette sélection a maintenant été testée et validée avec succès par l'équipe MX du HZB à la source de rayons X MAX IV à Lund, Suède et à BESSY II.
Dans l'étude, les équipes HZB et MAX IV ont vérifié l'efficacité de la bibliothèque d'entrée F2X en criblant l'endothiapepsine et le complexe protéique Aar2/RnaseH comme enzymes cibles. A l'étape suivante, l'équipe MX utilisera l'intégralité de la bibliothèque universelle.
"Pour l'étude en cours, les experts en criblage de fragments de HZB—BESSY II ont travaillé en étroite collaboration avec l'équipe du projet FragMAX de MAX IV", a déclaré le Dr Uwe Müller de l'équipe MX de HZB qui a aidé à mettre en place les trois lignes de lumière MX à BESSY II ainsi que la ligne de lumière BioMAX à MAX IV. « Cela a permis aux deux partenaires de développer davantage leurs propres plates-formes technologiques et de les utiliser pour l'imagerie des surfaces fonctionnelles de différentes protéines. Ce sera une excellente base pour une future collaboration entre MAX IV et HZB.